✨데이터 자체에 집중하고 데이터에서 답을 얻으려고 노력해야지
모델에서 마법처럼 성능 향상이 일어나는 경우는 드물다.
1. Logistic Regression
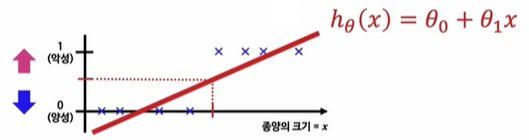
Linear Regression을 그대로 적용하면 분류 문제를 해결하기 어렵다.
예측값이 0보다 작거나 1보다 큰 값을 가질 수 있기 때문이다.
예측값이 항상 0에서 1 사이의 값을 갖도록 Hypothesis 함수를 수정하자!
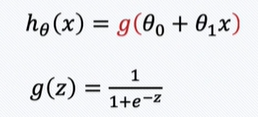
import numpy as np
import matplotlib.pyplot as plt
%matplotlib inline
z = np.arange(-10, 10, 0.01)
g = 1 / (1+np.exp(-z))
plt.figure(figsize=(12, 8))
# gca() : 설정값을 변경할 수 있는 함수
ax = plt.gca()
ax.plot(z, g)
# spines : 축 설정
ax.spines['left'].set_position('zero')
ax.spines['bottom'].set_position('center')
ax.spines['right'].set_color('none')
ax.spines['top'].set_color('none')
plt.show()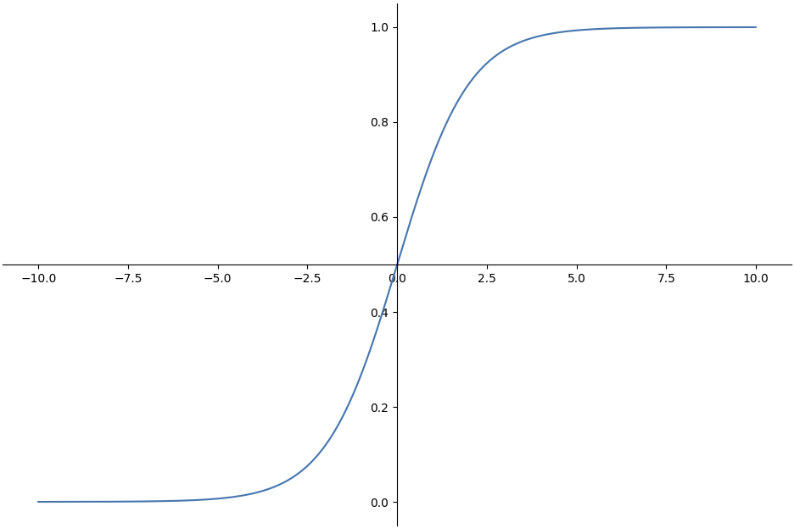
시그모이드 함수에 직선의 함수를 넣어서 결과를 판정한다.
예측값 = 주어진 x(종양의 크기)에서의 예측 결과가 1(악성)이 될 확률
Decision Boundary
Linear Regression에서 Cost Function은 단순하지만, Logistic Regression의 미분식은 복잡하기 때문에 Cost Function을 재정의할 필요가 있다.
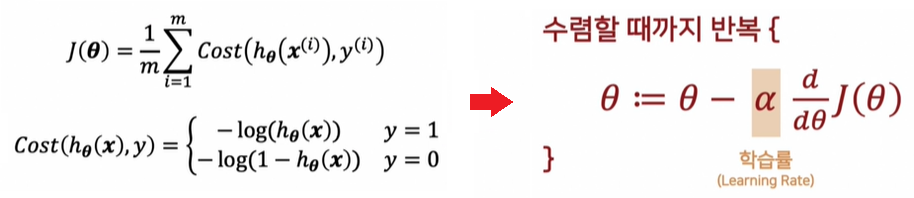
h = np.arange(0.01, 1, 0.01)
C0 = -np.log(1-h)
C1 = -np.log(h)
plt.figure(figsize=(12, 8))
plt.plot(h, C0, label='y=0')
plt.plot(h, C1, label='y=1')
plt.legend()
plt.show()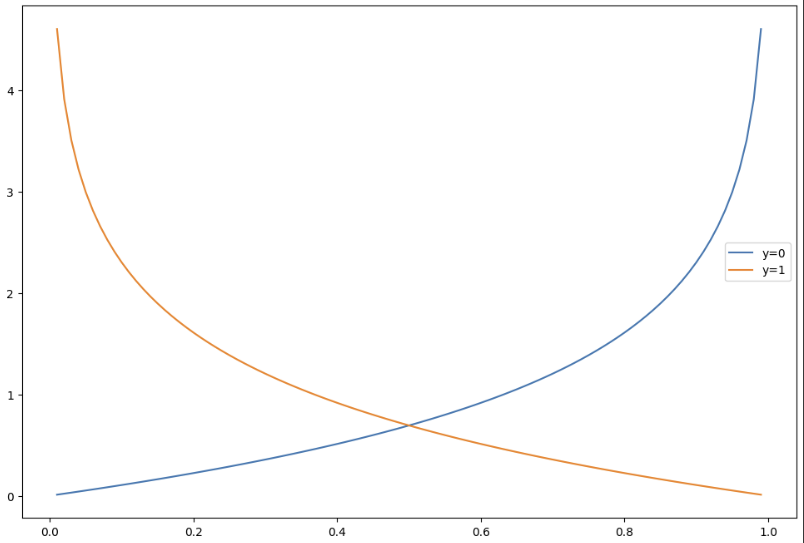
2. 실습 : wine data
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
# solver : 최적합 알고리즘 선택
# 데이터 수가 작으면 주로 liblinear
lr = LogisticRegression(solver='liblinear', random_state=13)
lr.fit(X_train, y_train)
y_pred_tr = lr.predict(X_train)
y_pred_test = lr.predict(X_test)
accuracy_score(y_train, y_pred_tr), accuracy_score(y_test, y_pred_test)
>>>
(0.7427361939580527, 0.7438461538461538)StandardScaler 적용해서 비교해보면 (+Pipeline)
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
estimators = [('scaler', StandardScaler()),
('clf', LogisticRegression(solver='liblinear', random_state=13))]
pipe = Pipeline(estimators)
pipe.fit(X_train, y_train)
y_pred_tr = pipe.predict(X_train)
y_pred_test = pipe.predict(X_test)
accuracy_score(y_train, y_pred_tr), accuracy_score(y_test, y_pred_test)
>>>
(0.7444679622859341, 0.7469230769230769)모델 간 비교를 위해 DecisionTreeClassifier 적용
from sklearn.tree import DecisionTreeClassifier
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(X_train, y_train)# AUC 그래프를 통한 모델 간 비교
from sklearn.metrics import roc_curve
models = {
'logistic regression': pipe,
'decision tree': wine_tree
}
plt.figure(figsize=(10, 8))
plt.plot([0, 1], [0, 1]) # 기준선
for model_name, model in models.items():
pred = model.predict_proba(X_test)[:, 1]
fpr, tpr, thresholds = roc_curve(y_test, pred)
plt.plot(fpr, tpr, label=model_name)
plt.grid()
plt.legend()
plt.show()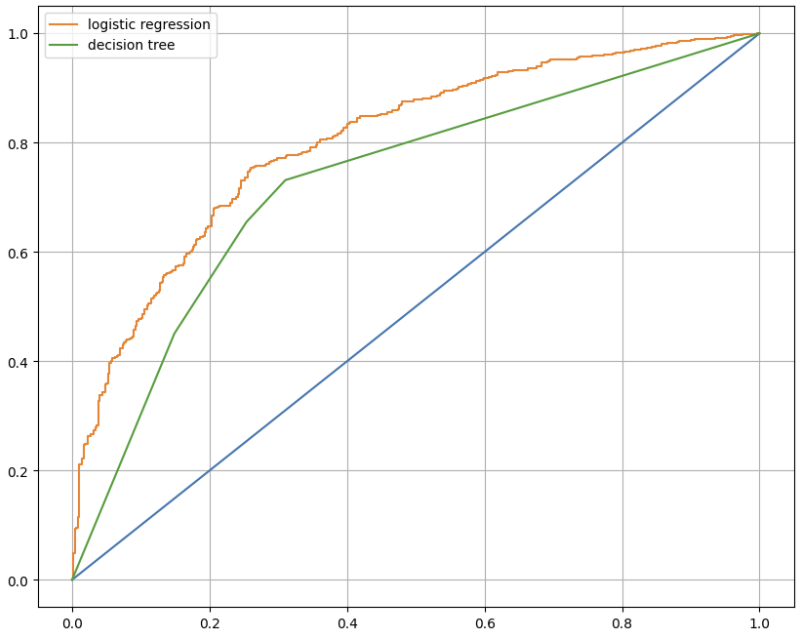
3. 실습 : PIMA 인디언 당뇨병 예측
# 모든 컬럼이 int, float ➡ 혹시 모르니 모두 float로 변경
pima = pima.astype('float')# 상관관계 확인
import seaborn as sns
plt.figure(figsize=(12, 10))
sns.heatmap(pima.corr(), cmap='YlGnBu')
plt.show()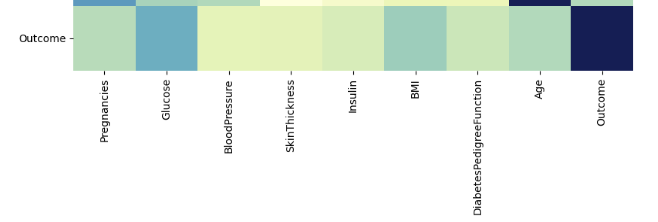
# 결측치 확인
(pima == 0).astype(int).sum()
>>>
Pregnancies 111
Glucose 5
BloodPressure 35
SkinThickness 227
Insulin 374
BMI 11
DiabetesPedigreeFunction 0
Age 0
Outcome 500결측치는 데이터에 따라 그 정의가 다르다. 예를 들어 혈압이 0일 수는 없다.
PIMA 인디언에 대한 정보와 의학적 지식이 부족하므로 결측치를 평균값으로 대체한다.
zero_features = ['Glucose', 'BloodPressure', 'SkinThickness', 'BMI']
pima[zero_features] = pima[zero_features].replace(0, pima[zero_features].mean())X = pima.drop(['Outcome'], axis=1)
y = pima['Outcome']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2,
random_state=13, stratify=y)
estimators = [('scaler', StandardScaler()),
('clf', LogisticRegression(solver='liblinear', random_state=13))]
pipe_lr = Pipeline(estimators)
pipe_lr.fit(X_train, y_train)
pred = pipe_lr.predict(X_test)from sklearn.metrics import (accuracy_score, recall_score, precision_score,
roc_auc_score, f1_score)
print(accuracy_score(y_test, pred))
print(recall_score(y_test, pred))
print(precision_score(y_test, pred))
print(roc_auc_score(y_test, pred))
print(f1_score(y_test, pred))
>>>
0.7727272727272727
0.6111111111111112
0.7021276595744681
0.7355555555555556
0.6534653465346535다변수 방정식의 계수 값 확인하기
coeff = list(pipe_lr['clf'].coef_[0])
labels = list(X_train.columns)features = pd.DataFrame({'Features': labels, 'importance': coeff})
features.sort_values(by=['importance'], ascending=True, inplace=True)
features['positive'] = features['importance'] > 0
features.set_index('Features', inplace=True)
features['importance'].plot(kind='barh', figsize=(11, 6),
color=features['positive'].map({True: 'blue', False: 'red'}))
plt.xlabel('Importance')
plt.show()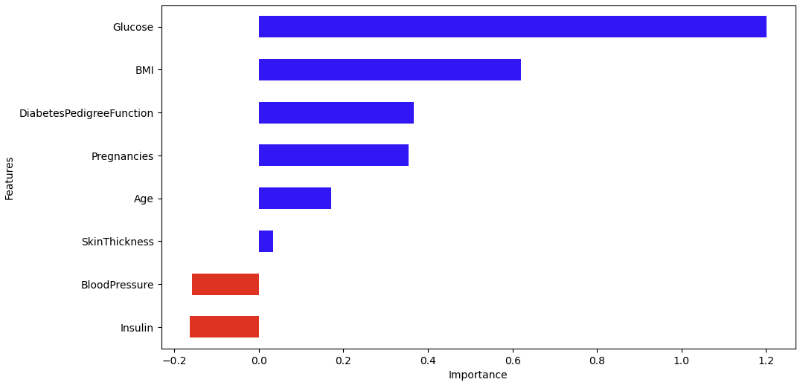
4. 정밀도와 재현율의 trade-off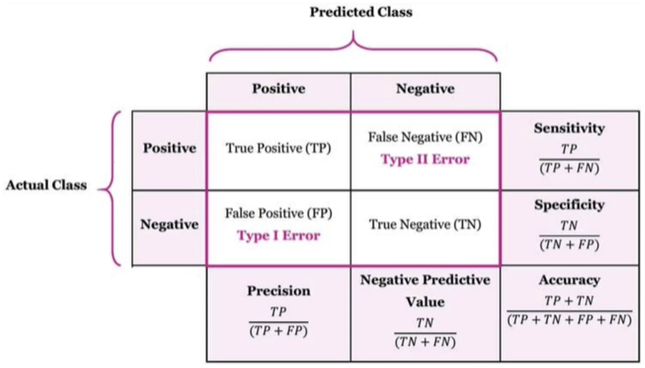
thresholds로 정밀도와 재현율을 바꿀 수 있지만, 이것이 모델의 성능을 향상시키지는 않는다. 더 많은 데이터가 들어올 때 작은 thresholds의 움직임은 큰 변화를 주지 않고, thresholds를 극단적으로 바꾸는 것도 좋지 않다. ➡ 잘 사용하지 않으나 방법을 알고 있자!
앞서 실습한 wine 데이터를 이용했다.
from sklearn.metrics import classification_report
print(classification_report(y_test, lr.predict(X_test)))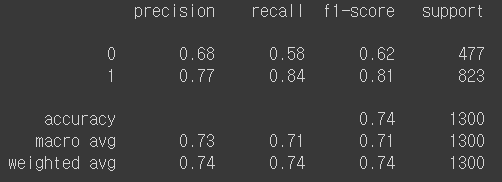
- quality > 5 ➡ 1
- precision(1) : 1이라고 예측한 것 중에서 1인 확률
- recall(1) : 실제 1인 것 중에서 1이라고 예측한 확률
- support : 개수
- macro avg : 클래스별 평균
- weighted avg : 클래스별 가중평균
from sklearn.metrics import confusion_matrix
confusion_matrix(y_test, lr.predict(X_test))
>>>
array([[275, 202],
[131, 692]])from sklearn.metrics import precision_recall_curve
pred = lr.predict_proba(X_test)[:, 1]
precisions, recalls, thresholds = precision_recall_curve(y_test, pred)
plt.figure(figsize=(10, 8))
# thresholds의 크기만큼만
plt.plot(thresholds, precisions[:len(thresholds)], label='precision')
plt.plot(thresholds, recalls[:len(thresholds)], label='recall')
plt.grid()
plt.legend()
plt.show()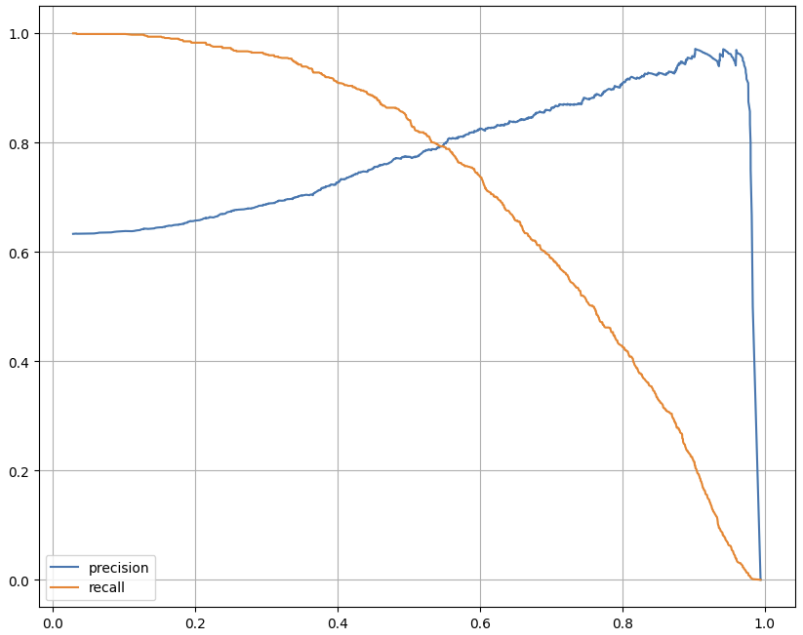
threshold 변경하기
# predict() ➡ threshold=0.5
pred_predict = lr.predict(X_test)
pred_predict
>>>
array([1, 0, 1, ..., 1, 0, 1])pred_proba = lr.predict_proba(X_test)
pred_proba
>>>
array([[0.40537064, 0.59462936],
[0.51023887, 0.48976113],
[0.10180603, 0.89819397],
...,
[0.22538943, 0.77461057],
[0.67355628, 0.32644372],
[0.31430379, 0.68569621]])from sklearn.preprocessing import Binarizer
binarizer = Binarizer(threshold=0.6).fit(pred_proba)
pred_bin = binarizer.transform(pred_proba)[:, 1]
pred_bin
>>>
array([0., 0., 1., ..., 1., 0., 1.])print(classification_report(y_test, pred_bin))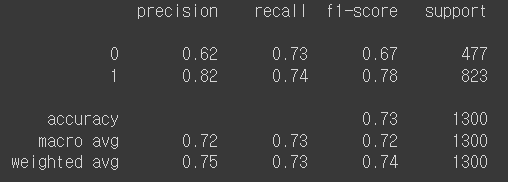
이전 결과(threshold=0.5)와 비교해보자.
5. 앙상블 기법
여러 개의 분류기를 생성하고 그 예측을 결합하여 최종 예측을 하는 방법
5-1. Voting & Bagging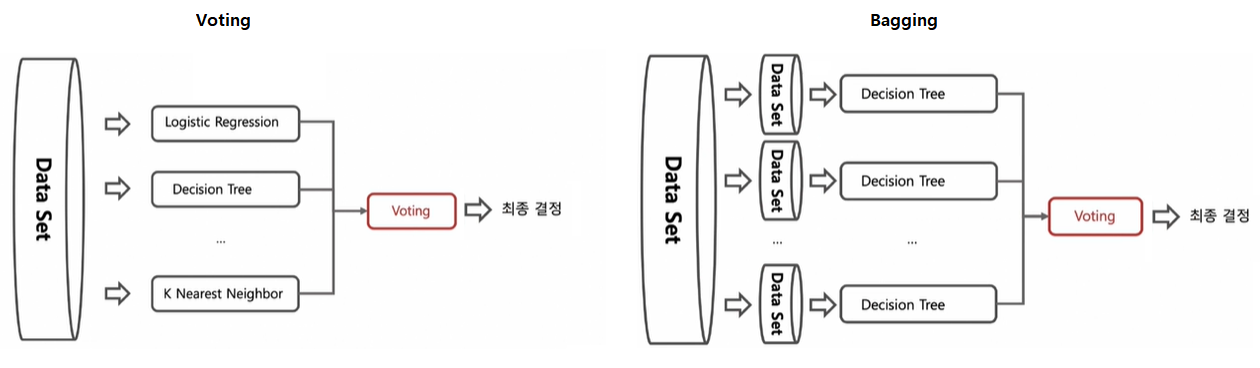
Bagging : 데이터를 중복을 허용하여 샘플링하고(Bootstrapping) 각각의 데이터에 같은 알고리즘을 적용
5-2. Soft Voting & Hard Voting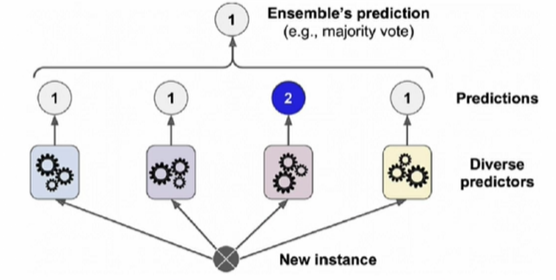
Soft Voting : 1과 2의 확률의 평균을 각각 구해서 비교한다.
Hard Voting : 다수결의 원칙과 비슷하다. 2를 제외한다.
5-3. 랜덤포레스트(Random Forest)
- 대표적인 Bagging 방법
- 부트스트래핑으로 샘플링된 각각의 데이터에 결정나무 알고리즘을 적용하여 얻은 결과를 소프트보팅하여 최종 결과를 얻음
- 앙상블 기법 중에서 비교적 속도가 빠르고, 다양한 영역에서 높은 성능을 보여줌
6. 실습 : UCI HAR(Human Activity Recognition)
- 머신러닝을 통한 행동인식연구 : 센서신호 ➡ 특징추출 ➡ 모델학습 ➡ 행동추론
- 허리에 스마트폰을 장착한 사람들의 행동을 관찰한 데이터
- 시간 영역 데이터를 머신러닝에 적용하기 위해 여러 통계적 데이터로 변환함
- 시간 영역의 평균/분산/피크/중간 값, 주파수 영역의 평균/분산 등으로 변환한 수치
6-1. 데이터 가져오기
feature_name_df = pd.read_csv(url, sep='\s+',
names=['column_index', 'column_name'])
feature_name_df.head()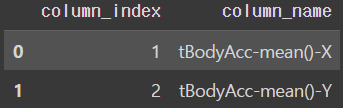
feature_name_df.info()
>>>
0 column_index 561 non-null int64
1 column_name 561 non-null objectfeature_name = feature_name_df.iloc[:, 1].values.tolist()X_train = pd.read_csv(X_train_url, sep='\s+', header=None)
X_test = pd.read_csv(X_test_url, sep='\s+', header=None)
X_train.columns = feature_name
X_test.columns = feature_name
y_train = pd.read_csv(y_train_url, sep='\s+', header=None, names=['action'])
y_test = pd.read_csv(y_test_url, sep='\s+', header=None, names=['action'])
X_train.shape, X_test.shape, y_train.shape, y_test.shape
>>>
((7352, 561), (2947, 561), (7352, 1), (2947, 1))y_train['action'].value_counts()
>>>
6 1407
5 1374
4 1286
1 1226
2 1073
3 9866-2. 결정나무 적용
dt_clf = DecisionTreeClassifier(random_state=13, max_depth=4)
dt_clf.fit(X_train, y_train)
pred = dt_clf.predict(X_test)
accuracy_score(y_test, pred)GridSearchCV를 이용하여 max_depth 다양하게 지정하기
from sklearn.model_selection import GridSearchCV
params = {
'max_depth': [6, 8, 10, 12, 16, 20, 24]
}
grid_cv = GridSearchCV(dt_clf, param_grid=params, scoring='accuracy',
cv=5, return_train_score=True)
grid_cv.fit(X_train, y_train)grid_cv.best_score_, grid_cv.best_params_
>>>
(0.8543335321892183, {'max_depth': 8})cv_results_df = pd.DataFrame(grid_cv.cv_results_)
cv_results_df[['param_max_depth', 'mean_test_score', 'mean_train_score']]
>>>
param_max_depth mean_test_score mean_train_score
0 6 0.843444 0.944879
1 8 0.854334 0.982692
2 10 0.847125 0.993369
3 12 0.841958 0.997212
4 16 0.841958 0.999660
5 20 0.842365 0.999966
6 24 0.841821 1.000000train과 test의 score 차이가 있다는 점을 주의하자.
# test 데이터 예측
max_depth = [6, 8, 10, 12, 16, 20, 24]
for depth in max_depth:
dt_clf = DecisionTreeClassifier(max_depth=depth, random_state=13)
dt_clf.fit(X_train, y_train)
pred = dt_clf.predict(X_test)
accuracy = accuracy_score(y_test, pred)
print(f'Max_depth={depth}, Accuracy={accuracy}')
>>>
Max_depth=6, Accuracy=0.8554462164913471
Max_depth=8, Accuracy=0.8734306073973532
Max_depth=10, Accuracy=0.8615541228367831
Max_depth=12, Accuracy=0.8595181540549711
Max_depth=16, Accuracy=0.8669833729216152
Max_depth=20, Accuracy=0.8652867322701052
Max_depth=24, Accuracy=0.8652867322701052test 데이터로 성능을 확인해봐도 Max_depth=8이 가장 좋다.
best_dt_clf = grid_cv.best_estimator_
pred1 = best_dt_clf.predict(X_test)
accuracy_score(y_test, pred1)
>>>
0.8734306073973532이렇게해도 같은 값을 얻을 수 있다.
6-3. 랜덤포레스트 적용
from sklearn.ensemble import RandomForestClassifier
params = {
'max_depth': [6, 8, 10],
'n_estimators': [50, 100, 200], # 나무 몇 그루 사용
'min_samples_leaf': [8, 12], # 맨 끝에 오는 leaf의 수
'min_samples_split': [8, 12] # 분할 기준에서 최소한으로 남는 데이터 수
}
rf_clf = RandomForestClassifier(random_state=13, n_jobs=-1) # n_jobs=-1 코어 다 사용
grid_cv = GridSearchCV(rf_clf, param_grid=params, cv=2, n_jobs=-1)
grid_cv.fit(X_train, y_train)cv_results_df = pd.DataFrame(grid_cv.cv_results_)
target_cols = ['rank_test_score', 'mean_test_score', 'param_n_estimators', 'param_max_depth']
cv_results_df[target_cols].sort_values('rank_test_score').head()
>>>
rank_test_score mean_test_score param_n_estimators param_max_depth
28 1 0.915125 100 10
25 1 0.915125 100 10
23 3 0.912813 200 8
20 3 0.912813 200 8
35 5 0.912541 200 10grid_cv.best_estimator_
>>>
RandomForestClassifier(max_depth=10, min_samples_leaf=8, min_samples_split=8,
n_jobs=-1, random_state=13)rf_clf_best = grid_cv.best_estimator_
rf_clf_best.fit(X_train, y_train)
pred1 = rf_clf_best.predict(X_test)
accuracy_score(y_test, pred1)
>>>
0.92059721750933156-4. 데이터 중요 특성 추출
best_cols_values = rf_clf_best.feature_importances_
best_cols = pd.Series(best_cols_values, index=X_train.columns)
top20_cols = best_cols.sort_values(ascending=False)[:20]
top20_colsimport seaborn as sns
plt.figure(figsize=(8, 8))
sns.barplot(x=top20_cols, y=top20_cols.index)
plt.show()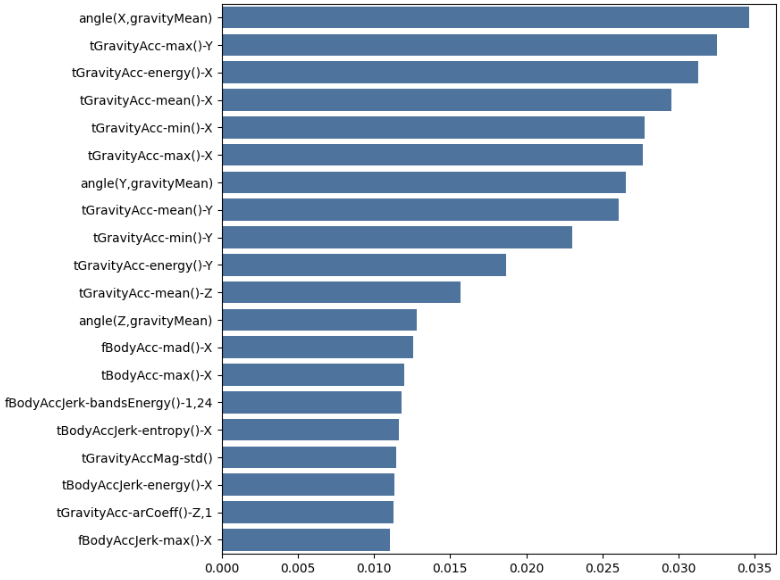
X_train_re = X_train[top20_cols.index]
X_test_re = X_test[top20_cols.index]
rf_clf_best_re = grid_cv.best_estimator_
rf_clf_best_re.fit(X_train_re, y_train.values.reshape(-1, ))
pred1_re = rf_clf_best_re.predict(X_test_re)
accuracy_score(y_test, pred1_re)561개 특성 중에서 20개 특성만 가지고 성능 확인해봐도 성능이 크게 나쁘지 않다.
상황에 맞게 선택하면 된다.
