오늘 리뷰할 논문은 ISBI cell tracking challenge 2015에서 우승한 U-Net 논문이다. U-Net이 stable diffusion에 쓰인다고 해서 호기심이 생겨 읽게 되었는데, 원래는 biomedical segmentation을 목적으로 만들어졌다.
아래 포스트를 먼저 읽으면 도움이 될 것이다.
Summary
U-Net 구조는 context를 capture하는 contracting path와 정교한 localization을 가능케 하는 (contract path에 대칭적인) expanding path로 구성된다.
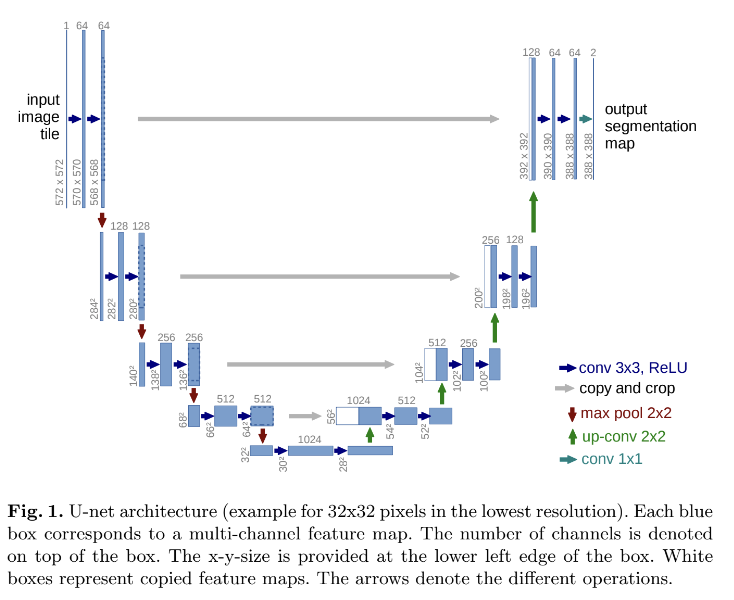
U-Net의 아이디어는 “fully convolutional network”에서 기반했는데, fully convolutional network는 일반적인 contracting network에 pooling operators 대신 upsampling operators를 사용하는, 연이은 layers를 추가해 보충한 것이다. 이런 layer들은 output의 resolution을 향상시킨다. localization을 위해서는, contracting path에서 온 high resolution features가 upsampled ouput과 조합된다. successive convolution layer가 이 정보를 바탕으로 더 정확한 output을 조립하도록 학습한다.
U-Net이 fully convolutional network와 다른 점은 upsampling 부분에서도 많은 수의 feature channels가 존재해 higher resolution layers로 context information을 전파할 수 있다는 것이다. 그래서 expansive path가 contracting path와 거의 대칭적이며, U자 모양 구조를 만든다.
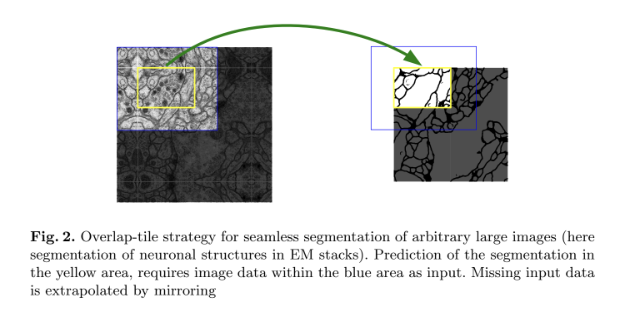
U-Net은 fully connected layers 없이 convolution만을 이용하며, resolution이 GPU memory에 제한되지 않고 큰 input image도 사용할 수 있게 Overlap-tile strategy를 사용한다. 이미지 가장자리에서 context가 missing할 때는 input image를 mirroring함으로써 extrapolate한다.
training data가 적기 때문에 data에 elastic deformations를 적용하여 excessive data augmentation를 한다. 이는 network가 그런 deformations(변형/기형)에 invariance을 배우게 하며, deformation은 tissue에서 가장 흔한 variation이기 때문에 biomedical segmentation에 중요하다.
cell segmentation tasks의 또 다른 과제는 같은 class의 인접한(touching) object를 구분하는 것이다. 이를 위해 weighted loss를 사용해 touching cells 간 background labels를 구분하는 것이 loss function에서 큰 weight를 가지게 했다.
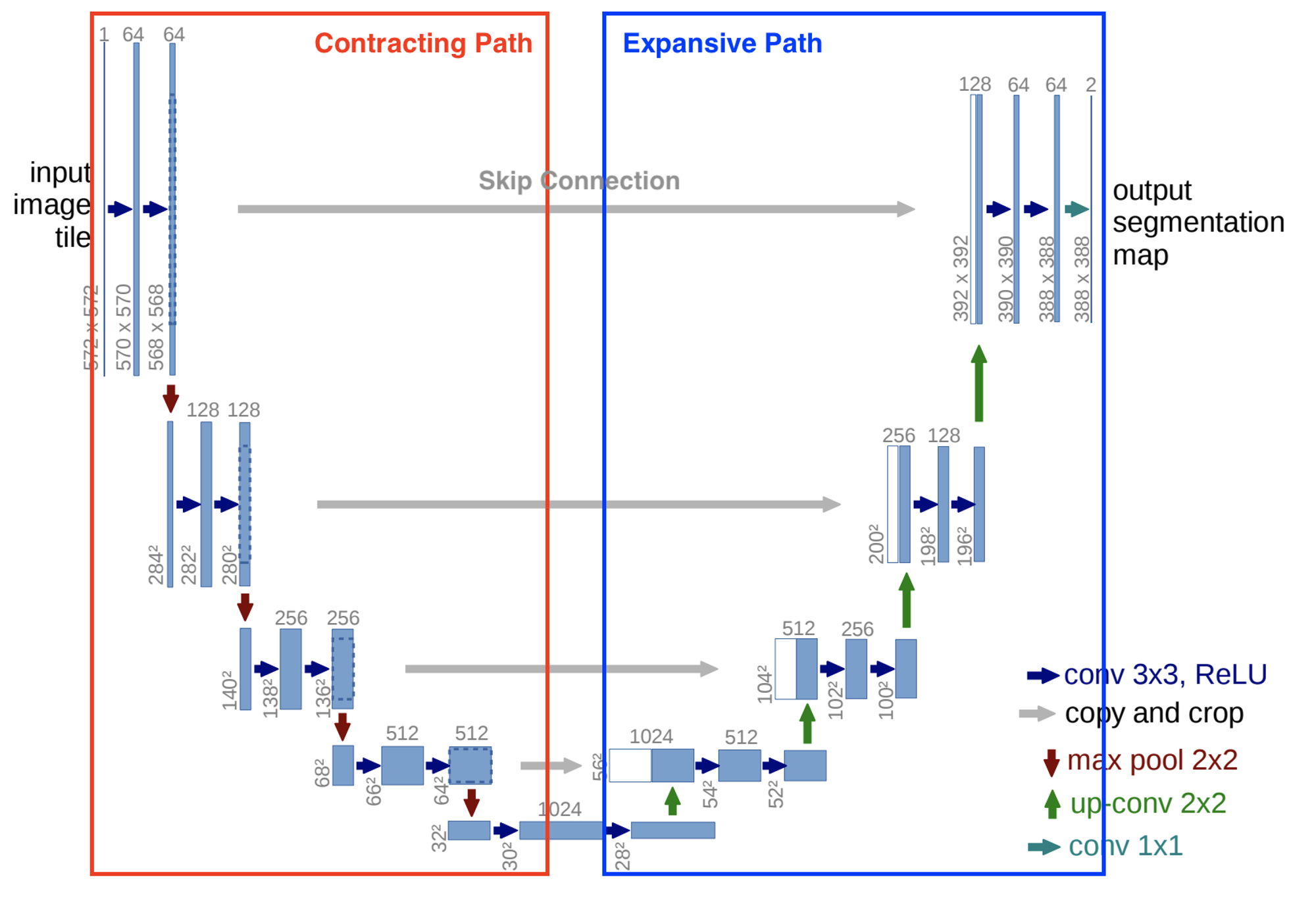
U-Net의 architecture을 살펴보자. 우선 contracting path는 '3x3 unpadded conv 2번(파란 가로 화살표), 2x2 max pool 1번(빨간 세로 화살표)'이 반복적으로 나타나며 downsampling마다 feature channel 수가 2배가 된다. expansive path에선 2x2 up-convolution(초록 세로 화살표)을 하는데 그떄마다 feature channel 수를 절반으로 줄인다. 그리고 상응하는 contracting path에서 온 cropped feature map과 concatenation을 한 후(회색 가로 화살표) 3x3 convolution 2번(파란 가로 화살표)을 한다. 모든 convolution마다 가장자리 pixel이 소실되기 때문에 cropping이 필수적이다. 마지막 1x1 convolution(하늘색 가로 화살표)은 64차원 feature vector을 원하는 숫자의 class로 mapping하기 위해 사용된다. network는 전체 23 convolution layer이다(파란색, 초록색, 하늘색 화살표). Figure 2와 같은 output segmentation map의 seamless tiling를 가능케 하기 위해 모든 2x2 max-pooling operations이 짝수 x, y size를 가진 layer에 적용되도록 input tile size를 선택하는 게 중요하다.
unpadded convolutions 때문에 output image(=Segmentation map)는 input보다 constant border width만큼 작다.
energy function은 조합된 final feature map에 대한 pixel-wise soft-max와 cross entropy loss function를 조합해 계산한다. softmax와 cross entropy loss function은 각각 아래와 같다.
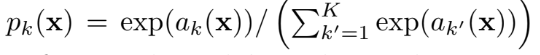
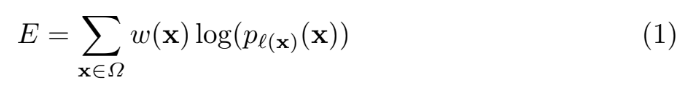
여기서 w는 특정 pixel에 더 중요도를 주기 위한 weight map인데, training set에서 특정 class에서 온 different frequency of pixels를 conpensate하기 위해서, 그리고 앞서 말했듯 touching cells 간 작은 경계를 학습하기 위해서 weight map을 사용한다. morphological operations을 이용해 separation border을 계산하며 weight map 식은 아래와 같다.
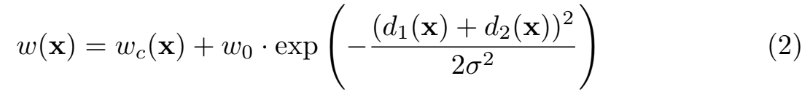
data augmentation은 shift와 rotation invariance, deformation과 gray value variations에 대한 robustness가 필요하다. 논문에선 특히 random elastic deformations을 하는데, coarse 3 by 3 grid에 random displacement vectors를 사용해 smooth deformations를 생성한다. contracting path 끝의 drop-out layers도 implicit한 data augmentation의 역할을 한다.
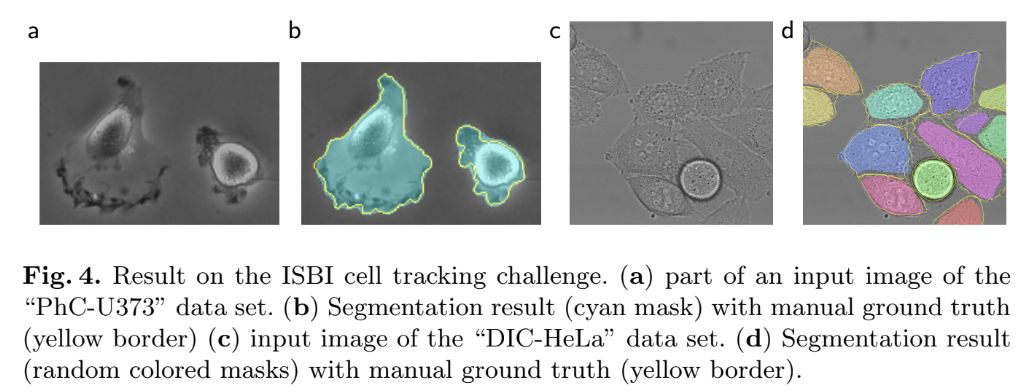
Strengths
- biomedical segmentation application이라는 목적에 맞게 잘 설계한 것 같다. touching boundary를 구분하기 위한 weight map이나 over-lap tile stratgey, random elastic deformations data augmentation이 모두 그런 노력의 일환이다.
- path를 contracting과 expansive으로 구분하고 copy and crop을 통한 skip connection이 성공적이었던 것 같다. channel을 늘리면서 downsampling하고 다시 줄이면서 upsampling하면서 원래 정보를 concatenate하는 게 성공의 핵심이었던 것 같다.
U-Net 구조가 특이해서 흥미롭게 읽었다. U-Net의 skip connection이나 resnet의 identity connection처럼 네트워크에 shortcut기능?을 넣는 게 전반적으로 성능에 좋은 영향을 줄 지도 모르겠다는 생각이 들었다.