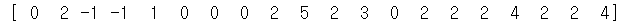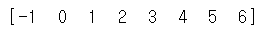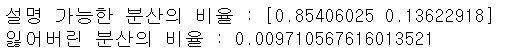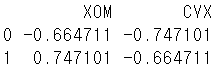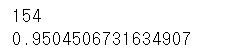✔ GMM(가우시안 혼합 모델)
- 타원형 데이터에는 잘 동작한다.
- 반달 모양의 데이터나 동심원 형태의 데이터에 취약합니다.
✔ DBSCAN
개요
- 국부적인 밀집도를 추정하는 매우 다른 방식을 사용합니다.
- 임의의 모양을 가진 클러스터를 식별할 수 있습니다.
- 간단하고 직관적인 알고리즘
ex
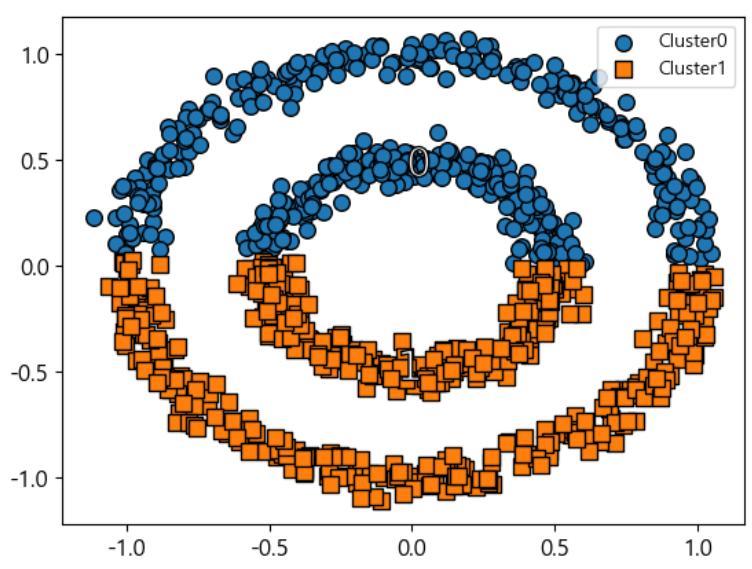
- 내부의 원 모양과 외부의 원 모양을 가진 형태의 데이터 분포의 경우 KNN, K-Means, GMM또는 계층적 클러스터링으로는 효과적인 군집화가 어렵지만 DBSCAN은 가능함
- DBSCAN은 특정 공간 내에 데이터 밀도 차이에 기반한 알고리즘 입니다.
알고리즘
- epsilon
- 주변 영역- min points
- 엡실론 영역에 포함되는 다른 데이터의 개수- Core Point(핵심 포인트)
- 주변 영역 내에 최소 데이터 개수 이상의 다른 데이터를 포함한 포인트- Neighbor Point(이웃 포인트)
- 주변 영역 내에 존재하는 다른 데이터- Border Point(경계 포인트)
- 주변 영역 내에 최소 데이터 개수 이상의 이웃 포인트를 가지고 있지 않지만, 핵심 포인트를 이웃으로 가지고 있는 데이터- Noise Point(잡음 포인트)
- 최소 데이터 개수 만큼의 이웃 포인트도 가지지 않고 햄심 포인트도 이웃 포인트로 가지지 않은 데이터
- 이상치 탐지의 원리
작동 방식
- 알고리즘이 각 샘플에서 epsilon 내에 샘플이 몇 개 놓였는지 세고, 이 지역을 샘플의이웃이라고 부릅니다.
- 이웃 내에 min_samples 개 이상의 데이터가 존재한다면, 핵심 포인트로 간주합니다.
- 핵심 샘플의 이웃에 있는 모든 샘플은 동일한 클러스터에 속하고, 이웃에는 다른 핵심 포인트가 포함될 수 있어서 핵심 포인트의 이웃의 이웃은 계속해서 하나의 클러스터를 형성
- 핵심 포인트도 아니고 이웃 포인트도 아닌 잡음 포인트는 이상치 처리
클래스
sklearn DBSCAN클래스 이용합니다.- 초기화 파라미터
- eps : 엡실론
- min_samples : 최소 이웃의 개수
- 반달 모양 데이터 군집 생성
from sklearn.datasets import make_moons X,y=make_moons(n_samples=1000,noise=0.05, random_state=42) plt.scatter(x=X[:,0], y=X[:,1])
- eps 0.05 min_samples를 5로 설정해서 수행
# eps 0.05 min_samples를 5로 설정해서 수행 from sklearn.cluster import DBSCAN # 모델 생성 및 훈련 dbscan=DBSCAN(eps=0.05,min_samples=5) dbscan.fit(X) # 군집된 결과 확인 (-1)은 노이즈 print(dbscan.labels_[:20])
- 핵심 포인트 확인하기
# 핵심 포인트의 개수 확인해보자. print(len(dbscan.core_sample_indices_)) # 핵심 포인트의 인덱스 print(dbscan.core_sample_indices_) # 핵심 포인트의 좌표 print(dbscan.components_)
- 실제 클러스터 확인하기
# 클러스터의 종류 확인하기 print(np.unique(dbscan.labels_))
- 이상치를 제외하고 0~6까지 총 7개의 군집으로 나누어졌다.
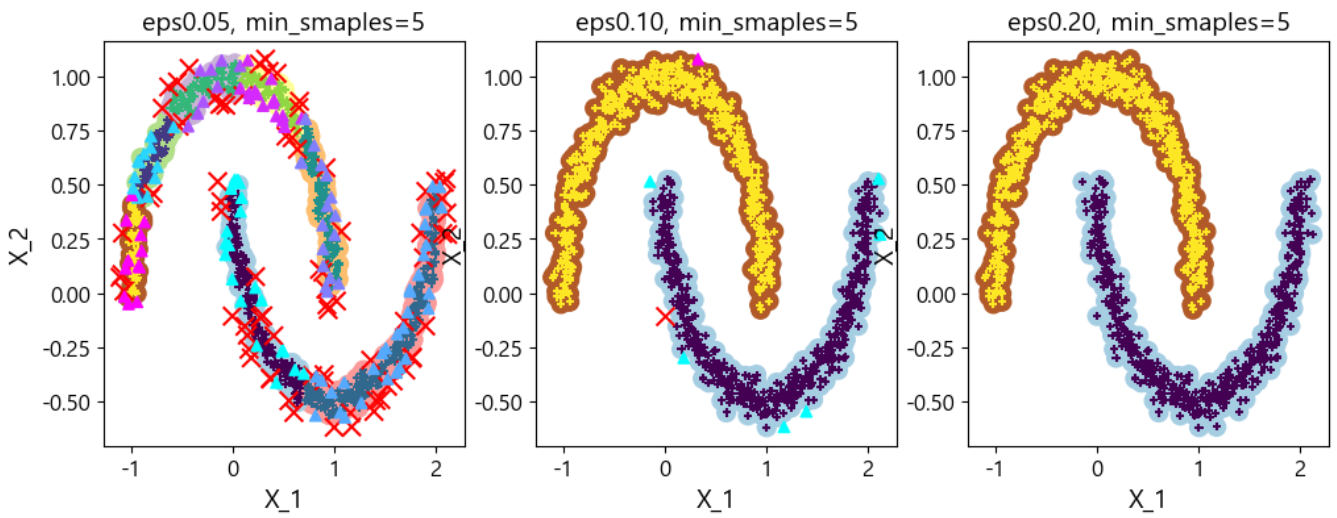
- eps 값을 조금씩 바꾸면서 결과를 다시 확인해보자.
- eps 값이 0.5가 된다면 label은 0, 1로 완벽하게 군집화가 됩니다.
- eps 값 변화하면서 확인하기
def plot_dbscan(dbscan, X, size, show_xlabels=True, show_ylabels=True): # 데이터 개수만큼 배열 만들고 핵심 포인트만 True 설정 core_mask=np.zeros_like(dbscan.labels_,dtype=bool) core_mask[dbscan.core_sample_indices_]=True # 이상치는 반대로 설정 anomalies_mask=dbscan.labels_==-1 # 이웃 포인트(핵심, 이상도 아닌 포인트) non_core_mask=~(core_mask | anomalies_mask) cores=dbscan.components_ anomalies=X[anomalies_mask] non_cores=X[non_core_mask] # plt.scatter(cores[:0],cores[:,1], c=dbscan.labels_[core_mask], # makrer='o', s=size, cmap='Paired') # plt.scatter(cores[:0],cores[:,1], c=dbscan.labels_[core_mask], # makrer='*', s=20) plt.scatter(cores[:, 0], cores[:, 1], c=dbscan.labels_[core_mask], marker='o', s=size, cmap='Paired') plt.scatter(cores[:, 0], cores[:, 1], c=dbscan.labels_[core_mask], marker='+', s=20) # plt.scatter(anomalies[:0],anomalies[:,1], c=dbscan.labels_[anomalies_mask], # makrer='x', s=100, cmap='r') # plt.scatter(non_cores[:0],non_cores[:,1], c=dbscan.labels_[non_core_mask], # makrer='^',cmap='g') plt.scatter(anomalies[:, 0], anomalies[:, 1], c=dbscan.labels_[anomalies_mask], marker='x', s=100, cmap='prism') plt.scatter(non_cores[:, 0], non_cores[:, 1], c=dbscan.labels_[non_core_mask], marker='^', cmap='cool') if show_xlabels: plt.xlabel("X_1",fontsize=14) else: plt.tick.params(labelbottom=False) if show_ylabels: plt.ylabel("X_2", fontsize=14) else: plt.tick.params(labelleft=False) plt.title("eps{:.2f}, min_smaples={}".format(dbscan.eps,dbscan.min_samples),fontsize=14)### 엡실론 조정 dbscan1=DBSCAN(eps=0.05, min_samples=5) dbscan2=DBSCAN(eps=0.1, min_samples=5) dbscan3=DBSCAN(eps=0.2, min_samples=5) dbscan1.fit(X) dbscan2.fit(X) dbscan3.fit(X) plt.figure(figsize=(12,4)) plt.subplot(131) plot_dbscan(dbscan1,X,size=100) plt.subplot(132) plot_dbscan(dbscan2,X,size=100) plt.subplot(133) plot_dbscan(dbscan3,X,size=100) plt.show()
예측
- DBSCAN은 predict 같은 예측 함수를 제공하지 않습니다.
- 군집한 뒤의 예측은 분류나 회귀 알고리즘이 더 잘하기 때문입니다.
- 군집 한 결과를 가지고 분류나 회귀 알고리즘을 수행한 뒤,
# 새로운 데이터에 대한 예측 from sklearn.neighbors import KNeighborsClassifier # 군집한 결과를 가지고 분류 모델이 훈련 knn=KNeighborsClassifier(n_neighbors=50) knn.fit(dbscan3.components_,dbscan3.labels_[dbscan3.core_sample_indices_])# 예측은 학습한 KNN으로 X_new=np.array([[-0.5, 0], [0,0.5]]) y_hat=knn.predict(X_new) print(y_hat) y_hat_proba=knn.predict_proba(X_new) print(y_hat_proba)
정리
- 매우 간단하지만 강력한 알고리즘
- 클러스터의 모양과 개수에 상관없이 감지할 수 있는 능력이 있음
- 이상치에 안정적이고 하이퍼 파라미터가 2개(eps, min_samples)
- 클러스터 간의 밀집도가 다르면, 모든 클러스터를 잡아내는 것이 거의 불가능
- 계산 복잡도는 O(mlogm) : m 데이터 개수
- sklearn에서는 데이터의 개수에 대해서는 선형으로 증가하지만, eps에 대해서는 o(m^2)만큼 메모리가 필요합니다.
✔ K-Means & GMM & DBSCAN
- K-Means는 Centroid 와의 거리를 이용
- 클러스터 간의 간격이 조금 멀 때 사용 - GMM은 모든 데이터의 분포를 정규 분포라고 가정
- 타원형 데이터에 적합 - DBSCAN은 밀집도 기반
- 데이터가 정규분포 형태가 아닐때
- 데이터의 분포가 연속적일 때
- 이상치 탐지를 합니다.
- 시각화 함수 - DB 클러스터링 결과 & DF
# K-Means때문에 istcenter를 넣었습니다. def visualize_cluster_plot(clusterobj,dataframe, label_name, iscenter=True): # 클러스터의 중앙점 찾기 if iscenter: centers=clusterobj.cluster_conters_ unique_labels=np.unique(dataframe[label_name].values) markers=['o','s','^','x','*'] # 이상치 여부 isNoise=False # 클러스터 순회 for label in unique_labels: label_cluster=dataframe[dataframe[label_name]==label] # 이 경우는 DBSCAN만 if label==-1: cluster_legend='Noise' isNoise=True else: cluster_legend='Cluster'+str(label) plt.scatter(x=label_cluster['ftr1'],y=label_cluster['ftr2'], s=70, edgecolor='k', marker=markers[label], label=cluster_legend) if iscenter: center_x_y=centers[label] plt.scatter(x=center_x_y[0],y=center_x_y[1],s=250, color='white',edgecolor='k',marker='$%d$'%label) if isNoise: legend_loc='upper center' else: legend_loc='upper right' plt.legend(loc=legend_loc) plt.show()
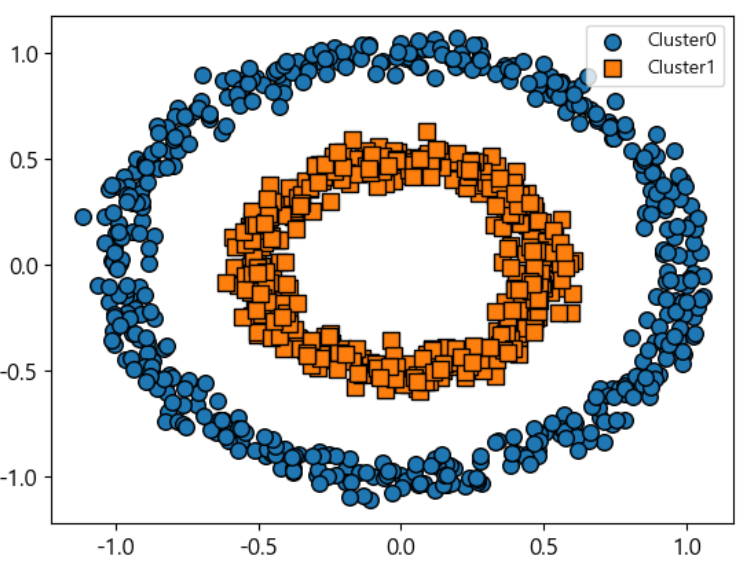
- 샘플데이터
from sklearn.datasets import make_circles X,y=make_circles(n_samples=1000, shuffle=True, noise=0.05, random_state=42, factor=0.5) clusterDF=pd.DataFrame(data=X, columns=['ftr1','ftr2']) clusterDF['target']=y print(np.unique(clusterDF['target'])) visualize_cluster_plot(None, clusterDF, 'target', iscenter=False)
- K-Means는 어떻게 처리 할까?
from sklearn.cluster import KMeans kmeans=KMeans(n_clusters=2, max_iter=1000, random_state=42) kmeans_labels=kmeans.fit_predict(X) clusterDF['kmeans_cluster']=kmeans_labels visualize_cluster_plot(kmeans, clusterDF, 'kmeans_cluster', iscenter=True)
- centroid 계산으로 반으로 나누어졌다.
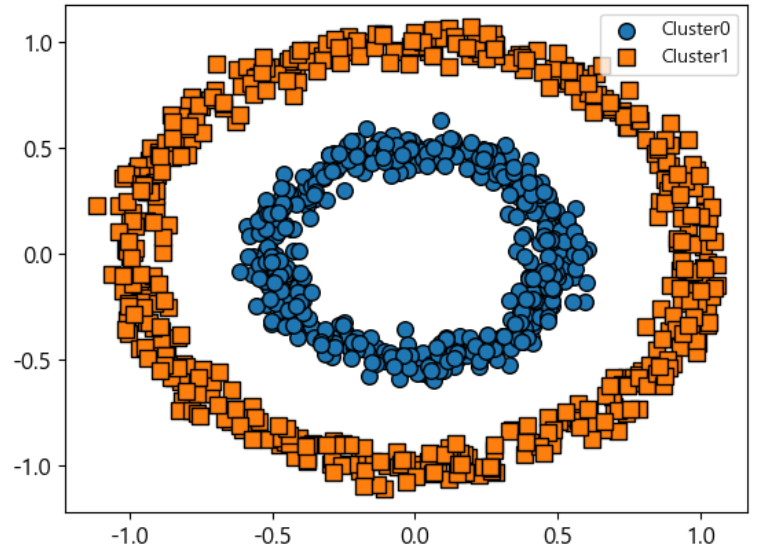
- GMM은 어떻게 처리할까?
from sklearn.mixture import GaussianMixture gmm=GaussianMixture(n_components=2, random_state=42) gmm_label=gmm.fit(X).predict(X) clusterDF['gmm_cluster']= gmm_label visualize_cluster_plot(gmm, clusterDF, 'gmm_cluster', iscenter=False)
- 타원형으로 처리하더니, 결국 대각선 방향으로 반을 갈라버렸다.
- 이제 DBSCAN을 봐보자.
from sklearn.cluster import DBSCAN dbscan=DBSCAN(eps=0.2, min_samples=7) dbscan_labels=dbscan.fit_predict(X) clusterDF['dbscan_cluster']= dbscan_labels visualize_cluster_plot(dbscan, clusterDF, 'dbscan_cluster', iscenter=False)
- 이쁘게 분류했다.
✔ BIRCH
- Balanced Iterative Reducing and Clustering
- 대규모 데이터 세트를 위해서 개발
- 특성 개수가 많지 않은 경우(20개 이하), K-Means보다 빠르고 비슷한 결과를 생성합니다.
- 훈련 과정에서 새로운 샘플을 클러스터에 빠르게 할당할 수 있는 정보를 담은 트리구조를 이용합니다.
- 제한된 메모리를 사용해서 대용량 데이터 세트를 다룰 수 있음
장점
- 한 번의 스캔으로 클러스터링
단점
- 숫자 데이터만 다룰 수 있고, 순서에 민감합니다.
from sklearn.datasets import load_iris iris=load_iris() X=iris.data from sklearn.cluster import Birch brc=Birch(n_clusters=3) brc.fit(X)print(iris.target) y_hat=brc.predict(X) print(y_hat)
✔ 유사도 전파
- 투표 방식을 이용
- 각 데이터는 자신을 대표할 수 있는데 비슷한 샘플에 투표
- 알고리즘을 수행하다가 수렴된다면, 각 대표와 투표한 샘플이 클러스터를 형성
- 크기가 다른 열러 개의 클러스터를 감지할 수 있지만, 알고리즘의 계산 복잡도가 O(m&2)이라서 대규모 데이터 세트에는 적합하지 않음
✔ 스펙트럼 군집
- 샘플의 유사도 행렬을 받아서 저차원 임베딩을 생성
- 차원 축소 - 축소된 저차원 공간에서 다른 군집 알고리즘을 수행(sklearn의 경우는 k-Means)
- 복잡한 클러스터 구조를 잘 감지합니다.
- 샘플의 개수가 많거나 클러스터의 크기가 너무 다르면 잘 동작하지 않음
✔ 이상치 탐지와 특이치 탐지를 위한 알고리즘
군집 알고리즘
- 군집을 했을 때, 잘 어울리지 못하는 데이터
PCA(주성분 분석)
- 샘플의 재구성 오차와 이상치의 재구성 오차를 비교하면, 후자가 훨씬 큼
Fast-MCD(Minimum Covariance Determinant)
- 데이터 세트를 정제할 때, 일반적인 데이터는 하나의 가우시안 분포에서 생성되었다고 가정하며, 이 가우시안 분포에서 생성되지 않은 데이터를 이상치로 간주
- 타원형을 잘 감지한다
아이솔레이션 포레스트
- 고차원 데이터 세트에서 이상치 감지를 위한 효과적인 알고리즘
- 무작위로 성장한 결정 트리로 구성된 랜덤 포레스트를 생성하고, 각 노드에서 특성을 랜덤하게 선택하고, 임계값을 설정하고 데이터 세트를 나누는 방식
LOG(Local Outlier Factor)
- 일반적인 데이터의 주위의 밀도와 이웃 주위의 밀도를 비교해서 격리된 정도를 확인
one-class SVM
- 하나의 평면을 이용해서 이 평면에 놓아지지 않는 데이터를 이상치로 간주
- 대규모 데이터 세트에 사용하면 시간이 오래 걸림
✔ 비지도 학습에서의 전처리
-
비지도 학습에서는 데이터를 적절하게 스케일링을 해야 합니다.
-
데이터 크기가 조정되지 않으면, PCA, K-Means 등은 큰 값을 갖는 변수들에 의해 결과가 좌우되고, 작은 값을 갖는 변수들은 무시되는 경우가 발생합니다.
-
범주형 데이터를 사용하게 된다면 KNN과 K-Means에서는 문제가 발생할 수 있습니다.
- 순서가 없는 데이터들은 원 핫 인코딩을 하게 되는데, 이렇게 되면 다른 feature와 크기가 다를 수 있고, 2가지 값만 가질 수 있기 때문에 영향을 많이 미치게 될 수 있습니다.
✔ 차원 축소 (Demesionality Reduction)
개요
- Machine Learning 문제들은 많은 특성을 소유하고 있는 경우가 많습니다.
- 특성이 많다면 훈련을 느리게 할 뿐 아니라, 좋은 솔루션을 찾기가 어려워 집니다.
- 이를 차원의 저주라고 합니다.
종류
- feature selection
- 특정 피처에 종속성이 강한 불필요한 featrue 는 제거하고, 데이터의 특성을 잘 나타내는 주요 feature만 선택 - feature extraction
- 기존들의 feature들을 저차원의 중요 feature로 압축해서 추출
- 기존 feature가 압축된 것이기 때문에 기존 feature와는 다른 값
- 더 함축적인 요약 특성으로 추출
주의
- 차원 축소를 수행하면, 일부 정보가 유실되기에, 훈련 속도는 빨라지지만, 시스템의 성능이 조금 나빠질 수 있으며, 파이프라인이 복잡해짐
- 이미지나 텍스트에서 차원 축소를 통해, 잠재적인 의미를 찾을 수 있습니다.
- 매우 많은 픽셀로 이루어진 이미지 데이터에서 모든 차원을 전부 활용해서 이미지 분류를 하게 된다면, 과대 적합될 가능성이 있기 때문에 차원을 축소하는 것이 예측 성능에 훨씬 도움이 될 수 있습니다.
차원의 저주
- 고차원 공간에서는 많은 것이 상당히 다르게 작동
- 단위 면적(1*1 사각형) 안에 존재하는 점을 무작위로 선택한 경우, 경계선에서 0.001 내에 위치할 가능성은 0.4% 정도 되지만, 차원이 높아지면 이 가능성은 매우 높아진다.
- 차원이 높아지면 데이터 사이의 거리도 멀어지게 됩니다.
- 차원이 높아지면 데이터 사이의 거리가 멀어진다는 것은, 예측해야 하는 새로운 데이터와 모델을 생성할 때, 사용한 훈련 데이터와의 거리도 멀어질 가능성이 높다라는 것을 의미
- 거리가 멀어지면 외삽(Extrapolation - 보외법)을 훨씬 더 많이 수행해야 해서 불확실성이 높아집니다.
- 보간은 두 개의 값 사이를 추정하기는 하는데, 최소값과 최대값 사이의 중간 값들을 유추
- 외삽은 다른 값들 사이의 관계에 기초에서 예측 - 차원이 높아지면 아주 많은 샘플이 필요합니다
투영
- 차원이 많은 경우, 거의 대부분은 특성은 변화가 없고, 다른 특성들이 서로 강하게 연관되어 있다라는 가정
- 스위스 롤 같은 문제가 발생
Manifold 학습
- 고차원 공간에서 휘어지거나 뒤틀린 모양
PCA(Principa Component Analysis) - 주성분 분석
- 여러 변수 간에 존재하는 상관 관계를 이용해서 이를 대표하는 주성분을 추출해 차원을 축소하는 방법
- 일반적으로 변수들은 공변(covary - 같이 변하는 성질)하므로 이러한 공변이 어떻게 일어나는지 알아내는 기법
분산 보존
- 여러 개의 축을 더 적은 개수의 축으로 변환을 할 때 분산의 값이 큰 방향으로 투영을 합니다.
- 분산의 값이 큰 방향으로 투영해야 정보가 가장 적게 손실되기 때문입니다.
- 분산 보존의 원리를 적용해서 만든 축을 주성분이라고 합니다.
- 이 주성분은 원점에 맞추어진 단위 벡터 형태로 표현하기 때문에 방향은 +나 - 방향이 될수 있음 - 선형 대수 입장에서 보면 PCA는 입력 데이터의 공분산 행렬을 고유 값 분해하고 이렇게 구한 고유 벡터에 입력 데이터를 선형 변환한 것이고, 이 고유 벡터(eigen vector)가 PCA의 주성분 벡터
- 이 벡터의 크기를 고유 값(eigen value)이라고 합니다.
- 행렬 분해를 이용해서 표현합니다.
수행 순서
- 입력 데이터 세트의 공분산 행렬을 생성
- 공분산 행렬의 고유 벡터와 고유 값을 계산합니다.
- 고유 값이 큰 순서대로 k(PCA의 차수)개 만큼 고유 벡터 추출
- 고유 값이 큰 순서대로 추출된 고유 벡터를 가지고 새로운 입력 데이터를 추출합니다.
SVD(특이값 분해)
- 고차원의 데이터를 저차원으로 분해하는 기법
- 추천 시스템을 만들 때 많이 활용합니다.
- numpy에서 제공합니다.
numpy.linal.svd함수
np.random.seed(42) m=60 angles=np.random.rand(m)*3*np.pi/2-0.5 X=np.empty((m,3)) # print(X) X_centered=X-X.mean(axis=0) # 특이값 분해 수행 U,s,Vt=np.linalg.svd(X_centered) # 주성분 c1=Vt.T[:,0] c2=Vt.T[:,1] print(c1) print(c2) # 실제 변환된 결과 W2=Vt.T[:,:2] X2D=X_centered.dot(W2) print(X2D)
- 이걸 직접 작업할 필요는 없고 sklearn의 PCA 클래스가 SVD를 이용한 주성분 분석을 구현하고 있다.
n_componentes파라미터에 원하는 주성분의 개수만 설정하면 됩니다.np.random.seed(42) m=60 angles=np.random.rand(m)*3*np.pi/2-0.5 X=np.empty((m,3)) # print(X) X[:,0]=np.cos(angles)+np.sin(angles)/2+0.1*np.random.randn(m)/2 # 0.1, 0.7은 잡음을 섞이 위한 것이라 아무 숫자나 가능 X[:,1]=np.sin(angles)*0.7+0.1*np.random.randn(m)/2 # 잡음 처리 해주기 X[:,2]=X[:,0]*0.1+X[:,1]*0.3+0.1*np.random.randn(m) X_centered=X-X.mean(axis=0) # sklearn from sklearn.decomposition import PCA # 2개의 주성분을 추출해주는 PCA 인스턴스 pca=PCA(n_components=2) # 데이터를 2차원으로 줄이기 X2D=pca.fit_transform(X) print(X2D)
- 훈련을 하고 난 뒤, PCA 인스턴스의
explained_variance_ratio_속성에 설명 가능한 분산의 비율을 저장합니다.- 이 개수는 주성분의 개수입니다.
# 분산의 비율 print("설명 가능한 분산의 비율 :",pca.explained_variance_ratio_) # 첫 번째 주성분이 0.854 만큼의 분산을 설명 # 두 번째 분산은 0.136만큼의 분산을 설명 # 두개의 분산이 있으면 0.99만큼의 분산을 설명하는 것 # 데이터 유실 print("잃어버린 분산의 비율 :", (1-pca.explained_variance_ratio_.sum()))
- PCA 인스턴스의
invers_transform함수를 이용하면 원래의 데이터로 복원이 가능합니다.
- 완전 복원은 불가능합니다.
- 분산이 100%가 아니기 때문입니다.np.random.seed(42) m=60 angles=np.random.rand(m)*3*np.pi/2-0.5 X=np.empty((m,3)) X[:,0]=np.cos(angles)+np.sin(angles)/2+0.1*np.random.randn(m)/2 # 0.1, 0.7은 잡음을 섞이 위한 것이라 아무 숫자나 가능 X[:,1]=np.sin(angles)*0.7+0.1*np.random.randn(m)/2 # 잡음 처리 해주기 X[:,2]=X[:,0]*0.1+X[:,1]*0.3+0.1*np.random.randn(m) X_centered=X-X.mean(axis=0) print(X[:5]) # sklearn from sklearn.decomposition import PCA # 2개의 주성분을 추출해주는 PCA 인스턴스 pca=PCA(n_components=2) # 데이터를 2차원으로 줄이기 X2D=pca.fit_transform(X) # print(X2D) # 2차원으로 줄어든 data를 복원하고 싶다. X3D_inverse=pca.inverse_transform(X2D) print(X3D_inverse[:5])
- 차이가 존재하긴 하지만, 엄청 큰 차이는 아닌가..?
# 2개가 배열이 같은지 확인 print(np.allclose(X,X3D_inverse))
- return은 boolean
# 오차의 평균 print(np.mean(np.sum(np.square(X3D_inverse - X), axis=1))) # 각 행의 오차 구하기 -이를 이용해서 이상치 탐지 가능 print(np.sum(abs(X3D_inverse-X), axis=1)) # 주성분 확인 print(pca.components_)
주식 데이터를 이용한 주성분 분석
- 주성분은 분산의 크기를 큰 방향으로 생성
- 분산 보존 법칙
- Data Load
# gz-> 압축 sp500_px=pd.read_csv('./data/sp500_data.csv.gz',index_col=0) oil_px=sp500_px[['XOM','CVX']] oil_px.head()
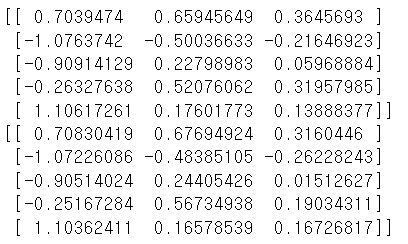
- 주성분 분석의 결과를 DataFrame으로 생성해서 출력
pca=PCA(n_components=2) pca.fit(oil_px) loadings=pd.DataFrame(pca.components_,columns=oil_px.columns) print(loadings)
- 결과 해석
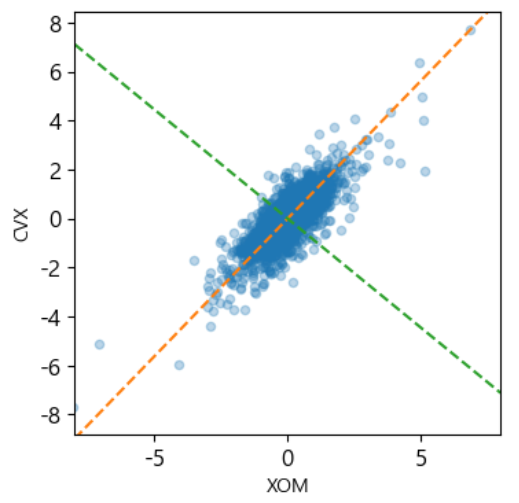
- 2개의 주성분으로 생성한 경우, 첫 번째 성분은 상관관계를 의미하는 계수이고, 두 번째 성분은 값이 달라지는 지점을 의미합니다.- 주성분을 시각화
# 주성분 시각화 plt.scatter(oil_px['XOM'],oil_px['CVX'])
def abline(slope, intercept, ax): x_vals=np.array(ax.get_xlim()) return(x_vals,intercept+slope*x_vals) ax=oil_px.plot.scatter(x="XOM", y='CVX', alpha=0.3, figsize=(4,4)) ax.set_xlim(-8,8) ax.set_ylim(-8.8) ax.plot(*abline(loadings.loc[0,'CVX']/loadings.loc[0,'XOM'],0,ax), '--',color='C1') # C2 녹색 ax.plot(*abline(loadings.loc[1,'CVX']/loadings.loc[1,'XOM'],0,ax), '--',color='C2') plt.show()
- 여러 개의 속성으로 확장하는 것이나 여러 개의 주성분을 추출하는 것도 가능
- 주성분 분석은 수치형 데이터에만 사용 가능합니다
- 범주형, 문자열 그리고 날짜 타입은 분산이 근본적으로 없기에 사용 불가능 합니다.
- 범주형이나 문자열은 개수나 분포의 비율이 의미를 갖는 데이터이지, 평균이나 합계를 구하는 데이터가 아닙니다.
주성분에서의 중요도
- 주성분의 상대적인 중요도를 표시해주는 시각화 방법을
scree plot이라고 합니다. - 상위 주성분들의 중요도를 파악해서 피처를 제거하고 분류나 회귀를 적용하는 것도 좋은 방법 중의 하나가 됩니다.
- feature의 중요도를 파악해보자.
syms=sorted(['AAPL','MSFT','CSCO','INTC','CVX', 'XOM','SLB','COP', 'JPM','WFC','USB', 'AXP','WMT','TGT','HD','COST']) top_sp=sp500_px.loc[sp500_px.index>='2011-01-01',syms] top_sp.head()# feature의 중요도를 파악할 것이라서 주성분의 개수를 설정하지 않음 sp_pca=PCA() sp_pca.fit(top_sp)# 설명 가능한 분산의 비율을 확인 print(sp_pca.explained_variance_)explained_variance=pd.DataFrame(sp_pca.explained_variance_) ax=explained_variance.head(10).plot.bar(legend=False, figsize=(4,4)) ax.set_label('COMPONANT') plt.show()# 5개의 주성분에 미치는 중요도를 DF로 생성3
적절한 차원 수 선택
- 데이터 시각화를 위해서 차원을 축소할 때는 2개나 3개로 축소합니다.
- 그래프는 2차원이나 3차원만 표시 가능 - 차원수는 충분한 분산 비율을 설명될 때 까지 더해가면서 설정
- PCA 인스턴스를 만들 때 n_components에 0.0에서 1.0 사이의 숫자를 설정하면, 설명 가능한 분산 비율이 적용되어서 PCA를 수행
- 차원 수에 대한 분산 설명 비율을 그래프로 그려서 elbow를 찾아서 설정하기도 합니다.
MNIST 이미지 데이터에서 적절한 차원 수 찾기 - 압축해서 예측 작업에 사용
# 데이터 로드 from sklearn.datasets import fetch_openml from sklearn.model_selection import train_test_split mnist=fetch_openml('mnist_784', version=1, as_frame=False) mnist.target=mnist.target.astype(np.uint8) X=mnist['data'] y=mnist['target'] X_train,X_test,y_train,y_test=train_test_split(X,y) print(X_train.shape)
- 출력을 보면 784차원이다.
- 적절한 차원의 개수를 찾기 위해서 차원의 개수를 설정하지 않고 분산의 비율보다 큰 개수를 찾기 (권장 X)
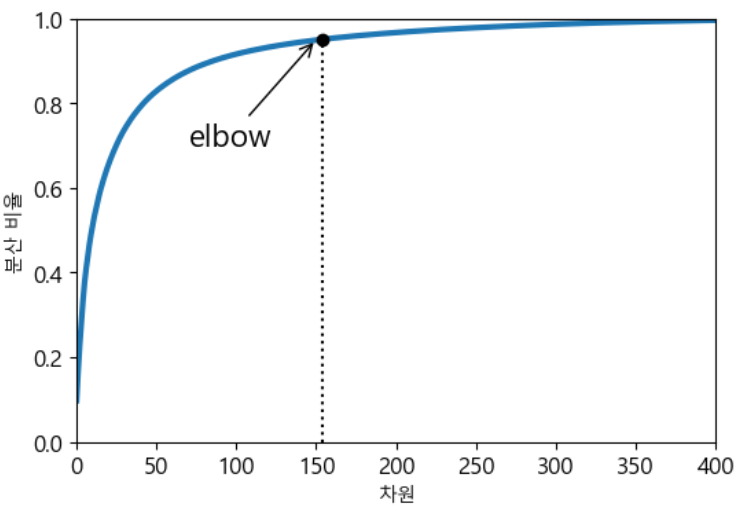
pca=PCA() pca.fit(X_train) cumsum=np.cumsum(pca.explained_variance_ratio_) d=np.argmax(cumsum>=0.95)+1 print(d)
- 154개가 나왔다.
- 방법 2 - 결과를 시각화해서 elbow를 찾는 방식(그다지 권장 X)
plt.figure(figsize=(6,4)) plt.plot(cumsum,linewidth=3) plt.axis([0,400,0,1]) plt.xlabel('차원') plt.ylabel('분산 비율') plt.plot([d,d],[0,0.95],'k:') plt.plot([d,d],[0.95,0.95],'k:') plt.plot(d,0.95,'ko') plt.annotate("elbow", xy=(150,0.95), xytext=(70, 0.7), arrowprops=dict(arrowstyle="->"), fontsize=16) plt.show()
- 방법 3 : 분산비율이 0.95이상인 PCA 만들면 된다.(만들때 집어넣기)(권장)
pca=PCA(n_components=0.95) X_reduced=pca.fit_transform(X_train) print(pca.n_components_) print(np.sum(pca.explained_variance_ratio_))
- 빠르게 잘 나온다.
차원 압축을 해볼거에요
- 차원을 축소하면 데이터의 크기가 줄어듦
- 분산 크기 95% 유지시 차원 개수가 154개
- 분산은 유지되고 데이터의 크기는 20% 정도로 압축 - 압축을 하면 SVM같은 알고리즘에서 속도가 향상
inverse_transform이라는 함수를 이용하면 원래의 차원으로 복원
- 투영 과정에서 일정량의 정보를 유실했기 때문에, 원본과 동일하지는 않음- 원본 데이터와 재구성된 데이터(압축 하고 복원된 데이터) 사이의 평균 제곱 거리를 재구성 오차(Reconstruction Error)라고 합니다.
- 복원한 이미지와 원래 이미지
pca=PCA(n_components=154) X_reduced=pca.fit_transform(X_train) X_recovered=pca.inverse_transform(X_reduced) print(X_recovered.shape)
- shape으로 잘 복원되었는지 확인할 수 있다.
def plot_digits(instances, images_per_row=5, **options): size=28 # 한 줄에 출력할 이미지 개수 설정 image_per_row=min(len(instances),images_per_row) # 1차원 이미지를 2차원으로 재구성하기 images=[instances.reshape(size,size) for instances in instances] # 행의 개수 계산 n_rows=(len(instances)-1)//images_per_row+1 row_images=[] n_empty=n_rows*images_per_row-len(instances) images.append(np.zeros((size*size*n_empty))) for row in range(n_rows): rimages=images[row*images_per_row : (row+1)*images_per_row] row_images.append(np.concatenate(rimages,axis=1)) image=np.concatenate(row_images, axis=0) plt.imshow(image, cmap=mpl.cm.binary, **options) plt.axis('off')plt.figure(figsize=(7,4)) plt.subplot(121) plot_digits(X_train[::2100]) plt.title("원본 이미지", fontsize=16) plt.subplot(122) plot_digits(X_recovered[::2100]) plt.title("압축 이미지", fontsize=16) plt.show()
- 손실이 크게 난것 같지는 않은데...
랜덤 PCA
- PCA 인스턴스를 만들 때,
svd_solver매개변수에 randomized를 설정하면, sklearn은 랜덤 PCA를 수행하는데, 랜덤 PCA는 처음 몇개의 주성분에 대한 근사값을 빠르게 찾는 알고리즘이다. - 시간 복잡도가 훨씬 줄어듭니다.
- 완전한 SVD를 이용하는 방식은 O(m*n^2) +O(n^3) 이다.
- 랜덤 PCA는 O(m*주성분개수^2)+O(주성분개수^3) 이다.
- 당연히 주성분 개수가 feature 개수보단 적기 때문에 빨라질 수 밖에 없다. - sklearn의 PCA의
svd_solver는 기본 값 auto인데, atuo는 주성분 개수나 데이터의 개수나 차원의 개수의 80%보다 작으면 랜덤 PCA를 수행하고, 그렇지 않으면 완전 SVD 방식을 사용 - 완전한 SVD 수행을 할 것이라면
svd_solver에 full을 설정하면 된다.
%%time pca=PCA(n_components=154, svd_solver='full') X_reduced=pca.fit_transform(X_train)
%%time pca=PCA(n_components=154, svd_solver='randomized') X_reduced=pca.fit_transform(X_train)
- 확실하게 시간 차이가 난다.
%%time pca=PCA(n_components=154) X_reduced=pca.fit_transform(X_train)
- 80% 밑이기에 auto 는 randomized가 될 것이다.
점진적 PCA
- PCA의 또 다른 문제점은 SVD 알고리즘을 수행하기 위해서, 전체 데이터를 메모리에 로드하고 수행해야 한다.
- 점진적 PCA(Incremental PCA - IPCA)는 훈련 데이터를 미니 배치(mini batch)로 나눈 뒤, PCA 알고리즘에 한 번에 하나의 배치를 주입하고 훈련을 하는 방식
- 데이터가 아주 클 때, 온라인 학습(데이터가 수시로 대입되는 경우)을 할 때 나눠서 학습하면 매우 좋다.
- numpy의 memmap이라는 클래스를 사용하면, 하드 디스크의 이진 파일에 저장된 배열을 메모리에 들어있는 것 처럼 사용할 수 있는데, 이 클래스는 필요할 때 데이터를 메모리에 적재함.
- 이것이 IPCA랑 동일한 방식으로 수행 - sklearn에서는
IncrementalPCA클래스를 이용해서 이 기능을 제공합니다.
- 데이터를 나누어서 훈련하고 예측을 할 수 있습니다.