
훈련 세트와 테스트 세트
머신러닝은
지도학습, 비지도학습, 강화학습 으로 나누어진다 !
지도학습
: 정답(타깃)이 있어서 알고리즘이 정답을 맞히는 것을 학습한다
비지도학습
: 타깃 없이 입력 데이터만 사용한다 = 정답을 사용하지 않으므로 무언가를 맞힐 수가 없지만 데이터를 잘 파악하거나 변형하는 데에 도움을 줌
훈련 세트와 테스트 세트
머신러닝 알고리즘의 성능을 평가하려면 훈련 데이터와 평가에 사용할 데이터가 각각 달라야한다 !
이를 위해서는 일반적으로 이미 준비된 데이터 중에 일부를 떼어 내어 활용을 하고 평가에 사용하는 데이터를 테스트 세트, 훈련에 사용하는 데이터를 훈련 세트라고 부른다 (훈련에 사용한 데이터로 모델을 평가하는건 좋지 않다, 일부를 뗴어내어 테스트 세트로 사용해야 한다)
from sklearn.neighbors import KNeighborsClassifier
kn = KNeighborsClassifier()print(fish_data[4]) # [29.0, 430.0]print(fish_data[0:5]) # [[25.4, 242.0], [26.3, 290.0], [26.5, 340.0], [29.0, 363.0], [29.0, 430.0]]train_input = fish_data[:35]
train_target = fish_target[:35] # 0~34 까지 샘플 = 훈련 세트
test_input = fish_data[35:]
test_target = fish_target[35:] # 35~48 까지 샘플 = 테스트 세트kn = kn.fit(train_input, train_target)
kn.score(test_input, test_target) # 0.0ㄴ 정확도가 0.0이다 왤까?
: 마지막 14개를 테스트 세트로 떼어 놓으면 훈련 세트에는 빙어가 하나도 들어있지 않아서 모델을 훈련시키면 빙어를 올바르게 분류할 수가 없다
=> 훈련 세트와 테스트 세트를 나누려면 도미와 빙어가 골고루 섞이게 만들어야 한다 !
샘플링 편향
훈련 세트와 테스트 세트에 샘플이 골고루 섞여 있지 않으면 샘플링이 한쪽으로 치우쳤다는 의미로 샘플링 편향이라고 부른다 ~~
넘파이
파이썬의 대표적인 배열 라이브러리로, 고차원의 배열을 손쉽게 만들고 조작할 수 있는 간편한 도구를 많이 제공한다.
<생선 데이터를 2차원 넘파이 배열로 변환하기>
import numpy as np
input_arr = np.array(fish_data)
target_arr = np.array(fish_target)
print(input_arr)
print(input_arr.shape) # (49, 2)이 배열에서 랜덤하게 샘플을 선택해 훈련 세트와 테스트 세트로 만들어야 한다. 배열을 섞은 후에 나누는 방식 대신 무작위로 샘플을 고르는 방법 사용!
ㄴ 주의할 점 !
: input_arr와 target_arr에서 같은 위치는 함께 선택되어야 한다는 점!!
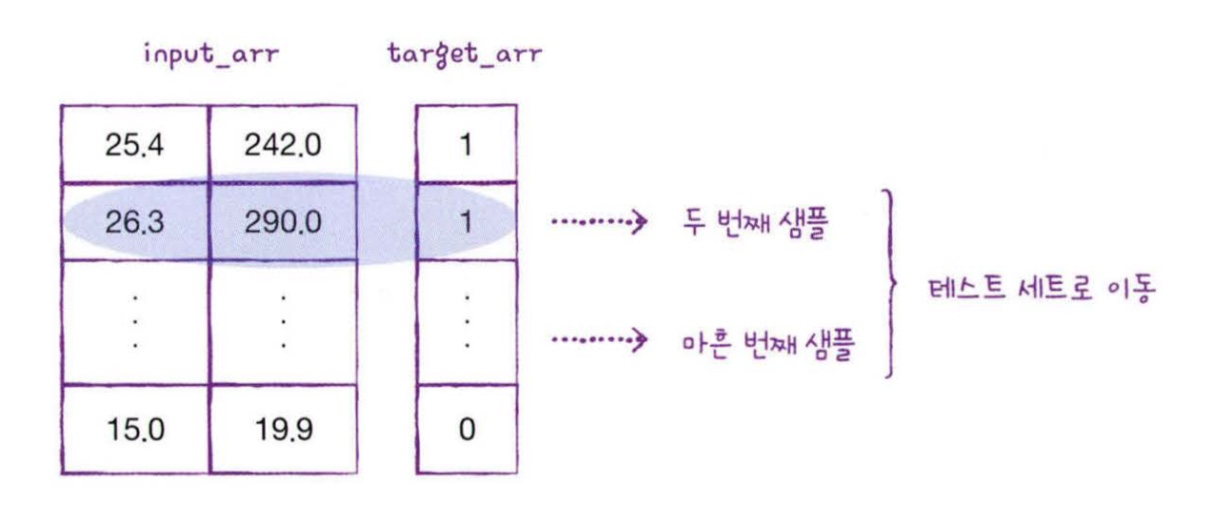
ex) input_arr의 두번째 값은 훈련 세트로, target_arr의 두 번째 값은 테스트 세트로 가면 안됨
이렇게 하면 훈련 세트와 테스트 세트로 나눌 인덱스 값을 잘 기억해야하는데 항상 기억할 수는 없으니까 다른 방버이 필요하다. 바로 인덱스를 섞은 다음 input_arr과 target_arr에서 샘플을 선택하기 !!
np.random.seed(42)
index = np.arange(49)
np.random.shuffle(index)
print(index)output
[13 45 47 44 17 27 26 25 31 19 12 4 34 8 3 6 40 41 46 15 9 16 24 33
30 0 43 32 5 29 11 36 1 21 2 37 35 23 39 10 22 18 48 20 7 42 14 28
38]이제 인덱스를 사용해서 전체 데이터를 훈련 세트와 테스트 세트로 나누어야 한다 ~
이때 배열 인덱싱 기능을 써서 1개의 인덱스가 아닌 여러개의 인덱스로 한 번에 여러 개의 원소를 선택할 수 있음
<훈련 세트 만들기>
train_input= input_arr[index[:35]]
train_target = target_arr[index[:35]]<테스트 세트 만들기>
test_input = input_arr[index[:35]]
test_target = target_arr[index[:35]]훈련 세트와 테스트 세트에 도미와 빙어가 잘 섞였는지 산점도로 그려 보장 ~~
import matplotlib.pyplot as plt
plt.scatter(train_input[:,0], train_input[:,1])
plt.scatter(test_input[:,0], test_input[:,1])
plt.xlabel('length')
plt.ylabel('weight')
plt.show()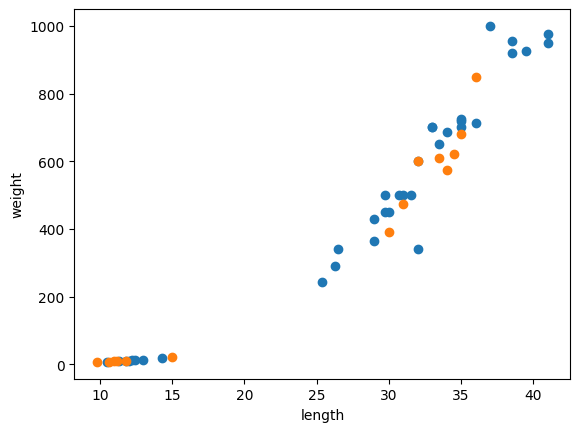
ㄴ 파랑이 훈련 세트, 주황이 테스트 세트
ㄴ 양쪽에 도미와 빙어가 모두 섞여있다
위에서 만든 훈련 세트와 테스트 세트들로 k-최근접 이웃 모델을 훈련 시킬 것임
fit()메서드를 실행할 때 마다 KNeighborsClassifier 클래스의 객체는 이전에 학습한 모든 것을 잃어버리기 때문에 이전 모델을 그대로 두고 싶다면 클래스 객체를 새로 만들어야 함.
<모델 훈련 시키기>
kn = kn.fit(train_input, train_target)<모델 테스트 하기>
kn.score(test_input, test_target) # 1.0<predict() 메서드로 테스트 세트의 예측 결과와 실제 타깃 확인하기>
kn.predict(test_input) # array([0, 0, 1, 0, 1, 1, 1, 0, 1, 1, 0, 1, 1, 0])test_target # array([0, 0, 1, 0, 1, 1, 1, 0, 1, 1, 0, 1, 1, 0])ㄴ 테스트 세트에 대한 예측 결과가 정답과 일치하다 !
데이터 전처리
넘파이의 column_stack()함수는 전달 받은 리스트를 일렬로 세운 다음 차례대로 나란히 연결할 수 있다 ~
ex) np.column_stack(([1,2,3],[4,5,6]))
output
array([[1, 4],
[2, 5],
[3, 6]])이 column_stack()을 활용해서 fish_length와 fish_weight를 합칠 것이당
fish_data = np.column_stack((fish_length, fish_weight))
print(fish_data[:5])output
[[ 25.4 242. ]
[ 26.3 290. ]
[ 26.5 340. ]
[ 29. 363. ]
[ 29. 430. ]]ㄴ 리스트 처럼 한 줄로 길게 출력 되지 않고 행과 열을 맞추어 가지런히 정리된 모습으로 보여진다 ~~
np.concatenate()함수는 첫 번째 차원을 따라 배열을 연결해주는 역할을 한다
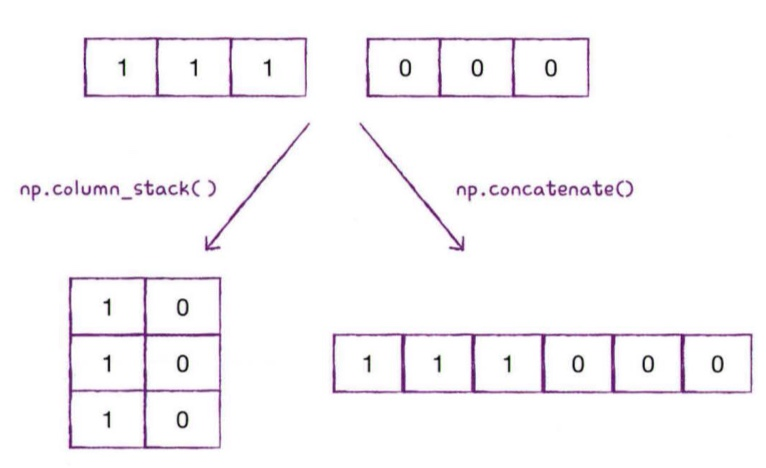
<np.concatenate()함수를 이용해서 타깃 데이터 만들기>
fish_target = np.concatenate((np.ones(35),np.zeros(14)))
print(fish_target)output
[1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1.
1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 1. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0.]사이킷런으로 훈련 세트와 테스트 세트 나누기
사이킷런은 다양한 유틸리티 도구를 제공하는데, 대표적인 도구가 train_test_split()함수이다. 얘는 전달 되는 리스트나 배열을 비율에 맞게 훈련 세트와 테스트 세트로 나누어줌 그리고 나누기 전에 알아서 섞어준다 ~~
<임포트 형식>
from sklearn.model_selection import train_test_split
<훈련 세트와 테스트 세트 나누기>
train_input, test_input, train_target, test_target = train_test_split(fish_data, fish_target, random_state = 42)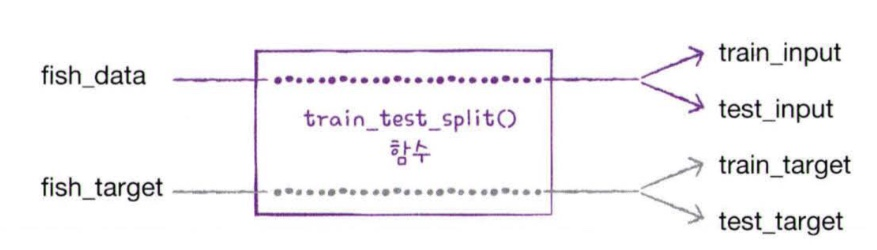
이렇게 fish_data, fish_target 2개의 배열을 전달 했으므로 2개씩 나뉘어서 총 4개의 배열이 반환된다. 이 함수는 기본적으로 25%를 테스트 세트로 떼어냄 !!
print(train_input.shape, test_input.shape) # (36, 2) (13, 2)ㄴ 입력 데이터는 2개의 열이 있는 2차원 배열이고
print(train_target.shape, test_target.shape) # (36,) (13,)ㄴ 타깃 데이터는 1차원 배열이다
잘 섞였는지 테스트 데이터를 출력하면
print(test_target) # [1. 0. 0. 0. 1. 1. 1. 1. 1. 1. 1. 1. 1.]ㄴ 13개의 테스트 세트 중에 10개가 도미(1) 이고 3개가 빙어 (0)이다 -> 빙어의 비율이 조금 모자란다
-> 원래 도미와 빙어의 개수가 35:14 2.5:1이어야 하는데, 이 테스트 세트의 도미와 비엉의 비율은 3.3 : 1이다.. 다르당..
왜 이러는 거지 ?
=> 무작위로 데이터를 나누었을 때, 샘플이 골고루 섞이지 않을 수가 있음 ! 그래서 훈련 세트와 테스트 세트에 샘플의 클래스 비율이 일정하지 않다면 모델이 일부 샘플을 올바르게 학습할 수 없을 것이다~~
어떻게 해결하지 ?
=> stratify매개 변수에 타깃 데이터를 전달하면 클래스 비율에 맞게 데이터를 나눠줌 !
train_input, test_input, train_target, test_target = train_test_split(fish_data, fish_target, stratify = fish_target, random_state = 42)
print(test_target)output
[0. 0. 1. 0. 1. 0. 1. 1. 1. 1. 1. 1. 1.]ㄴ 빙어가 하나 는 것을 볼 수 있다 이제 테스트 세트의 비율이 2.25:1이다 ~~
수상한 도미 한 마리
k-최근접 이웃 훈련 !
<훈련 데이터로 모델을 훈련하고 테스트 데이터로 모델을 평가>
from sklearn.neighbors import KNeighborsClassifier
kn = KNeighborsClassifier()
kn.fit(train_input, train_target)
kn.score(test_input, test_target)output
1.0ㄴ 테스트 세트의 도미와 빙어를 모두 올바르게 분류했음
<김팀장이 준 도미 데이터를 넣고 결과 확인하기>
print(kn.predict[[25, 150]])output

ㄴ 난 이렇게 오류가 뜨는데.. 왜지?
ㄴ 원래는 [0.]이 나와야 한다
<샘플을 다른 데이터와 함께 산점도로 그려보기>
import matplotlib.pyplot as plt
plt.scatter(train_input[:,0], train_input[:,1])
plt.scatter(25, 150, marker ="^") # 새로운 샘플은 marker 매개변수를 ^로 지정하여 삼각형으로 표현
plt.xlabel('length')
plt.ylabel('weight')
plt.show()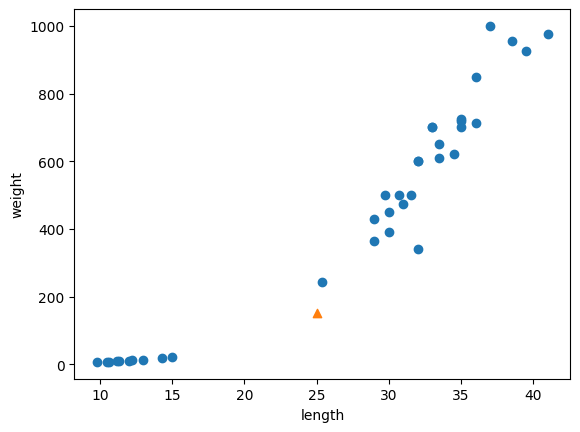
ㄴ 이 샘플은 분명히 오른쪽 위로 뻗어있는 도미 데이터에 더 가까운데, 왜 이 모델은 왼쪽 아래에 낮게 깔린 빙어 데이터에 가깝다고 판단한 것일까?
k-최근접 이웃은 주변의 샘플 중에서 다수인 클래스를 예측으로 사용함.
KNeighborsClassifier 클래스는 주어진 샘플에서 가장 가까운 이웃을 찾아 주는 nkeighbors()메서드를 제공한다. 이 메서드는 이웃까지의 거리와 이웃 샘플의 인덱스를 반환함.
그럼 이 샘플의 주변 샘플을 알아봐보자 !!
distances, indexes = kn.kneighbors([[25,150]])plt.scatter(train_input[:,0], train_input[:,1])
plt.scatter(25, 150, marker ="^")
plt.scatter(train_input[indexes,0], train_input[indexes,1],marker = "D") # marker = "D"로 지정하면 산점도를 마름모로 그림
plt.xlabel('length')
plt.ylabel('weight')
plt.show()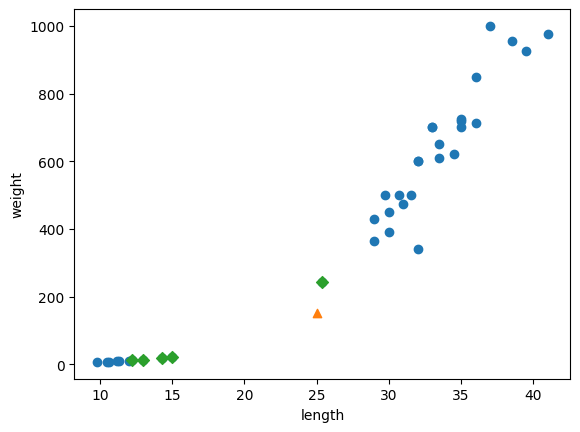
삼각형 샘플에 가장 가까운 5개의 샘플이 초록 다이아몬드로 표시됨
-> 가장 가까운 이웃에 도미가 하나밖에 포함되지 않았음.. 나머지 4개는 다 빙어 !!
<직접 데이터 확인하기>
print(train_input[indexes])output
[[[ 25.4 242. ]
[ 15. 19.9]
[ 14.3 19.7]
[ 13. 12.2]
[ 12.2 12.2]]]<타깃 데이터로 더 명확하게 확인하기>
print(train_target[indexes])output
[[1. 0. 0. 0. 0.]]길이가 25cm, 무게가 150g인 생선에 가장 가까운 이웃에는 빙어가 압도적으로 많아서 샘플의 클래스를 빙어로 예측하는 것은 무리가 아님. 근데 왜 가장 가까운 이웃을 도미가 아니라 빙어라고 생각한 걸까?
<kneighbors() 메서드에서 반환한 distances 배열(=이웃 샘플까지의 거리가 담김) 출력해보기>
print(distances)output
[[ 92.00086956 130.48375378 130.73859415 138.32150953 138.39320793]]ㄴ 거리가 92와 130이라고 했는데 그래프에 나타난 거리 비율은 92의 거리보다 족히 몇배는 되어 보인다 !!
-> x축은 범위가 좁고 (10~40), y축은 범위가 넓음 (0~1000) = y축으로 조금만 멀어져도 거리가 아주 큰 값으로 계산 될 것!! 그래서 도미 샘플이 이웃으로 선택받지 못한 거다 ~~
<x축 범위를 동일하게 맞추어서 시각화하기>
plt.scatter(train_input[:,0], train_input[:,1])
plt.scatter(25, 150, marker = "^")
plt.scatter(train_input[indexes, 0], train_input[indexes, 1], marker = 'D')
plt.xlim((0,1000)) # x축 범위 저장하는 함수
plt.xlabel('length')
plt.ylabel('weight')
plt.show()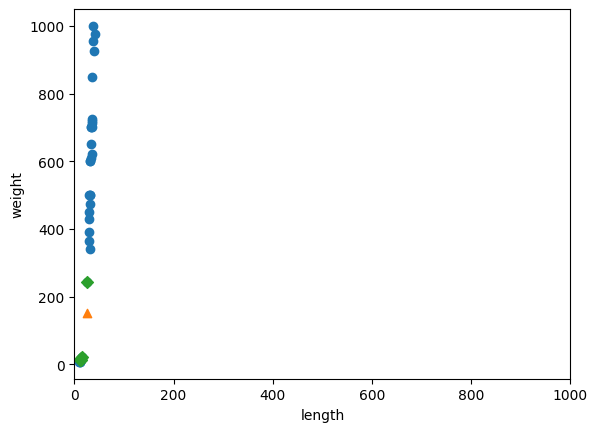
ㄴ 길이와 무게의 값이 놓인 범윅 매우 다른데, 이를 두 특성의 스케일이 다르다고도 한다 ~
데이터를 표현하는 기준이 다르면 알고리즘이 올바르게 예측할 수 없고 특히 알고리즘이 거리 기반일 때 더 그런다.
= 알고리즘들은 샘플간의 거리에 영향을 많이 받으므로 특성값을 일정한 기준으로 맞춰주어야 하고 이런 작업을 데이터 전처리라고 부른다 !!
가장 널리 사용하는 전처리 방법 중 하나는 표준점수가 있다. 이는 특성값이 평균에서 표준편차의 몇 배 만큼 떨어져 있는지를 나타냄 (= 실제 특성값의 크기와 상관 없이 동일한 조건으로 비교가 가능하당)
ㄴ 그럼 계산하는 방법은 ?
: 평균을 빼고 표준편차를 나누어 주면 된다 ~~
mean = np.mean(train_input, axis = 0) # 평균 계산
std = np.std(train_input, axis = 0) # 표준 편차 계산
print(mean, std)output
[ 27.29722222 454.09722222] [ 9.98244253 323.29893931]<원본 데이터에서 표준 점수 반환하기>
train_scaled = (train_input - mean) / std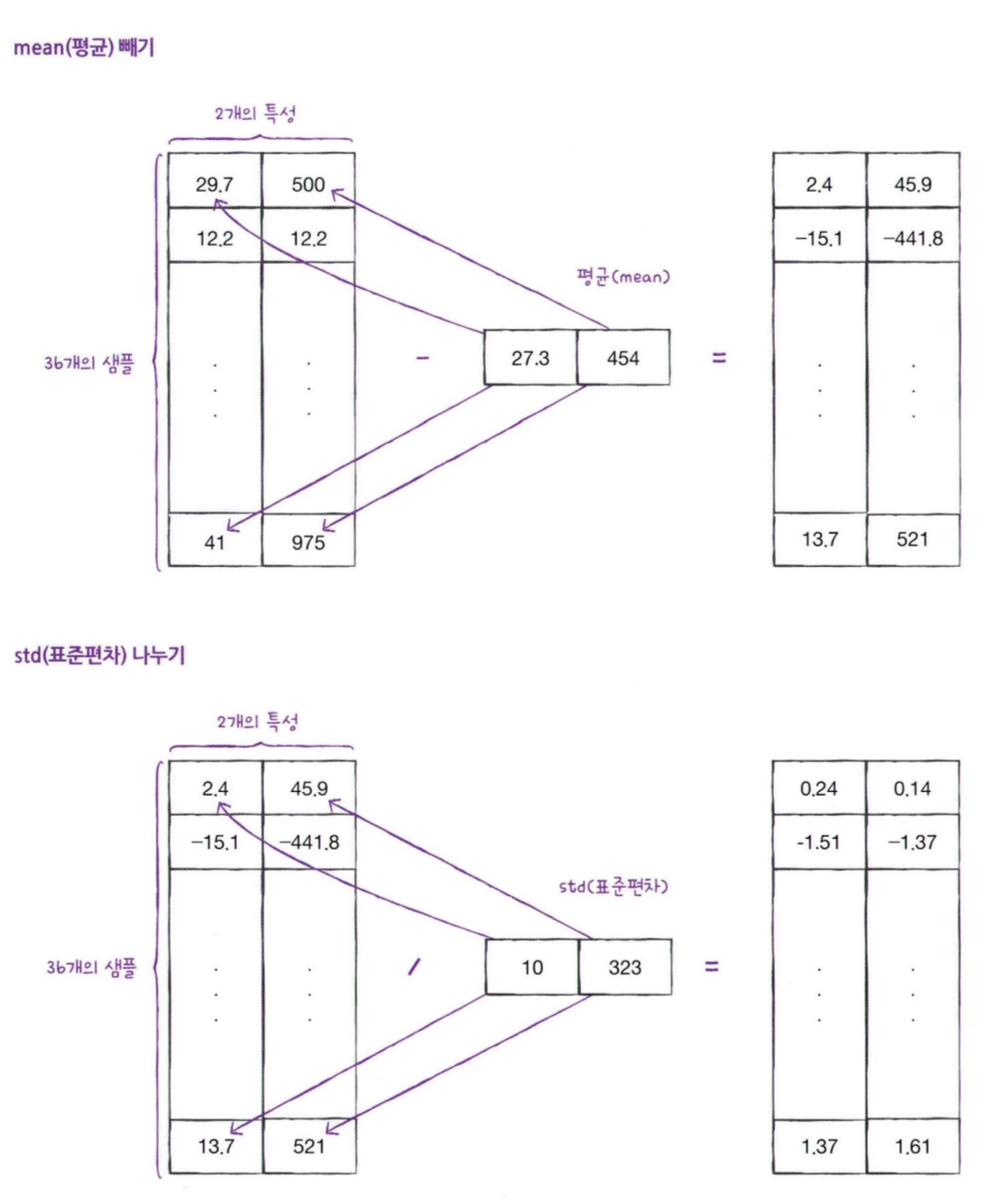
전처리 데이터로 모델 훈련하기
<샘플을 다시 산점도로 그리기>
plt.scatter(train_scaled[:,0], train_scaled[:,1])
plt.scatter(25, 150, marker ="^")
plt.xlabel('length')
plt.ylabel('weight')
plt.show()output
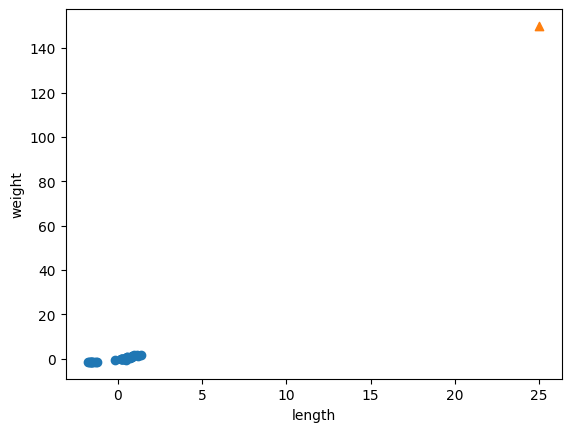
오른쪽 맨 꼭대기에 샘플 하나만 덩그러니 있고.. 예상과는 다르다 !!
왜냐? = 훈련 세트를 표준 점수로 반환했기 때문에 값의 범위가 크게 달라짐. 샘플 [25,150] 을 동일한 비율로 변환하지 않으면 이런 현상이 발생한다
<동일한 기준으로 샘플을 변환하고 다시 산점도 그리기>
new = ([25,150] - mean)/std
plt.scatter(train_scaled[:,0], train_scaled[:,1])
plt.scatter(new[0], new[1], marker ="^")
plt.xlabel('length')
plt.ylabel('weight')
plt.show()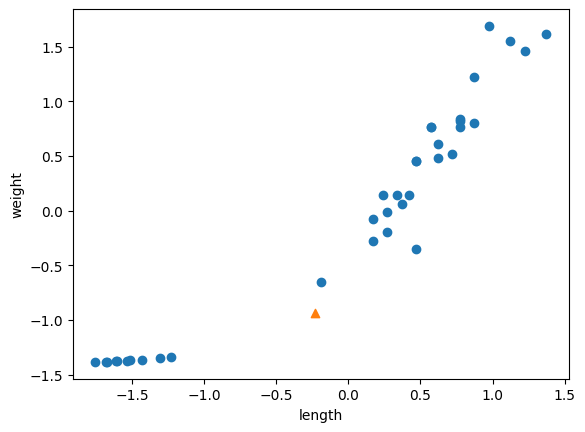
ㄴ x축과 y축의 범위가 -1.5 ~ 1.5 로 바뀌었음 ! 훈련 데이터의 두 특성이 비슷한 범위를 차지 하고 있드아..
<k-최근접 이웃 모델 훈련하기>
kn.fit(train_scaled, train_target)훈련하고 테스트 세트로 평가할 때 주의할 점!
테스트 세트도 훈련 세트의 평균과 표준편차로 변환해야 한다. 그렇지 않으면 데이터의 스케일이 같아지지 않아서 훈련한 모델이 쓸모가 없게 됨 !
<테스트 세트의 스케일 변환하기>
test_scaled = (test_input - mean)/std<모델 평가하기>
kn.score(test_scaled, test_target)output
1.0ㄴ 모든 테스트 세트의 샘플을 완벽하게 분류함 !!
<훈련 세트의 평균과 표준편차로 변환한 샘플 사용해서 모델의 예측 출력>
print(kn.predict[new])
난 왜 predict만 하면 오류가 나지..
아무튼 원래 출력값은
[1.]이 나와야 한다. 도미(1)로 예측한 것 !
<k-최근접 이웃 구한 후 산점도로 그리기>
distances, indexes = kn.kneighbors([new])
plt.scatter(train_scaled[:,0], train_scaled[:,1])
plt.scatter(new[0], new[1], marker ="^")
plt.scatter(train_scaled[indexes, 0], train_scaled[indexes, 1], marker = "D")
plt.xlabel('length')
plt.ylabel('weight')
plt.show()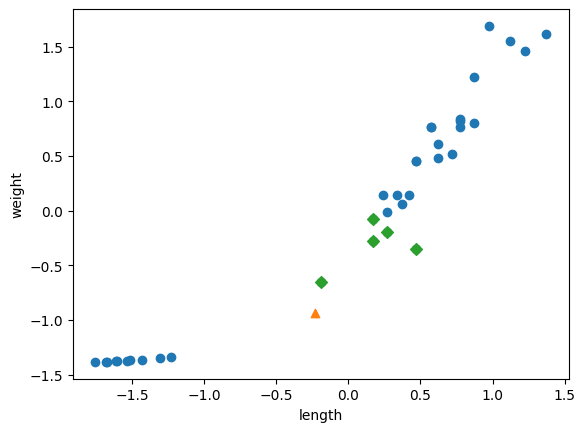
ㄴ 김팀장 샘플(삼각형)에서 가장 가까운 샘플은 모두 도미다 !!
