(2021)Unsupervised Anomaly Segmentation using Image-Semantic Cycle Translation
Paper Review
0. Abstract
UAS(Unsupervised Anomaly Segmentation)의 목적은 학습 동안 보지 못한 anomalies를 찾는 것이다. 현존하는 방법들은 주로 Information Bottleneck 기반이다. 학습을 통해 low-dimension에서 healthy data로 복원함으로 normal의 distribution을 찾고 이를 통해 outlier인 lesion을 찾는 방법이다. 그러나 dimension 감소는 localization information을 감소 시킨다는 단점이 있다.
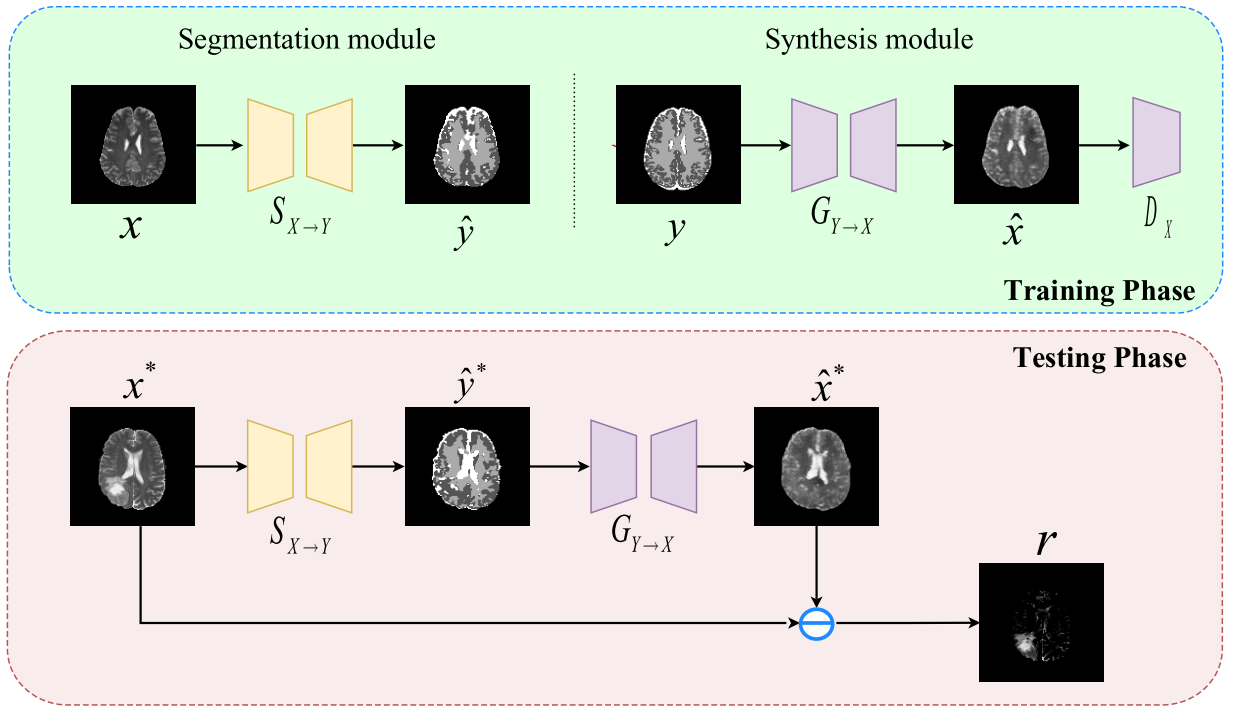
본 논문에서는 healthy anatomy의 semantic space를 healthy-data의 distribution으로 대체함으로 이러한 문제를 해결하였다. 게다가 segmentation, synthesis, clcle translation이라는 세 단계의 과정을 도입하였다. (image→semantic→image)
실험 결과, BraTS, ISLES dataset에서 이전의 methods와 비교했을 때 좋은 결과를 얻었다.
1. Introduction
Labeling은 수고가 많이 들고 medical images는 얻기가 힘들다. 게다가 model은 training data에 bias가 있어서 unseen data의 lesion을 잘 찾지 못한다. 이를 해결하기 위해 UAS(Unsupervised Anomaly Segmentation)가 제안 되었다. Supervised learning과 달리 annotate되지 않은 health images만 가지고 학습을 한다. 대부분의 방식은 Information Bottleneck에 기반해 healthy data의 normative distribution을 학습한다. 하지만 local information이 줄어든다는 단점이 있었으며 이를 해결하기 위해 많은 시도들이 있었지만 'compress and recover' 방식을 벗어나지는 못했다.
본 연구의 목적은 normative distribution을 semantic domain으로 대체하는 것이다. 그리고 semantic domain을 다시 image domain으로 복원함으로 (image→semantic→image) 방식을 따른다.
본 논문에서는 BraTS, ISLES 두 가지의 brain lesion dataset에 제안 된 접근 법을 적용해 보았다. 실험 결과 기존의 방식들 보다 정확하게 lesion을 찾는 것을 확인할 수 있다.
2. Method
1) Overall Concept

를 healthy image, 를 anatomical tissues의 pixel wise labels이라고 가정한다. Anatomical tissue는 GM(Gray Matter), WM(White Matter), CSF(Cerebrospinal Fluid)로 구성된다.
Segmentor 는 standard cross-entropy loss로 health data를 사용해 학습하였다.
Anatomical tissue의 pixel-wise label은 SPM과 같은 tool을 사용하여 많은 수고가 들지 않고 얻을 수 있었다.
Synthesis는 conditional GAN을 사용하여 semantic space to image domain의 pixel-to-pixel translation을 수행하였다. Generator 와 discriminator 는 adversarial loss로 학습이 된다.
게다가 일관된 image synthesis를 유지하기 위해 L1 loss가 추가되었다.
위 objective를 합쳐서 mini-max two-player game 방식으로 학습을 한다.
2) Anomaly Segmentation
Lesion은 input image와 recovered image를 통해 얻은 을 통해 표시된다.
3. Experiments and Results
1) Dataset
BRATS(Multimodal Brain Tumor Image Segmentation) 2019와 ISLES(Ischemic Stroke Lesion Segmentation) 2015 dataset에 대해 실험을 진행하였다. Anatomical tissue의 semantic label은 MATLAB의 SPM tool을 사용하였다.
2) Implementation Details
Input image는 128x128로 down-sampled 되었고 [0, 1]로 normalized 되었다.
Segmentation module은 U-Net을 사용하였고 synthesis module의 generator와 discriminator는 "Perceptual Losses for Real-Time Style Transfer and Super-Resolution"의 model을 기반으로 하였다.
Segmentor는 30 epoch, Synthesis는 15 epoch 학습하였으며 learnig rate 2e-4, (0.5, 0.999)의 Adam optimizer를 사용하였다.
는 10에서 가장 좋은 quality를 얻었으며 batch size는 128이다.
3) Ablation Study
.png)
Discrete, continuous의 두 가지의 segmentation maps를 label로 두고 비교 실험을 하였다. Metric으로는 AUPRC(Area Under the Precision-Recall Curve), Dice-score을 사용하였다.
위 그림에서 2, 6, 8 column이 discrete-semantic-intermediated baseline이고 3, 4, 5, 7, 9 column은 continuous-semantic-intermediated improved model의 결과이다.
.png)
위 표를 보면 continuous 방식이 AUPRC, Dice score가 더 높다. 이는 continuous 방식이 discrete 방식보다 cycle translation framework에서 우수함을 보여준다.
4) Comparisons with Existing Methods
.png)
UAS model인 AE, VAE, GMVAE, fAnoGAN과 함께 BraTS, ISLES dataset에 비교 실험하였다.
위 그림에서 A ~ C block은 BraTS의 결과이며 D ~ F block은 ISLES의 결과이다.
각 block에서 1 row는 input image와 AE, VAE, GMVAE, fAoGAN, 본 model에 의해 생성된 reconstruction이다. 2 row는 lesion의 ground truth와 AE, VAE, GMVAE, fAoGAN, 본 model의 residuals를 나타낸다.
.png)
다른 UAS model과 비교 했을 때 본 model이 AUPRC, Dice score가 가장 높음을 확인할 수 있다.
.png)
GM(Gray Matter), WM(White Matter), CSF(Cerebrospinal Fluid), LES(lesion)의 posterior probabilistic distribution을 학습된 segmentor의 output에 대해 비교하였다.
LES를 보면 다른 normal tissue와는 구별되는 prediction distribution을 갖고 있음을 보여준다. 이를 통해 semantic space의 효과를 입증한다.
반면에 GM과 LES의 distribution이 비슷한 것을 확인할 수 있는데 synthesis가 LES를 GM으로 착각할 수 있다. 또한 T2 modality에서는 CSF와 lesion이 함께 highlighted되어 LES를 CSF로 착각할 여지가 있다.
4. Conclusion
본 논문에서는 healthy-data distribution을 학습하기 위해 semantic space를 적용하는 방식을 제안하였다. 그리고 segmentation과 synthesis 모델을 사용하여 'image→semantic→image' 방식의 cycle translation framework를 제안하였다.
실험 결과, BraTS, lSLES dataset에서 본 모델이 이전 모델들에 비해 좋은 결과를 보여줌을 확인할 수 있다.