[논문 리뷰] Suggestive Annotation: A Deep Active Learning Framework for Biomedical Image Segmentation
논문 리뷰
논문을 읽게 된 배경
이 논문은 medical image segmentation 에서 segmentation 시, label값이 되는 annotation 을 자동으로 해주는 기법에 관한 연구가 나왔다는 소식을 듣고, 읽어보게 되었다.
논문명
Suggestive Annotation: A Deep Active Learning Framework for Biomedical Image Segmentation
저자
Lin Yang, Yizhe Zhang, Jianxu Chen, Siyuan Zhang, Danny Z. Chen
게재 학술지
Comments: Accepted at MICCAI 2017
Subjects: Computer Vision and Pattern Recognition (cs.CV)
Abstract
biomedical image의 경우, modality, image settings, object, noise 등 다양한 variation을 갖기 때문에 새로운 application에 deep learning을 적용할 때, 새로운 training data를 필요로 한다.
이 작업은 오직, biomedical 전문가만이 효율적으로 annotation 을 수행할 수 있기 때문에, 많은 노력과 비용을 필요로 한다.
본 논문에서는 가장 효과적인 annotation area를 제안하는 fully convolutional network(FCN)과 active learning 을 결합한 deep active learning framework를 제안한다.
우리는 가장 대표적이고 불분명한 annotation 을 결정하기 위해 FCN으로부터 제공받은 불확실하고 유사한 정보를 사용하고, 최대 대이터 셋에 대한 가장 일반적인 version을 형성한다.
2015 MICCAI Gland Challenge dataset과 lymph node ultrasound image segmentation dataset을 사용했을 때, 우리가 제안한 모델이 segmentation performance에서 sota(state-of-the-art)성능을 내었다. (training data의 50%만 사용함.)
Introduction
biomedical image segmentation은 annotation에 대해 많은 비용과 노력이 들기 때문에 충분한 training data를 얻는 것이 어렵다는 문제가 있다.
natural scene image에서의 applicatation과 비교했을 때, 두가지 이유로 biomedical image 에서 training data를 얻는 것이 훨씬 어렵다.
(1) biomedical 전문가에 의해서만 annotation data 를 얻을 수 있다.
(2) biomedical image 에는 natural scene image보다 훨신 많은 object instance가 포함되어 있다.
수동 annotation 작업으로 인한 부담을 완화하기 위해, weakly supervise segmentation algorithm 들이 제안되었다.
그러나 제안된 알고리즘은 높은 성능을 내기 위해 어떤 data sample들이 선택되어야 하는지에 대해서는 다루지 않았다.
이러한 필요에 의해, active learning은 learning model이 training data를 선택할 수 있도록 한다.
active learning을 사용했을 때, natural scene image segmentation에서 더 적은 training data을 사용하고도 sota 성능을 낼 수 있음이 보고되었다.
하지만, 이 모델은 pretrained region proposal model과 pretrained image desciptor network 을 기반으로 한다. 이러한 사전 훈련된 모델은 biomedical application의 넓은 variation 때문에, biomedical image setting에서는 쉽게 얻어질 수 없다.
우리는 FCN을 통해, region proposal없이 domain specific image descriptor와 직관적인 segmentation을 도출하였다.
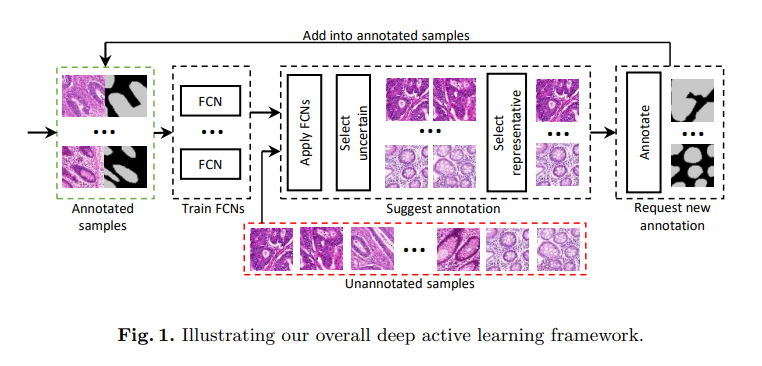
Fig. 1은 본 논문의 main idea와 deep active learning framework의 단계를 보여준다.
아주 적은 양의 training data에서 시작하여 우리는 여러개의 FCN set을 반복적으로 학습한다.
각 stage의 끝단계마다 FCN으로부터 유용한 정보(uncertainty estimation, similarity estimation)를 추출한다. 그리고 그 다음 annotation 할 이미지를 결정한다.
새로운 annotation data를 얻은 다음, 다음 단계는 획득가능한 annotated image를 사용하여 시작된다. 위 단계는 간단해보이지만, 우리는 FCN과 deep active learning framework를 결합하기 위하여 해결해야 할 몇가지 문제(challenges)가 있다.
Challenges from the perspective of FCNs.
(1) 두 annotation stage간의 시간 간격(time interval)이 허용가능해지도록 FCN은 빠르게 훈련되어야 한다.
(2) 일반화 성능이 좋아야 한다. (good generality)
우리는 (1)을 위해 batch normalization과 residual network를 사용한다.
따라서 우리는 비슷한 수의 feature channels수를 유지하면서 parameter수를 줄이기 위해 (좋은 일반화 성능을 내기 위해) bottle neck 디자인을 사용한다.
Method
본 논문에서 제안한 방법은 세가지 메인 요소로 이루어진다.
첫번째로, 본 논문의 실험에서 사용된 두 가지의 데이터셋에서 sota성능을 달성한 새로운 FCN이고,
두번째로, FCN sets의 uncertainty estimation과 similarity estimation이고,
세번째로, 가장 효과적인 training data을 결정하는 annotation 제안 알고리즘이다.
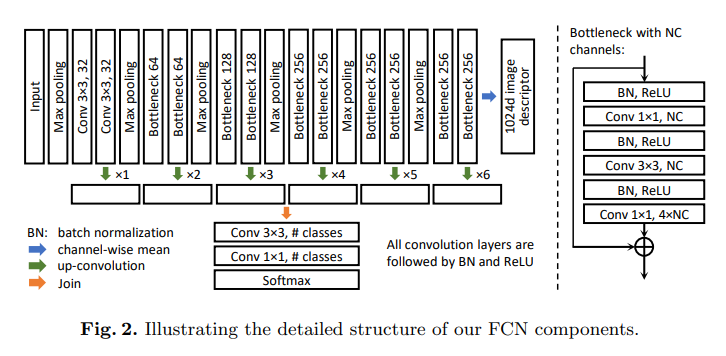
1) A new fully convolutional network
본 논문에서는 batch normalization과 residual network를 기반으로 새로운 FCN을 설계하여 학습 속도를 절감하고, 일반화 성능을 향상시켰다.
FCN의 encoding 파트는 DCAN의 구조를 따른다.
residual network와 batch normalization에서 볼 수 있듯이 이러한 변형을 거친 모델은 원래 모델과 비교하였을 때 더 적은 훈련단계로도 같은 정확도를 달성할 수 있다.
이 점은 FCN이 어느 정도 성능을 달성하는 데에 몇시간이나 소요되기 때문에, FCN과 active learning을 결합할 때에 아주 중요한 요소이다.
그러므로, 본 논문에서는 원래의 convolution layer를 batch normalization을 탑재한 residual modules로 교체하였다.
active lerarning을 시작할 때, 적은 양의 training data만 사용가능하기 때문에,
너무 많은 free parameter를 갖는 것은 모델 학습을 어렵게 만든다.
그러므로 본 논문에서는, 각각의 residual module의 끝에서 비슷한 수의 feature channel수를 유지하면서 parameter 수를 줄이기 위해 bottleneck design을 사용하였다.
smooth result를 위해 feature map의 크기를 점진적으로 확대되게 하였다.
마지막으로, 3x3 convolution layer와 1x1 convolutional layer가 각각의 다른 범위의 feature map을 결합하는데에 사용되었다.
-> 새로운 FCN은 매우 적은 수의 training data로도 sota 성능을 달성하였다.
2) Uncertainty estimation and similarity estimation
가장 valuable annotation area를 찾기 위해, uncertainty sampling을 사용하였다.
그러나 딥러닝 모델은 비슷한 type의 instance에 대해 uncertain한 경향이 있기 때문에, uncertainty sampling을 사용하는 것은 annotation area가 중복되는 결과를 초래할 수 있다.
이 문제를 피하기 위해, 본 논문에서는 uncertainty 하고 가장 representive한 sample을 찾는 것을 목표로 한다.
이를 달성하기 위하여, 결과의 uncertainty를 추정하고, 이미지 간의 유사성을 측정할 필요가 있다.
이 section에서는, FCNs에서 그러한 sample을 추출하는 방법을 보여준다.
Bootstrapping은 학습 모델의 uncertainty를 평가하는 데 가장 표준화된 방법이다.
Bootstrapping의 기본 아이디어는 복원추출하여 선택된 training data의 subset을 사용하기 위해 각 model 을 제한하면서 모델의 한 set을 학습시키고, 이 모델들 사이의 variance를 계산하는 것이다.
본 논문에서는 FCNs의 uncertainty를 측정하기 위해 이 절차를 따른다.
각각의 FCN에 내재된 variance는 variance를 overextimation하게 될 수 있다.
실제로, FCN은 uncertainty에 대해 good estimation을 제공한다.
Fig3. (d)에서 볼 수 있듯이, 각각의 pixel에 대해 추정된 uncertainty는 testing errors에 대해 강한 correalation을 갖는다.

불확실한 training sample를 선택하는 것은 FCNs이 잠재적인 error를 바로잡는 것을 도와준다.
마지막으로, 각 training sample에 대한 전반적인 uncertainty는 그에 해당하는 픽셀의 평균적인 uncertainty로서 계산된다.
CNN based image desciptor는 natural scene image에서는 좋은 결과를 달성하였다.
FCN의 encoding 파트는 natural 한 CNN이다.
input image , encoding 파트의 마지막 convolutional layer의 output은 의 high level feature 이다.
그 다음은, 이미지의 shifting, rotation의 variance를 제거하는데, domain-specific image descriptor로 압축된 feature인 을 생성하기위해, 의 channel-wise mean을 계산해준다.
(1) 여기에는 다른 개별적인 image descriptor network를 학습시킬 필요가 없다.
(2) FCNs은 object에 대한 segmentaton을 계산하려하기 때문에 는 풍부하고 정확한 shape information을 포함한다.
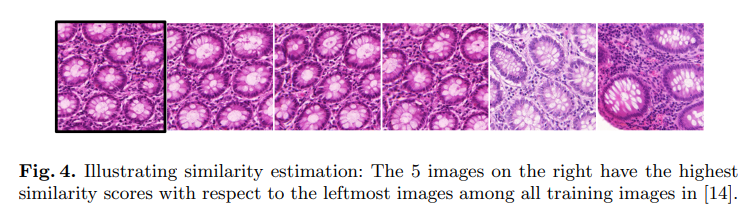
마지막으로, 본 논문에서는 두 이미지 와 간의 유사성을 로 정의한다.
Fig. 4는 유사성 측정 예시를 보여준다.
3) Annotation suggestion
annotation data의 효과를 극대화하기 위해, (1) uncertainty, (2) representativeness를 가져야 한다.
(1) uncertainty : annotated area는 network가 segment하기에 어려울 필요가 있다.
(2) representativeness : 가능한 많은 annotate되지 않은 image를 위하여 유용한 characteristic이나 feature를 bear(?)할 필요가 있다.
이 section에서는, similarity와 uncertainty의 estimation에 기반하여, 위 두가지 특성을 가장 잘 만족하는 annotation area를 어떻게 제안하는 지를 보여준다.
각각의 annotation suggestion 단계에서는, 와 을 선택하는 것을 목표로 한다.
- : 모든 unannotated image
- : 중 k개 select한 subset(highly uncertain and representative)
Experiments and Results
Conclusions
본 논문에서는 FCNs과 active learning을 결합하여 biomedical image segmentation에 대한 새로운 deep active learning framework를 제안했다.
우리의 새로운 모델은 segmentation perfomance에서 sota성능을 달성하고, 효율적이고 자동적으로 annotation area를 제안하는 것에 기여하였다.
key words
- active learning
[ref]
biomedical segmentation paper
- Ronneberger, O., Fischer, P., Brox, T.: U-net: Convolutional networks for biomedical image segmentation. In: MICCAI. pp. 234–241 (2015)
- Xu, Y., Li, Y., Liu, M., Wang, Y., Lai, M., Chang, E.I.: Gland instance segmentation by deep multichannel side supervision. In: MICCAI. pp. 496–504 (2016)
- Sirinukunwattana, K., Pluim, J.P., Chen, H., Qi, X., Heng, P.A., Guo, Y.B., et al.:
Gland segmentation in colon histology images: The GlaS challenge contest. Medical
Image Analysis 35, 489–502 (2017) - Xu, Y., Li, Y., Wang, Y., Liu, M., Fan, Y., Lai, M., et al.: Gland instance segmentation using deep multichannel neural networks. arXiv preprint arXiv:1611.06661
(2016)
supervised learning
- Hong, S., Noh, H., Han, B.: Decoupled deep neural network for semi-supervised
semantic segmentation. In: NIPS. pp. 1495–1503 (2015)
active learning
- Settles, B.: Active learning literature survey. University of Wisconsin, Madison 52(55-66), 11 (2010)
- Jain, S.D., Grauman, K.: Active image segmentation propagation. In: CVPR. pp.
2864–2873 (2016)
segmentation network
- Chen, H., Qi, X., Yu, L., Heng, P.A.: Dcan: Deep contour-aware networks for
accurate gland segmentation. In: CVPR. pp. 2487–2496 (2016)