[논문리뷰 | NLP] UmlsBERT: Clinical Domain Knowledge Augmentation of Contextual Embeddings Using the Unified Medical Language System Metathesaurus (2021) Summary
1
[논문리뷰]
목록 보기
29/42
Title
- UmlsBERT: Clinical Domain Knowledge Augmentation of Contextual Embeddings Using the Unified Medical Language System Metathesaurus

0. 논문 읽기 전에 알면 좋을 것
ㄱ. Metathesaurus / UMLS
UMLS는 미국 국립 의학 도서관에서 개발한 통합 의학 언어 시스템임.
메타세사우루스는 UMLS(Unified Medical Language System)의 핵심 구성 요성 중 하나임.
1. 다양한 의학 용어의 집합
- 메타세사우루스는 수많은 의학적 용어, 이름, 코드, 개념, 기호 등을 포함하고 있음.
2. 다양한 소스
- 메타세사우루스는 100개 이상의 다양한 의학 언어 리소스 및 표준에서 용어와 개념을 집계함.
- 이를 통해 사용자는 다양한 소스에서 동일한 의미를 가진 용어나 개념을 식별하고 연결할 수 있음.
3. 관계 구조
- 개념 간의 관계와 계층 구조를 포함하여 복잡한 의학 지식을 표현함.
- 예를 들어, 한 질병이 다른 질병의 하위 분류인지, 두 약물이 유사한 효과를 가지는지 등의 관계를 알 수 있음.
4. 언어 독립적
- 메타세사우루스는 여러 언어의 의학 용어를 포함하므로, 다양한 언어로 된 의학 문서나 데이터를 다룰 때 유용함.
5. 의학적 컨텍스트의 통합
- 메타세사우루스는 의학적 컨텍스트에서의 용어와 개념의 사용 방식을 통합하여 제공
- 이를 통해 의료 전문가나 연구자는 용어나 개념의 의미를 정확하게 파악할 수 있음.
ㄴ. CUI (Concept Unique Identifier)
- UMLS(Unified Medical Language System) 내에서 특정 의학적 개념을 고유하게 식별하기 위한 고유한 코드임.
- UMLS는 다양한 의학 용어와 분류 체계를 통합하는 큰 데이터베이스이며, CUI는 그 안에서 다양한 용어나 표현이 동일한 의미를 가진 의학적 개념을 나타내는 경우 그들을 하나의 고유 식별자로 연결하는 역할을 함.
- 'lungs', 'lung', 'pulmonary'와 같은 단어나 표현은 호흡기와 관련된 같은 의학적 개념을 나타낼 수 있으므로, 이러한 단어들은 동일한 CUI로 연결될 수 있음.
Abstract
UmlsBERT는 의료 분야의 자연어 처리에서 높은 성능을 보이는BioBERT및Bio_ClinicalBERT와 같은 문맥 기반 단어 임베딩 모델들을 발전시킨 모델임.
- 기존 모델들은 특정 도메인의 말뭉치를 중심으로 학습되지만, 전문가의 구조화된 지식을 반영하지 않음.
- UmlsBERT는 통합 의학 언어 시스템 (UMLS) 메타세사우루스를 활용하여 도메인 지식을 통합하는 새로운 지식 증강 전략을 사용함.
- 이를 통해 UMLS 내에서 동일한 '개념'을 가진 단어들을 연결하고, UMLS의 의미론적 유형 지식을 활용하여 의학적으로 의미 있는 입력 임베딩을 생성
- 이 두 가지 전략을 적용함으로써 UmlsBERT는 단어 임베딩에 임상 도메인 지식을 인코딩할 수 있으며, 이름 인식 (NER) 및 임상 자연어 추론 작업에서 기존 도메인 특화 모델을 능가gka.
- 요약하면, UmlsBERT는 UMLS 메타세사우루스를 활용하여 의료 도메인 지식을 임베딩하는 새로운 방법을 제시하며, 기존의 도메인 특화 모델보다 더 나은 성능을 보여줌.
1. Introduction
- 본 연구는 최근 의료 분야에서 수집된 데이터의 양이 크게 증가하고 있음을 지적하고, 이러한 데이터 중 텍스트 형태로 되어 있기에 NLP 모델이 필요하다고 강조함.
- 문맥 기반 단어 임베딩 모델(ELMo, BERT)들은 일반 도메인에서 테스트되었지만, 생물의학 말뭉치에서 사전 학습함으로써 생물의학 NLP 작업에서도 좋은 성능을 보여줌.
- 하지만 그러나 현재의 생물의학 응용 프로그램은 임베딩 사전 학습 과정에 지식 베이스에서 구조화된 전문가 도메인 지식을 포함하지 않음.
- 이 논문에서는 UMLS Metathesaurus를 사용하여 문맥 임베딩에 임상 도메인 지식을 통합하는 새로운 architecture를 소개
- BERT 기반 모델의 사전 학습 단계에서 임상 메타세사우루스 (UMLS Metathesaurus)에서 도메인 (임상) 지식을 사용하여 '의미론적으로 풍부한' 문맥 표현을 구축하는 것을 제안
- UMLS의 CUI 속성을 사용하여 임상 단어 간의 연결을 포함하는 UmlsBERT의 Masked Language Modelling 작업에 대한 새로운 다중 라벨 손실 함수를 제안
- 동일한 의미론적 유형의 단어 간의 관계를 고려하도록 모델에 강제하는 의미 유형 임베딩을 도입
- UmlsBERT가 다양한 임상 명명된 엔터티 인식 (NER) 작업 및 하나의 임상 자연어 추론 작업에서 인기 있는 두 가지 임상 기반 BERT 모델 (BioBERT 및 Bio_ClinicalBERT) 및 일반 도메인 BERT 모델을 능가함을 보여줌.
2. Related Work
(1) ELMo (2018)
- 양방향 언어 모델을 사용하여 단어의 임베딩을 주변 문맥에 따라 변경하는 방식 도입
(2) BERT
- 양방향 Transformer를 사용하여 문맥에 따른 표현 생성
(3) 지식 기반 향상
3.1. Sense-BERT
- WordNet에서 어휘 의미론을 포함하여 모델의 사전 학습 목표를 개선
3.2. GlossBERT
- BERT 모델의 문장 쌍 분류 작업에서 문맥-용어 쌍을 사용하여 단어 의미의 모호성을 개선
(4) 생물의학 분야에서의 개선
4.1. BioBERT
- 생물의학 말뭉치를 포함시키면 모델의 작업 성능이 향상됨.
4.2. Bio_ClinicalBERT
- MIMIC-III v1.4. database의 임상 텍스트에서 BioBERT를 추가로 사전 학습함.
3. Data
MIMIC-III dataset
- UmlsBERT 모델을 사전 학습하기 위해 사용된 주요 데이터셋임.
NOTEEVENTS Lable
- UmlsBERT의 학습에 사용된 주요 테이블로, 2,083,180개의 임상 노트와 검사 보고서를 포함하고 있음.
평가 dataset
- UmlsBERT 모델의 새로운 기능을 평가하기 위해 사용되었으며, 주요 데이터셋으로는 English MedNLI 자연어 추론 작업 및 네 개의 i2b2 NER 작업이 포함됨.
UMLS 2020AA 버전
- UMLS 용어의 식별을 위해 사용된 UMLS의 버전
4. Method
4.1. BERT Model
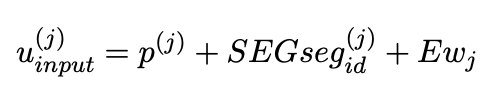
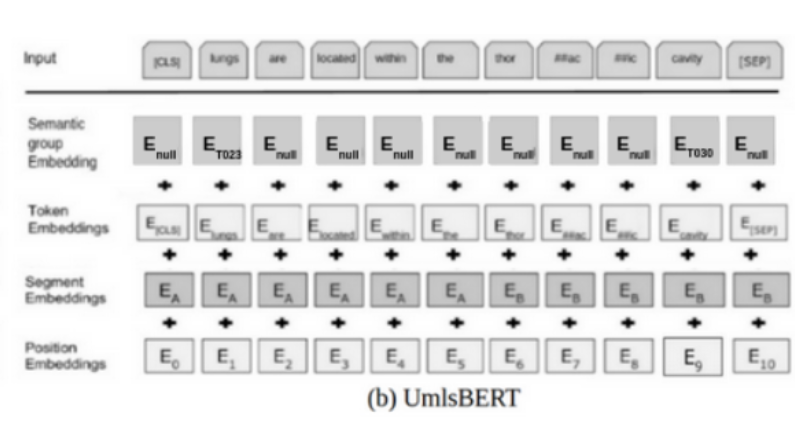
- 입력 문장을 토큰으로 변환하고 토큰을 토큰 임베딩, 세그먼트 임베딩, 위치 임베딩 레이어에 공급하고 임베딩을 얻음.
- 그 뒤 모든 임베딩을 합산해 BERT에 입력으로 제공함.
4.2. Enhancing Contextual Embeddings with Clinical Knowledge
4.2.1. Semantic type embeddings

- 위의 그림은 BERT와 UmlsBERT 입력 벡터 간의 차이점을 보여주며, 후자는
Semantic Type Embedding을 통해 특정 단어들의 Embedding을 강화한다는 것을 시각적으로 나타냄.
- 의료 용어와 관련이 없는 단어들은 0으로 채워진 텐서인
Enull로 사용됨.
lungs와cavity라는 단어는 각각Body Part, Organ, or Organ Component와Body Space or Junction라는 Semantic Type Embedding으로 강화됨.

(1) Semantic Type Embeddings
- 새로운 임베딩 행렬인 ST를 도입하였으며, 이는 BERT의 Transformer 히든 차원과 UMLS 의미 유형의 고유한 수를 기반으로 함.
(2) UMLS 연관
- 각 단어에 대한 UMLS 의미 유형이 있는데,
heart단어는Body Part, Organ, or Organ Component라는 의미 유형과 연관되어 있음.
(3) cTakes 활용
- UMLS 용어와 그들의 UMLS 의미 유형을 식별하기 위해 오픈 소스 Apache clinical Text Analysis and Knowledge Extraction System (cTakes)를 사용함.
(4) 입력 벡터 업데이트
- UMLS에 식별되지 않은 단어에 대해 의미 유형 벡터는 0으로 채워진 벡터로 설정됨.
(5) 가설
- 훈련 코퍼스에서 드문 단어의 입력 벡터를 풍부하게 만들기 위해 의미 유형 표현을 사용할 수 있기 때문에 의미 유형의 임상 정보를 입력 텐서에 포함시키는 것이 모델의 성능에 도움이 될 것이라는 가설을 제시함.
4.2.2. Updating the loss function of Masked LM task
- UmlsBERT의 Masked LM 사전 훈련 작업에 대한 손실 함수는 동일한 CUI (Concept Unique Identifier)를 공유하는 단어들 간의 연결을 고려하여 업데이트 됨.
(1) 변경된 손실 함수
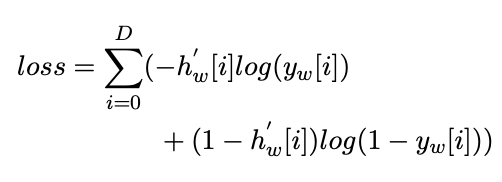
- 기존 BERT 모델의 Masked LM 사전 훈련 작업의 손실 함수는 onehot vector간의 cross entropy Loss였음.
- UmlsBERT에서는 CUI 정보를 사용하여 손실 함수를 '부드럽게' 만들고, multi-label 시나리오로 업데이트함.
(2) 새로운 접근 방식
- masked 단어 w에만 해당하는 one-hot vector(hw)를 사용하는 대신 masked 단어의 동일한 CUI를 공유하는 모든 단어의 존재를 나타내는 이진 벡터 를 사용함.
(3) 손실 함수 업데이트
- multi label 시나리오에서 제대로 작동하도록 cross entropy loss를 binary cross entropy loss로 업데이트함.
(4) 결과
- 이러한 변경으로 인해 UmlsBERT는 생물의학적 맥락에서 동일한 CUI와 연관된 단어 간의 의미론적 관계를 학습하게 됨.
- 'lungs'라는 masked 단어를 예측할 때, UmlsBERT 모델은 'lung', 'lungs', 'pulmonary' 모두 동일한 UMLS Metathesaurus의 CUI:
C0024109와 연결되어 있기 때문에 이 세 단어를 식별하려고 시도함.

- 기존 BERT 모델은 오직 'lungs' 단어만을 예측하려고 함.
- UmlsBERT는 동일한 CUI (Concept Unique Identifier)와 연관된 모든 단어 (예: 'lungs', 'lung', 'pulmonary')를 식별하려고 시도함.
4.3. UmlsBERT Training
(1) 초기화
- UmlsBERT는 이미 훈련된 Bio_ClinicalBERT로 초기화
(2) pre-trained
- 이후 UmlsBERT는 MIMIC-III 노트에서 업데이트된 Masked LM 작업을 사용하여 추가적으로 사전 훈련
(3) fine-tuning
- UmlsBERT 위에 단일 선형 계층을 추가하고 해당 작업에 fine tuning
(4) 토큰화
- 든 모델 간에 동일한 어휘와 WordPiece 토큰화를 사용
(5) UMLS Metathesaurus 선택 이유
- 의학 지식을 통합할 수 있는 임상 맥락 임베딩 모델을 만드는 것이 목표였으며, UMLS는 많은 생물의학 어휘의 집합
(6) 훈련 상세 정보
- UmlsBERT는 1,000,000 단계 동안 훈련되며, 배치 크기는 64, 최대 시퀀스 길이는 128, 학습률은 5x10^-5임.
- 모든 다른 하이퍼파라미터는 기본값으로 유지됨.
(7) 하드웨어 사양
- UmlsBERT는 2개의 nVidia V100 16GB GPU와 128GB의 시스템 RAM을 가진 Ubuntu 18.04.3 LTS에서 훈련됨.
5. Results
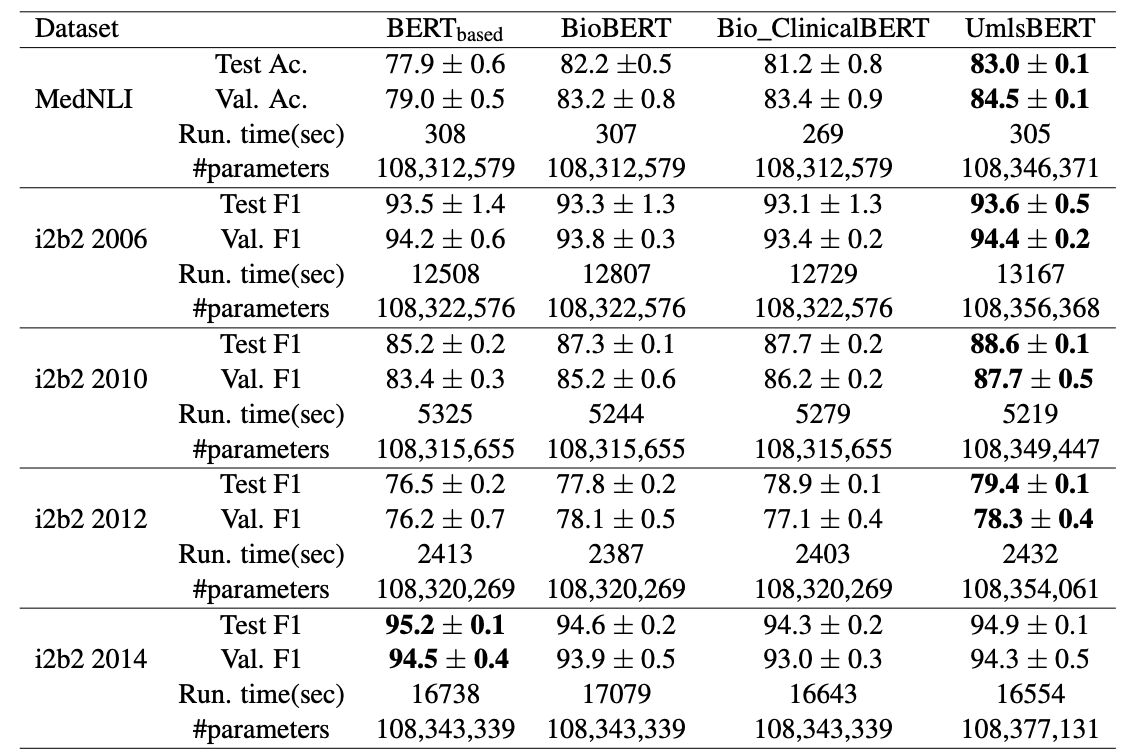
- 다양한 임상 NLP Task에서 제안된 모델의 효율성을 보여주기 위해 사용 가능한 여러 BERT 모델 간의 비교를 제공

- 세 가지 의미적 카테고리(두 개의 임상 및 하나의 일반)에서 여섯 단어의 가장 가까운 이웃을 보여줌.
- UmlsBERT만이 UMLS Metathesaurus의 CUI기반으로 임상적 의미를 가진 단어 연관성을 찾음.
- 반면 일반적인 카테고리에서는 다른 모델과 큰 차이가 없음.
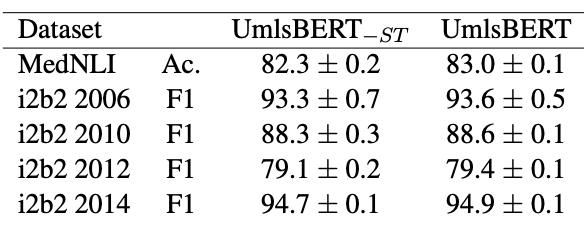
- UmlsBERT의 2가지 버전 (하나는
semantic type embedding을 포함하고, 다른 하나는 포함하지 않음)에 대한 성능 결과를 보여줌.
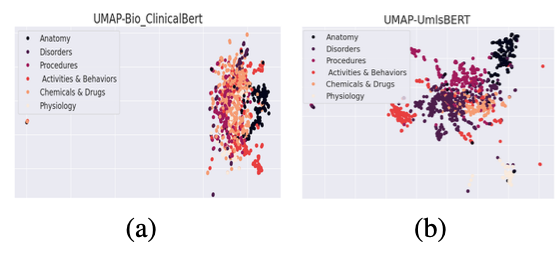
- UMAP 시각화를 통해 2가지 임베딩의 Clustering을 보여줌.
(a) Bio_ClinicalBERT의 입력 Embedding
- 단어 Embedding만 사용
(b) UmlsBERT의 입력 Embedding
- 단어 Embedding + semantic type Embedding 함께 사용
- 더 풍부한 정보를 제공함.
6. Conclusion & Future Work
- 이 Architecture는 의학(생물의학) 분야의 지식을 얻어 단어 임베딩 모델의 사전 훈련 과정에 통합함.
- UmlsBERT는 UMLS Metathesaurus에서 의미적으로 유사한 다양한 임상 용어의 관계를 학습할 수 있음.
- UmlsBERT는 각 (생물의학) 단어의 의미 유형 정보를 활용하여 더 의미있는 입력 임베딩을 생성할 수 있음.
- 수정 사항이 모델의 성능을 향상시킬 수 있음을 확인했으며, UmlsBERT 모델은 다양한 down stream 작업에서 다른 생물의학 BERT 모델들을 능가함.
Future work로
- UmlsBERT의 출력 임베딩 상단에 더 복잡한 계층이 사용될 때 의학 지식을 통합하여 문맥적 임베딩을 확대하는 효과를 검토해보면 좋을 듯함.
- 논문에서 조사한 개념 연결을 확장하는 단어 간의 UMLS 계층적 관계를 탐색해보길
- 델의 장점과 단점을 더 깊게 조사하기 위해 다른 데이터셋 및 생물의학 작업(예: 관계 추출 작업)에서 모델을 테스트 해보면 좋을 듯함.
🎯 Summary
- 저자가 뭘 해내고 싶어 했는가?
- 저자는 UmlsBERT라는 새로운 BERT 기반 아키텍처를 소개함.
- 이 아키텍처는 생물의학 분야의 지식을 단어 임베딩 모델의 사전 훈련 과정에 통합함으로써, 다양한 down stream 의학 NLP 작업에서 기존의 생물의학 BERT 모델들을 능가하는 성능을 달성하려는 것이 목표임.
- 이 연구의 접근 방식에서 중요한 요소는 무엇인가?
- 이 연구의 핵심 요소는 UMLS Metathesaurus에서 의미적으로 유사한 임상 용어의 관계를 학습하도록 UmlsBERT를 설계한 것임.
- 각 (생물의학) 단어의 의미 유형 정보를 활용하여 입력 임베딩을 풍부하게 만드는 것임.
- 어느 프로젝트에 적용할 수 있는가?
- 의학적 문맥에서 단어와 문장의 의미를 더 정확하게 파악하는 프로젝트에 적용될 수 있음.
- 의학적 문서 분류, 임상 용어 인식, 관계 추출 등의 다양한 의학 NLP Task에 적용 가능
- 참고하고 싶은 다른 레퍼런스에는 어떤 것이 있는가?
- BioBERT: a pre- trained biomedical language representation model for biomedical text mining.
- Deep learning with word embeddings improves biomed- ical named entity recognition
- Publicly available clini- cal BERT embeddings.
- 느낀점은?
- UMLS Metathesaurus를 통해 임상 용어의 관계를 학습한 것이 이 BERT의 핵심임.
- Semantic type embeddings을 추가한 부분이 인상적이였음. 기존 BERT 구조에서 특정 단어들인 의학 단어에 도 focus를 둔 부분이 idea가 좋다고 생각함.
- Transformer와 BERT는 2018-2021년도 핫한 모델이였음을 다시 알게 됨. 파생된 BERT 모델을 더 공부해봐야겠음.
📚 References
논문
