LightGBM
- XGBoost보다 학습에 걸리는 시간이 훨씬 적다.
- 메모리 사용량도 상대적으로 적다.
- 카테고리형 피처의 자동 변환과 최적 분할(원-핫 인코딩 등을 사용하지 않- 고도 카테고리형 피처를 최적으로 변환하고 이에 따른 노드 분할 수행)
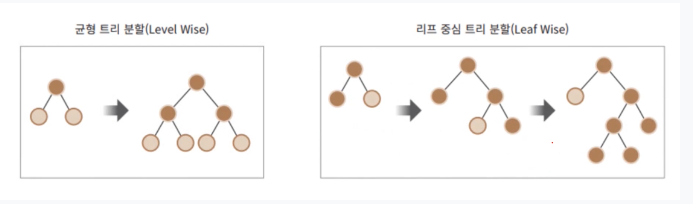
- 일반 GBM 계열의 트리 분할 방법과 다르게
리프 중심 트리 분할(Leaf Wise)방식을 사용한다. - LightGBM의 리프 중심 트리 분할 방식은 트리의 균형을 맞추지 않고, 최대 손실 값(max delta loss)을 가지는 리프 노드를 지속적으로 분할하면서 트리의 깊이가 깊어지고 비대칭적인 규칙 트리가 생성된다.
- 학습 반복시마다 균형 트리 분할 방식보다 예측 오류 손실을 최소화 할 수 있다는 것이 LightGBM의 구현 사상
LightGBM 설치
LightGBM 하이퍼 파라미터
- XGBoost와 많은 부분이 유사하다.
- LightGBM은 Xgboost와 다르게 리프 노드가 계속 분할되면서 트리의 깊이가 깊어지므로 이러한 특성에 맞는 하이퍼 파라미터 설정이 필요하다. (max_depth를 매우 크게 가짐)
주요파라미터
- num_iterations [default=100]: 반복 수행하려는 트리의 개수를 지정한다. 크게 지정할수록 예측 성능이 높아질 수 있으나, 너무 크게 지정하면 오히려 과적합으로 성능이 저하될 수 있다. 사이킷런 GBM과 XGBoost의 사이킷런 호환 클래스의
n_estimators와 같은 파라미터이므로 LightGBM의 사이킷런 호환 클래스에서는 num_iterations로 이름이 변경되었다. - learning_rate [default=0.1]: 0에서 1사이의 값을 지정하며 부스팅 스텝을 반복적으로 수행할 때 업데이트되는 학습률 값이다. 일반적으로 n_estimators를 크게 하고 learning_rate를 작게 해서 예측 성능을 향상시킬 수 있으나, 마찬가지로 과적합 이슈와 학습 시간이 길어지는 부정적인 영향도 고려해야 한다. GBM, XGBoost의 learning_rate와 같은 파라미터이다.
- max_depth [default=-1]: 트리 기반 알고리즘의 max_depth와 같다. 0보다 작은 값을 지정하면 깊이에 제한이 없다. 지금까지 소개한 Depth wise 방식의 트리와 다르게 LightGBM은 Leaf wise 기반이므로 깊이가 상대적으로 더 깊다.
- min_data_in_leaf [default=20]: 결정 트리의 min_samples_leaf와 같은 파라미터이다. 하지만 사이킷런 래퍼 LightGBM 클래스인 LightGBMClassifier에서는 min_child_samples 파라미터로 이름이 변경된다. 최종 결정 클래스인 리프 노드가 되기 위해서 최소한으로 필요한 레코드 수이며, 과적합을 제어하기 위한 파라미터이다.
- num_leaves [default=31]: 하나의 트리가 가질 수 있는 최대 리프 개수이다.
- boosting [default=gbdt]: 부스팅의 트리를 생성하는 알고리즘을 기술한다.
- gbdt : 일반적인 그래디언트 부스팅 결정 트리
- rf : 랜덤 포레스트
- bagging_fraction [default=1.0]: 트리가 커져서 과적합되는 것을 제어하기 위해서 데이터를 샘플링하는 비율을 지정한다. 사이킷런의 GBM과 XGBClassifier의 subsample 파라미터와 동일하기에 사이킷런 래퍼 LightGBM인 LightGBMClassifier에서는 subsample로 동일하게 파라미터 이름이 변경된다.
- feature_fraction [default=1.0]: 개별 트리를 학습할 때마다 무작위로 선택하는 feature의 비율이다. 과적합을 막기 위해 사용된다. GBM의 max_features와 유사하며, XGBClassifier의 colsample_bytree와 똑같으므로 LightGBM Classifier에서는 동일하게 colsample_bytree로 변경된다.
- lambda_l2 [default=0.0]: L2 regulation 제어를 위한 값이다. feature 개수가 많을 경우 적용을 검토하며 값이 클수록 과적합 감소 효과가 있다. XGBClassifier의 reg_lambda와 동일하므로 LightGBMClassifier에서는 reg_lambda로 변경된다.
- lambda l1 [default=0.0]: L1 regulation 제어를 위한 값이다. L2와 마찬가지로 과적합 제어를 위한 것이며, XGBClassifier의 reg_alpha와 동일하므로 LightGBMClassifier에서는 reg_alpha로 변경된다.
Learning Task 파라미터
- objective: 최솟값을 가져야 할 손실함수를 정의한다. Xgboost의 objective 파라미터와 동일하다. 애플리케이션 유형, 즉 회귀, 다중 클래스 분류, 이진 분류인지에 따라서 objective인 손실 함수가 지정된다.
하이퍼 파라미터 튜닝 방안
- num_leaves의 개수를 중심으로 min_child_samples(min_data_in_leaf), max_depth를 함께 조정하면서 모델의 복잡도를 줄이는 것이 기본 튜닝 방안
- num_leaves : 개별 트리가 가질 수 있는 최대 리프의 개수, LightGBM의 주요 파라미터다.
- min_data_in_leaf : 사이킷런 래퍼 클래스에서는 min_child_samples로 이름이 바뀐다. 과적합을 개선하기 위한 주요한 파라미터다.
- max_depth : 명시적으로 깊이의 크기를 제한한다.
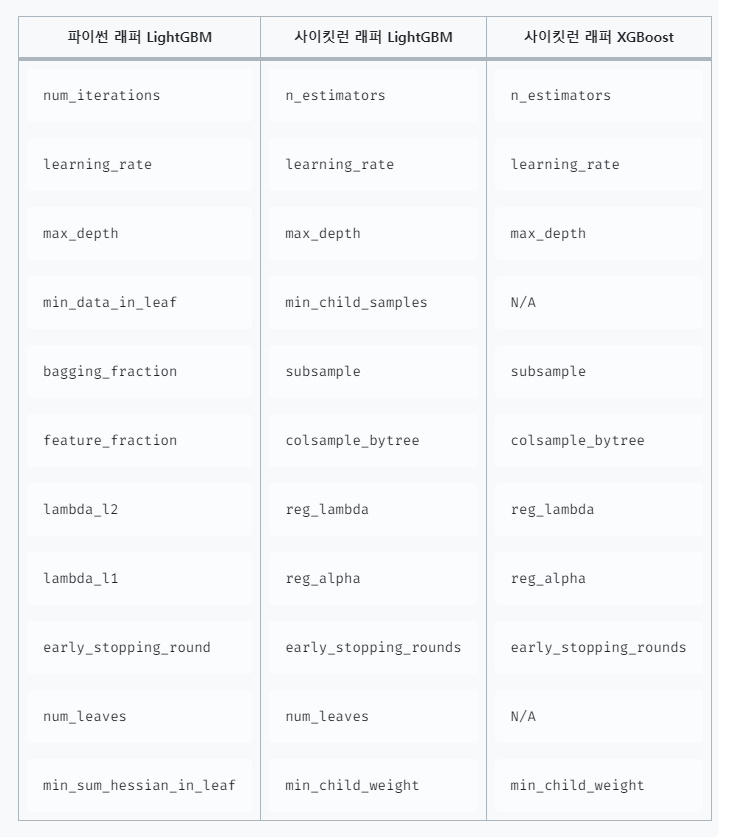
파이썬 래퍼 LightGBM과 사이킷런 래퍼 XGBoost, LightGBM 하이퍼 파라미터 비교
사아킷런 래퍼 LightGBM의 하이퍼 파라미터는 사이킷런 XGBoost에 맞춰서 변경하여 많은 하이퍼 파라미터가 똑같다.
LightGBM 적용 - 위스콘신 유방암 예측
# LightGBM - 위스콘신 유방암 예측
from lightgbm import LGBMClassifier
import pandas as pd
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
dataset = load_breast_cancer()
ftr = dataset.data
target = dataset.target
# 전체 데이터 80, 20 추출
x_train, x_test, y_train, y_test = train_test_split(ftr, target, test_size = 0.2, random_state=156)
# XGBoost와 동일하게 n_estimators 400 설정
lgbm_wrapper = LGBMClassifier(n_estimators=400)
# LightGBM도 XGBoost와 동일하게 조기 중단 수행
evals = [(x_test, y_test)]
lgbm_wrapper.fit(x_train, y_train, early_stopping_rounds=100, eval_metric='logloss',
eval_set=evals, verbose=True)
preds = lgbm_wrapper.predict(x_test)
pred_proba = lgbm_wrapper.predict_proba(x_test)[:,1]# get_clf_eval 활용
# XGB 모델 예측 성능 평가
from sklearn.metrics import confusion_matrix, accuracy_score
from sklearn.metrics import precision_score, recall_score
from sklearn.metrics import f1_score, roc_auc_score
def get_clf_eval(y_test , pred):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
f1 = f1_score(y_test,pred)
roc_auc = roc_auc_score(y_test, pred)
print('오차 행렬')
print(confusion)
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f},\
F1: {3:.4f}, AUC:{4:.4f}'.format(accuracy, precision, recall, f1, roc_auc))
get_clf_eval(y_test, preds)시각화
# 시각화
from lightgbm import plot_importance
import matplotlib.pyplot as plt
%matplotlib inline
fig, ax = plt.subplots(figsize=(10,12))
plot_importance(lgbm_wrapper, ax = ax)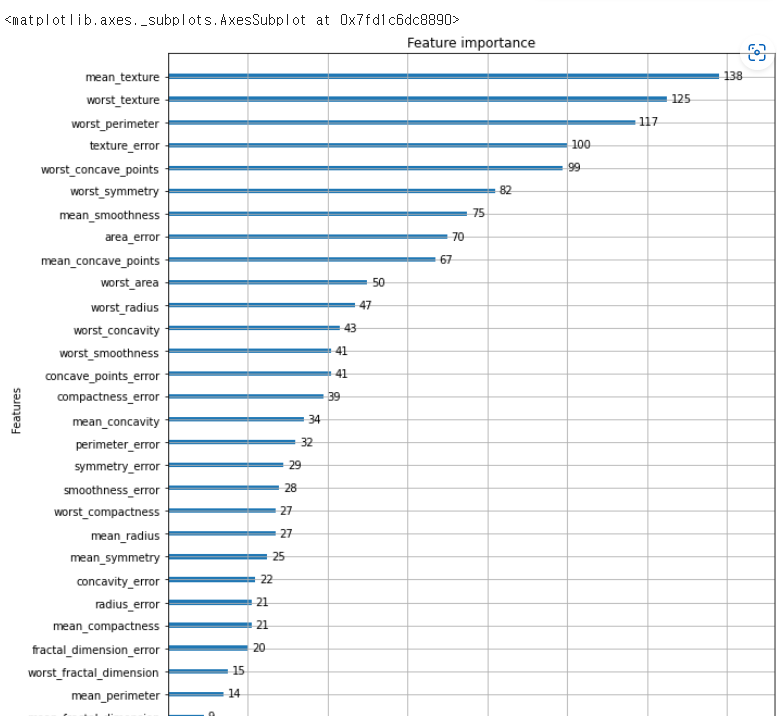
HyperOpt
지금까지는 GridSearchCV를 이용하여 하이퍼 파라미터 튜닝을 수행했다. 하이퍼 파라미터의 수가 많은 XGBoost나 LightGBM과 같은 모델은 GridSearchCV를 이용하여 튜닝 시 많은 수행 시간이 요구된다. (저 모델들이 하이퍼 파라미터가 상대적으로 많기 때문...!)
그렇다면 더 효율적인 하이퍼 파라미터 방식이 있을까? 바로 베이지안 최적화 기반의 HyperOpt가 있겠다!
베이지안 최적화의 개요
-
목적 함수의 식을 제대로 알 수 없는 함수에서, 최대 또는 최소의 함수 반환 값을 만드는 최적 입력값을 가능한 적은 시도를 통해 빠르고 효과적으로 찾아주는 방식
-
베이지안 확률에 기반을 두고 있는 최적화 기법
- 베이지안 확률이 새로운 데이터를 기반으로 사후 확률을 개선해 나가듯이, 베이지안 최적화는 새로운 데이터를 입력받았을 때, 최적 함수를 예측하는 사후 모델을 개선해 나가며 최적 함수 모델을 만들어 낸다.
-
베이지안 최적화의 주요 요소
- 대체 모델 (Surrogate Model) - 획득 함수 (Acquisition Function)대체 모델은 획득 함수로부터 최적 함수를 예측할 수 있는 입력값을 추천받고 이를 기반으로 최적 함수 모델 개선, 획득 함수는 개선된 대체 모델을 기반으로 최적 입력값을 계산하는 프로세스
이때, 입력값은 하이퍼 파라미터에 해당함, 즉, 대체 모델은 획득 함수로부터 하이퍼 파라미터를 추천받아 모델 개선 수행, 획득 함수는 개선된 모델을 바탕으로 더 정확한 하이퍼 파라미터를 계산함!
베이지안 최적화 프로세스
- 랜덤하게 하이퍼 파라미터를 샘플링하고 성능 결과 관측
- 관측된 값을 기반으로 대체 모델은 최적 함수 및 신뢰 구간 (= 결과 오류 편차 = 추정 함수의 불확실성을 의미) 를 추정
- 추정된 최적 함수를 기반으로 획득 함수는 다음으로 관찰할 하이퍼 파라미터를 계산 후 이를 대체 모델에 전달
- 획득 함수로부터 전달된 하이퍼 파라미터를 수행하여 관측된 값을 기반으로 대체 모델 다시 갱신
- 2번 ~ 4번을 반복하며 대체 모델의 불확실성 개선 및 점차 정확한 최적 함수 추정 가능
HyperOpt 사용법 익히기
위에서 정리한 베이지안 최적화 프로세스 2단계에서 대체 모델은 최적 함수를 추정할 때 다양한 알고리즘을 사용할 수 있다. 일반적으로는 가우시안 프로세스 (Gaussian Process) 를 적용하지만, HyperOpt는 트리 파르젠 Estimator (TPE, Tree-structure Parzen Estimator) 를 사용한다.
HyperOpt는 다음과 같은 프로세스를 통해 사용할 수 있다.
- 검색 공간 설정
- 목적 함수 절정
- fmin() 함수를 통해 베이지안 최적화 기법에 기반한 최적의 입력 값 찾기
HyperOpt 설치
!pip install hyperopt
(1) hp 모듈로 입력 변수 및 검색 범위 설정
# p257
## 검색 공간 설정
# hp 모듈을 사용하여 입력 변수명 및 검색 공간 설정
from hyperopt import hp
#-10 ~ 10까지 1긴격을 가지는 입력변수 x와 -15~ 15까지 1간격으로 입력 변수 y설정.
search_space = {"x": hp.quniform("x",-10,10,1),
"y":hp.quniform("y",-15,15,1)}(2) 목적 함수 정하기
from hyperopt import STATUS_OK
# 목적 함수르 생성.
# 변숫값과 변수 검색 공간을 가지는 딕셔너리를 인자로 받고, 특정 값을 반환
def objective_func(search_space):
x = search_space["x"]
y = search_space["y"]
retval= x**2 -20*y
return retval(3) 최적의 함수 유추하기
- 목적 함수의 반환값이 최소가 될 수 있는 최적의 입력 값을 베이지안 최적화 기법에 기반하여 찾아야 함
- HyperOpt는 이러한 기능을 fmin()함수를 통해 제공함.
from hyperopt import fmin, tpe, Trials
import numpy as np
# 입력 결과값을 저장한 Trials 객체값 생성
trial_val = Trials()
# 목적 함수의 최솟값을 반혼하는 최적 입력 변숫값을 5번의 입력값 시도(max_evals= 5)로 찾아냄.
## fmin() 함수는 아래의 주요 인자를 가짐
best_01 = fmin(fn=objective_func, ## 목적 함수
space=search_space, ## 검색 공간
algo=tpe.suggest, ## 베이지안 최적화 적용 알고리즘
max_evals=5, ## 입력 시도 횟수
trials=trial_val ## 시도한 입력 값 및 입력 결과 저장
) ## fmin()을 시도할 때마다 동일한 결과를 가질 수 있도록 설정하는 랜덤 시드
print("best:",best_01)
import numpy as np
trial_val = Trials()
# 목적 함수의 최솟값을 반혼하는 최적 입력 변숫값을 5번의 입력값 시도(max_evals= 5)로 찾아냄.
best_02 = fmin(fn=objective_func, space=search_space,
algo=tpe.suggest, max_evals=20,
trials=trial_val)
print(best_02) (4) trial_val의 results 및 vals 확인
# trial_val의 results 및 vals 확인
trial_val.results
trial_val.vals(5) trial_val을 dataframe 형태로 변환하여 확인
import pandas as pd
losses = [loss_dict['loss'] for loss_dict in trial_val.results]
result_df = pd.DataFrame(
{
'x':trial_val.vals['x'],
'y':trial_val.vals['y'],
'losses':losses
}
)
result_dfHyperOpt을 사용하여 XGBoost 하이퍼 파라미터 튜닝하기 p262
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
import pandas as pd
import numpy as np
### 데이터 로드
## 유방암 데이터셋 로드
dataset = load_breast_cancer()
features = dataset.data
labels = dataset.target
## 데이터를 Pandas DataFrame으로 로드
cancer_df = pd.DataFrame(data=features, columns=dataset.feature_names)
cancer_df['target'] = labels
cancer_df.head(5)
### 데이터 분리
## 학습 및 검증 데이터셋으로 데이터 분리
X_features = cancer_df.iloc[:, :-1]
y_label = cancer_df.iloc[:, -1]HyperOpt 설정 1 - 검색 공간 설정
- 하이퍼 파라미터의 검색 공간을 설정함
# 전체 데이터 80, 20 추출
X_train, X_test, y_train, y_test = train_test_split(X_features, y_label, test_size = 0.2, random_state=156)
# 앞에서 추출한 학습 데이터를 다시 학습과 검증 데이터로 분리
X_tr,X_val,y_tr,y_val = train_test_split(X_train, y_train, test_size=0.1,random_state=156)
##1. 검색 공간 설정
from hyperopt import hp
# max_depth는 5에서 20까지 1간격으로, min_child_weight는 1에서 2까지 1간격으로
# colsample_bytree는 0.5에서 1사이, learning_rate는 0.01에서 0.2 사이 정규 분포된 값으로 검색.
xgb_search_space = {'max_depth': hp.quniform('max_depth', 5, 20, 1),
'min_child_weight': hp.quniform('min_child_weight', 1, 2, 1),
'learning_rate': hp.uniform('learning_rate', 0.01, 0.2),
'colsample_bytree': hp.uniform('colsample_bytree', 0.5, 1),
}
HyperOpt 설정 2 - 목적 함수 설정
- 검색 공간에서 설정한 하이퍼 파라미터들을 입력 받아서 XGBoost를 학습시키고, 평가 지표를 반환하도록 구성되어야 함
## 2. 목적 함수 설정 p264
## 검색 공간에서 설정한 하이퍼 파라미터들을 입력 받아서 XGBoost를 학습시키고, 평가 지표를 반환하도록 구성되어야 함.
from sklearn.model_selection import cross_val_score
from xgboost import XGBClassifier
from hyperopt import STATUS_OK
# fmin()에서 입력된 search_space 값으로 입력된 모든 값은 실수형임.
# XGBClassifier의 정수형 하이퍼 파라미터는 정수형 변환을 해줘야 함.
# 정확도는 높을수록 더 좋은 수치임. -1 * 정확도를 곱해서 큰 정확도 값일수록 최소가 되도록 변환
def objective_func(search_space):
# 수행 시간 절약을 위해 nestimators는 100으로 축소
xgb_clf = XGBClassifier(n_estimators=100, max_depth=int(search_space['max_depth']),
min_child_weight=int(search_space['min_child_weight']),
learning_rate=search_space['learning_rate'],
colsample_bytree=search_space['colsample_bytree'],
eval_metric='logloss')
accuracy = cross_val_score(xgb_clf, X_train, y_train, scoring='accuracy', cv=3)
# accuracy는 cv=3 개수만큼 roc-auc 결과를 리스트로 가짐. 이를 평균해서 반환하되 -1을 곱함.
return {'loss':-1 * np.mean(accuracy), 'status': STATUS_OK} HyperOpt 설정 3 - fmin()을 사용하여 최적 하이퍼 파라미터 찾기
## 3. fmin()을 사용하여 최적 하이퍼 파라미터 찾기
from hyperopt import fmin, tpe, Trials
trial_val = Trials()
best = fmin(fn=objective_func,
space=xgb_search_space,
algo=tpe.suggest,
max_evals=50, # 최대 반복 횟수를 지정합니다.
trials=trial_val)- 획득한 최적의 하이퍼 파라미터를 이용하여 XGBoost의 인자로 입력
- 입력 전에 정수형 하이퍼 파라미터의 형변환은 필수!
## 모델 로드
# 획득한 최적의 하이퍼 파라미터를 이용하여 모델 선언
xgb_wrapper = XGBClassifier(n_estimators=400,
learning_rate=round(best['learning_rate'], 5),
max_depth=int(best['max_depth']),
min_child_weight=int(best['min_child_weight']),
colsample_bytree=round(best['colsample_bytree'], 5)
)
## 모델 학습
## early stopping
evals = [(X_tr, y_tr), (X_val, y_val)]
## model train
xgb_wrapper.fit(
X_tr, y_tr,
early_stopping_rounds=50,
eval_metric='logloss',
eval_set=evals,
verbose=True
)
## 모델 평가
## eval
preds = xgb_wrapper.predict(X_test)
pred_proba = xgb_wrapper.predict_proba(X_test)[:, 1]
get_clf_eval(y_test, preds, pred_proba) 요약
GridSearchCV를 이용하여 XGBoost, LightGBM 하이퍼 파라미터를 튜닝할 경우 수행 시간이 오래 걸린다는 단점이 있다. 이때 생각할 수 있는 것이 바로 베이지안 최적화 기반의 HyperOpt이다.
HyperOpt
- 1) 검색 공간 설정
- 2) 목적 함수 설정
- 3) fmin() 함수를 이용한 최적 하이퍼 파라미터 찾기 순서로 진행된다. 가령 HyperOpt를 이용하여 XGBoost 하이퍼 파라미터를 최적화 할 경우,
- 1) 데이터 로드 및 분리
- 2) HyperOpt로 최적 하이퍼 파라미터 찾기
- 3) 최적 하이퍼 파라미터를 XGBoost 인자로 입력하여 모델 학습 및 평가 순으로 진행할 수 있겠다.
캐글 산탄데르 고객 만족 예측
캐글의 산탄데르 고객 만족 데이터셋에 대해 LigthGBM과 XGBoost를 활용하여 예측한다. feature은 총 370개, 클래스 레이블 명은 TARGET이고, 이때 해당 값이 1이면 불만을 가진 고객, 0이면 만족한 고객을 의미한다.
이때, 불균형 데이터셋이므로 모델 성능 평가는 정확도가 아닌 roc-auc로 진행한다.
데이터 확인
370개의 feature, 1개의 class 열로 구성된 dataframe
260개의 정수형 feature, 111개의 실수형 feature로 구성됨
결측값은 없음
출처:
https://asthtls.tistory.com/m/1277
https://velog.io/@bansohi/ML-ch4.8
https://velog.io/@sset2323/04-07.-LightGBM