1. 학습내용
계속해서 Scikit-Learn을 활용해 여러 알고리즘을 써보는 것을 실습했다.
설명이 안된 부분이 있다고 하셔서, 먼저 Regularization(정규화)과 L1, L2에 대해 배웠다.
Normalization(일반화) : 데이터에 scale을 조정하는 작업
Regularization(정규화) : predict function에 복잡도를 조정하는 작업
일반화는 어떤 데이터가 특정 데이터에 편향된 것을 줄이거나 늘여서 데이터 전체를 일관되게 유지하는 것을 말한다.
정규화는 규칙들을 제어해서 원하는 결과를 얻어 내는것을 말한다.
제약이라고 L1제약 , l2제약이 있다.
L1제약은 Lasso 같은 툴이, L2는 Lidge 툴을 말한다.
-Lasso (제약을 세계걸면 특성이 사라짐. linear regression과 거의 같아짐)
#Lasso
from sklearn.linear_model import Lasso
lasso = Lasso().fit(X_train, y_train)
print(lasso.score(X_train, y_train))
print(lasso.score(X_test, y_test))결과값: 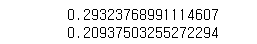
-Lidge
# Ridge / 릿지
from sklearn.linear_model import Ridge
ridge = Ridge().fit(X_train, y_train)
print(ridge.score(X_train, y_train))
print(ridge.score(X_test, y_test))결과값: 
L2제약은 각각의 데이터의 특성에 맞는 가중치를 적용해 전반적으로 제약을 적용하는것. (0이 되지 않음)
L1제약은 가중치가 큰 데이터는 크게, 적은건 적게 남아 약한것은 0에 가깝게 적용하는것이다.
어떤 알고리즘이 맞는 지 귀납적인 접근을 통해 하는 방법밖에 없다고.(일일히 실험해봐야..)
일반적으로 lidge가 성공할 확률이 높긴한데, 그래도 다 해봐야 한다.
Lasso 알고리즘을 할때 alpha 값을 이용을해서 오차를 줄일려는 노력을 한다. 이때 내부적으로 반복을 한다. 이때 최대 반복 횟수를 적는다 몇번 반복해서 이 값이 안뜨면 경고창이 뜬다.
보통 이걸 max_iter라고 한다. 그러나 저 값을 백만이라 적는다고 해서 백만번 까지 돌리지는 않는다. 그래프를 그렸을때 해당되는 값이 최적의 지점에 다다랐을 때 까지 돌리는 것이다.
여유있게 제약을 두지 않아도 에러가 뜬다.
lasso001 = Lasso(alpha=0.01, max_iter=50000).fit(X_train, y_train)
print(lasso001.score(X_train, y_train))
print(lasso001.score(X_test, y_test))여기서 alpha값은 패널티의 효과를 조절해주는 파라미터인데, alpha의 값이 커지면 패널티의 영향력이 커지고, alpha의 값이 작아질수록 선형회귀와 같아진다.
아래는 각각 알파값을 lasso에 0.01과 0.0001값을 주었을 때 나타나는 train값과 test값이다.
0.01:
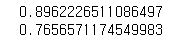
0.0001:
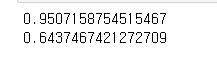
lasso는 제약을 심하게 걸면 불필요하다고 생각했던 것들을 빼고 학습을 해버리는 경우가 생긴다.
어제한 내용중에 fit이 있었는데 이 fit() 메서드는 선형 회귀 모델에 필요한 두 가지 변수를 전달하는 거다. 데이터를 또한 알고리즘에 적용시킨다고 볼수 있다.
어제 내용을 다시 복습하기 위해 그래프의 절편과 기울기를 나타내는 걸 정리해 보았다.
기울기: linefitter.coef
절편: linefitter.intercept
어쨌든 저 Lasso를 이용한 결과값을 그래프로 보기 위해 아래와 같이 출력했다. (안에 결정계수를 보기 위함)
plt.plot(lasso.coef_, 's', label='Lasso alpha=1')
plt.plot(lasso001.coef_, '^', label='Lasso alpha=0.01')
plt.plot(lasso00001.coef_, 'v', label='Lasso alpha=0.0001')
plt.plot(lasso01.coef_, 'o', label='Lasso alpha=0.1')
plt.legend()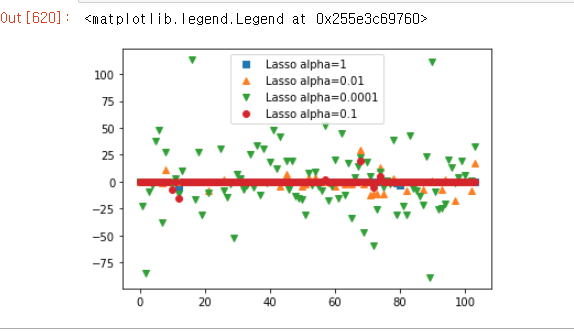
제약을 거니 약한 것은 전부 0에 붙어버린것을 알수 있다.
또 많이 활성화 된 것이 0.01이다. 이건 데이터의 특징을 0.01이 잘잡아내었고, 나머지는 무시했다는 뜻이다.
제약을 건다는건 데이터에 대해 과도하게 집착하는 것을 막는것 overfitting이고 이걸 막는게
전반적으로 속성을 죽이는게 Lidge알고리즘 그냥 되는 데이터들만 살리는 것을 Lasso알고리즘 이렇게 정리했다.
#분류에 대한 선형 모델
from sklearn.linear_model import LogisticRegression #sklearn패키지가 다름
from sklearn.svm import LinearSVC
X, y = mglearn.datasets.make_forge() #forge는 인위적으로 만들어진 분류 데이터 셋이다.
fig, axes = plt.subplots(1,2, figsize=(10,3)) #figsize(가로길이,세로길이) 그래프 사이즈 .subplots은 한번에 여러 그래프를 보여주기 위해 사용되는 코드.뒤에 's'가 붙으면 한번에 그래프개수를 설정할 수 있음
for model, ax in zip([LinearSVC(max_iter=5000), LogisticRegression()], axes):
clf = model.fit(X, y)
mglearn.plots.plot_2d_separator(clf, X, fill=False, eps=0.5, alpha=0.7, ax=ax)
mglearn.discrete_scatter(X[:,0], X[:,1], y, ax=ax)zip함수는 여러 개의 순회 가능한(iterable) 객체를 인자로 받고, 각 객체가 담고 있는 원소를 터플의 형태로 차례로 접근할 수 있는 반복자(iterator)를 반환한다. 그러니까 각 리스트를 짝지어 출력해준다고 보면 된다.
옵션을 주는걸 파라메터, 원하는 값에 근접해 가는 과정을 hyper parameter라고 한다.
데이터 전처리 과정 -> 알고리즘 선택 -> hyper parameter -> 최적화된 model
어제진도 나간 '유방암 데이터'를 이용한 LogisticRegression를 이어서 또 나갔다.
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target,
random_state=42)
logreg = LogisticRegression(max_iter=5000).fit(X_train, y_train)
print(logreg.score(X_train, y_train))
print(logreg.score(X_test, y_test))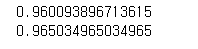
LogisticRegression은 hyper parameter를 할 수 있는 값이 또 있는데, 'C'라고 하는 옵션이다.
logreg100 = LogisticRegression(max_iter=5000, C=100).fit(X_train, y_train)
print(logreg100.score(X_train, y_train))
print(logreg100.score(X_test, y_test))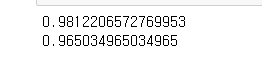
'C'는 오류에 대한 허용치를 말한다. 기본값은 1이다. 크면 클수록 알고리즘 내에 오류를 허용하지 않겠다는 의미이다. 조금더 타이트하게 살펴보면서 원하는 값이 나올때 까지 돌려보겠다는 말이다. 그런데 둘이 큰차이가 없다 (기본값이나 100이나) 이말은 제약 1안에서 오류가 다 잡힌다는 뜻이다.
#C가 0.01일때
logreg001 = LogisticRegression(max_iter=5000, C=0.01).fit(X_train, y_train)
print(logreg001.score(X_train, y_train))
print(logreg001.score(X_test, y_test))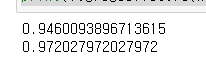
오류를 좀 느슨하게 준 편이다. 테스트 데이터로 하면 오류가 떨어진다. 오히려 새로운 데이터에 대한 적응력을 높이는 방법이기도 하다.
위에서 본 데이터에서 C값을 타이트 하게 줄수록 (값이 높을수록) 선이 데이터에 맞춰 경사가 가팔라지는 것을 알수있다.
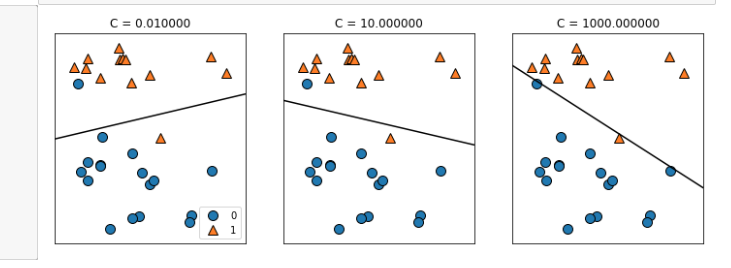
다음엔 카테고리컬 데이터에서 두개이상 3개 정도가 되는 것을 표현하는 알고리즘을 배웠다.
from sklearn.datasets import make_blobs #blob은 얼룩이란뜻.
대부분 텍스트나 바이너리 파일등 blob이란 스토리지에 저장된다.
X, y = make_blobs(random_state=42) #X는 데이터, y는 타겟
mglearn.discrete_scatter(X[:,0],X[:,1], y) #0에 있는데이터 x축, 1에 있는 데이터 y축, 색은 y로 결정.
plt.xlabel('feature 0')
plt.ylabel('feature 1')
plt.legend(['Class 0', 'Class 1', 'Class 2']) #데이터 라벨링 따로 하지 않고 강제로 list타입으로 이름 지어주는 방식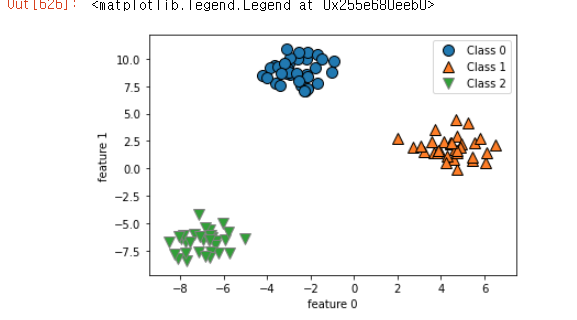
이런 식으로 나오는 건 한번의 선으로 구분 할수 없기 때문에, 식을 적어서 따로 적어주어야 한다.
보통 선형 그래프는 식이 y=xw+b이다. (x값이 피쳐값, w는 기울기, b가 절편이다.)
그런데 이번에 그릴려는 그래프의 선은 1개가 아닌 3개가 가로 질러 그려져야 하므로
식을 다르게 쓴다.
아래와 같이 쓴다.
linear_svm = LinearSVC().fit(X, y)이걸 프린트해보면, 3개의 절편값이 나오고, 데이터의 모양을 알수 있다.

mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
line = np.linspace(-15, 15) #라인 값 설정.
for coef, intercept, color in zip(linear_svm.coef_,
linear_svm.intercept_,
mglearn.cm3.colors): #색상표
plt.plot(line,-(line * coef[0] + intercept) / coef[1], c=color)
#line이 x축 -(line~coef[1]까지 y축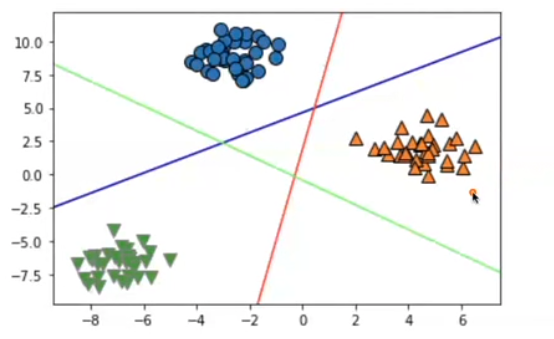
3개의 선이 겹치는 중간을 기점으로 3가지 영역을 나눌수 있다.
mglearn.plots.plot_2d_classification(linear_svm, X, fill=True)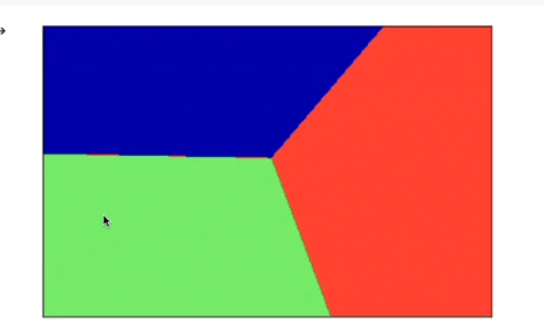
이걸 위의 3개의 선이 있는 그래프와 겹쳐지게 만들면 최종적으로 아래와 같이 나온다.
mglearn.plots.plot_2d_classification(linear_svm, X, fill=True, alpha=0.5)
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
line = np.linspace(-15, 15)
for coef, intercept, color in zip(linear_svm.coef_,
linear_svm.intercept_,
mglearn.cm3.colors):
plt.plot(line,-(line * coef[0] + intercept) / coef[1], c=color)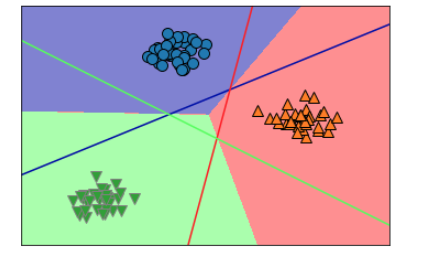
liner방법을 알아봤는데, 문제점이 있다면 3개의 클래스의 간격이 서로 영역을 침범하는게 많아질수록 이방법이 분류하는게 정확도가 떨어질 수 있다.
이때 사용하는 방법이 tree를 사용한 방법이다.
# Tree 계열 분류 알고리즘
mglearn.plots.plot_tree_progressive()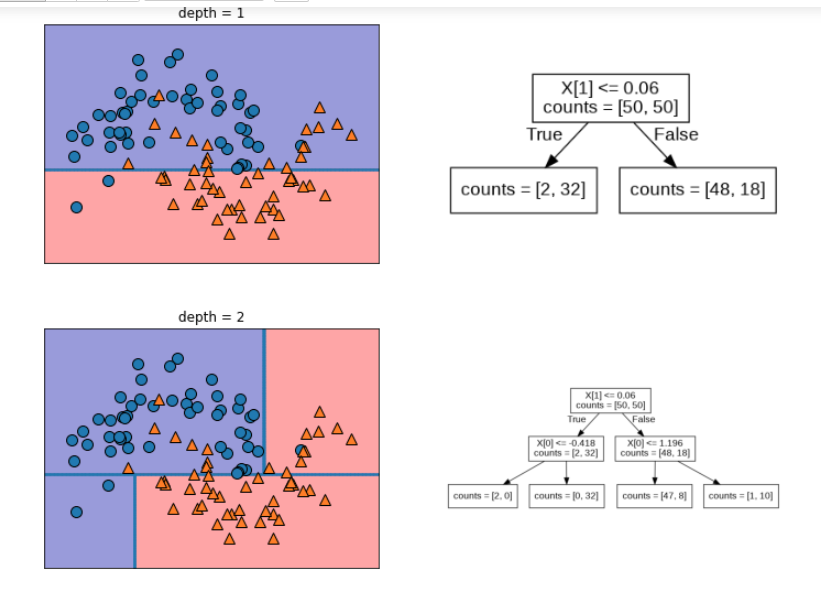
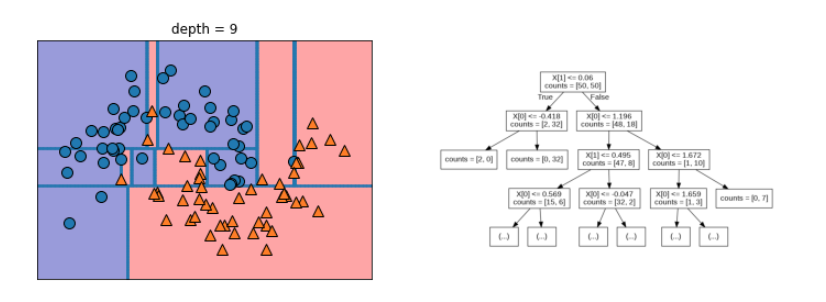
depth는 참고로 내려가는 단계다. (약간 벨런스 게임 같은 느낌..) 내려가면 갈수록 세세하게 분류하기 시작한다. 여기서 그래서 depth를 결정해서 어느 수치가 일반화가 잘되는지 봐야한다.
이걸 '유방암 데이터'를 이용해 사용해 보기로 했다.
from sklearn.tree import DecisionTreeClassifier
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data,
cancer.target,
random_state=42)
tree = DecisionTreeClassifier(random_state=0)
tree.fit(X_train, y_train)
print(tree.score(X_train, y_train))
print(tree.score(X_test, y_test))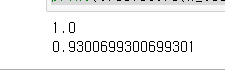
1.0이 나왔다는 건 할때까지 돌려봤다는 이야기다.
test값도 높게 나왔는데 이뜻은 경계를 어느정도 잘 학습하고 있다는 뜻이다.
이번에는 max_depth를 이용해 한계치를 지정해 주고 해보았다
지정해주는 것을 데이터의 과적합을 막기위해서다.
tree = DecisionTreeClassifier(max_depth=4, random_state=0)
tree.fit(X_train, y_train)
print(tree.score(X_train, y_train))
print(tree.score(X_test, y_test))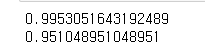
학습은 내려갔지만 test는 올라갔다. 너무 디테일하게 안쪼개니까 오히려 새로운 데이터에 대한 적응력이 좋아졌기 때문이다.
다음은 tree를 그리는 패키지를 활용해 보았다.
from sklearn.tree import plot_tree #tree를 그리는 기능
plot_tree(tree, class_names=['A','B'], filled=True, fontsize=4,
feature_names=cancer.feature_names) #위에 했는 tree라는 모델이 남아 있어 그데이터를 썼다. 그 밖에는 클래스에 이름을 붙이고, 폰트 크기를 조절했다.
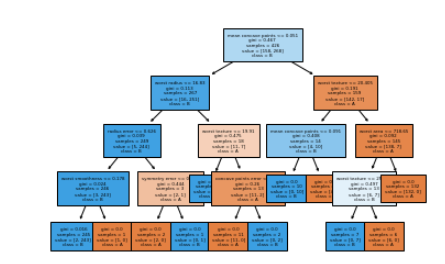
색깔이 옅은건 1번클래스에 가까운데 다른 클래스의 요소도 있다는 의미이다.
print(tree.feature_importances_) #전체 데이터의 중요도각 트리의 중요도를 백분율로 보여주고 있다.
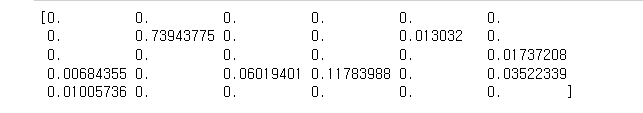
중요도를 봤을때 어느쪽에 데이터가 많이 쏠려있는지 알수 있다.
이번에는 새로운 데이터 파일을 업로드 해서 보았다.
여기서 os 모듈을 썼다.
(os 모듈은 Operating System의 약자로서 운영체제에서 제공되는 여러 기능을 파이썬에서 수행할 수 있게 해준다.
예를 들어, 파이썬을 이용해 파일을 복사하거나 디렉터리를 생성하고 특정 디렉터리 내의 파일 목록을 구하고자 할 때 os 모듈을 사용하면 된다.)
import os
ram_price = pd.read_csv('data/ram_price.csv')
plt.semilogy(ram_price.date, ram_price.price) #y축만 로그스케일로 표현할때,그래프임.
plt.xlabel('Year')
plt.ylabel('price')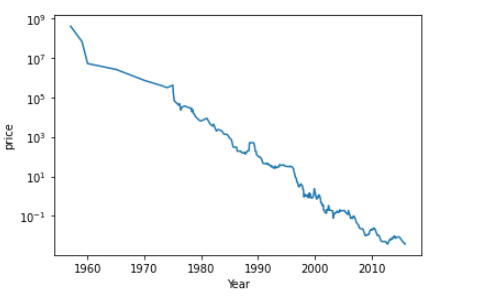
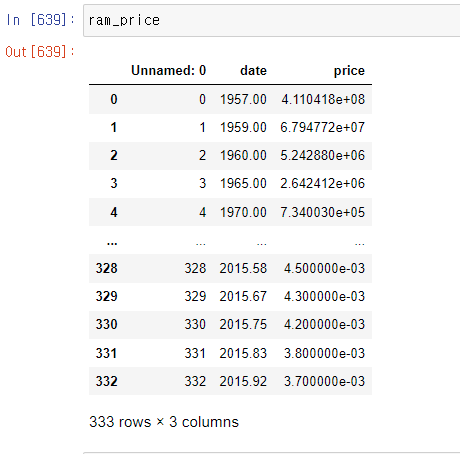
그래프에 나온 것 중에 90년대와 20년대를 반으로 나눠서 보고 싶다. 면 아래와 같이 하면된다.
from sklearn.tree import DecisionTreeClassifier
data_train = ram_price[ram_price.date < 2000]
data_test = ram_price[ram_price.date >= 2000]그런다음 학습데이터를 변수로 선언하는데 여기서 에러가 나와서 뒤에 [:,np.newaxis]를 추가했다.
X_train = data_train.date.to_numpy()[:,np.newaxis]
#numpy배열로 바꾸고, 학습용데이터로 바꾸기 위해 reshape[-1,1]로 해도 되고, 위와 같이 슬라이싱 하겠다는 의미로 []안에, 전체선택으로 ':'넣고, 새로운 축을 늘여주는 np.newaxis라고 입력한다.y_train = np.log(data_train.price) #y는 '가격'이므로 numpy배열인 log로 싸서 만들어야 한다. log는 데이터의 변별력을 높이기 위해 사용됨. 금액이 비슷해 변별력 세우기 위해 씀.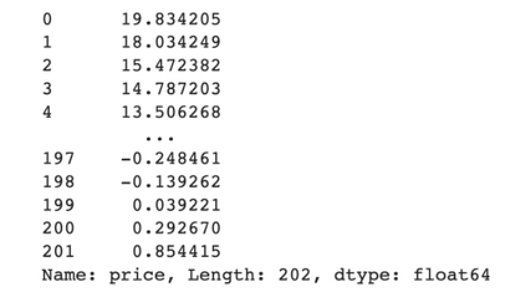
from sklearn.tree import DecisionTreeRegressor
tree = DecisionTreeRegressor().fit(X_train, y_train)
linear_reg = LinearRegression().fit(X_train, y_train)또 X_all이란 변수를 만들어 축을 추가해 준다.
X_all = ram_price.date.to_numpy()[:, np.newaxis] #전체 날짜.그런다음 예측되는 값을 만들고 변수로 저장한다.
pred_tree = tree.predict(X_all) #X로 예측한 price가 나옴.
pred_lr = linear_reg.predict(X_all) #linear reg의 알고리즘으로 함.근데 저건 log로 나오므로 원래 값으로 되돌릴려면 .exp로 할수 있다.(log로 해도 되는데 그럼 값이 튀게 나옴)
price_tree = np.exp(pred_tree)
price_lr = np.exp(pred_lr)price_tree
그럼 원래 값으로 돌아온 것을 알수 있다.
이걸로 그래프를 그리면 아래와 같이 나온다.
plt.semilogy(data_train.date, data_train.price, label='Train data')
plt.semilogy(data_test.date, data_test.price, label='Test data')
plt.semilogy(ram_price.date, price_tree, label='predict Tree')
plt.semilogy(ram_price.date, price_lr, label='Predict regression')
#선형회귀 사이사이 오차도 있다. 이걸 모아 평가하는게 MAE(mean absolute error).
장점은 90년도만 학습했는데도, 20년도 까지 학습이 가능하다.
plt.legend()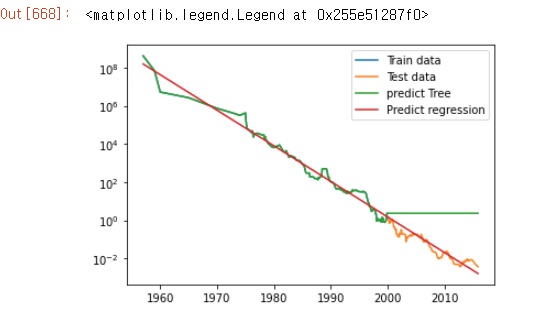
여기서 예측값이 가다가 끊기고 쭉 그대로 가버린다. 예측은 확인할 수 있는 값만 예측하고 그 이후는 본래 값이 없기 때문에 저렇게 직선으로 나오는 것이다.
tree 알고리즘을 봤으니 이제 forest알고리즘을 배워보았다.
# RandomForestclassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import make_moons
X, y =make_moons(n_samples=100, noise=0.25, random_state=3) #noise는 오차를 만드는것.
데이터가 나오는데 또 학습용과 test용으로 나눈다.
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)그런다음 알고리즘을 적용시킨다.
forest = RandomForestClassifier(n_estimators=5, random_state=2)#n_estimators옵션을 줄수 있음. 100개까지 줄수 있으며 100개의 나무를 줄수 있다는 의미.
forest.fit(X_train, y_train)그래프로 보기위해 5개의 그림판을 만들기 위해 아래와 같이 써준다.
fig, axes = plt.subplots(2,3, figsize=(20,10)) for문을 쓴다.
for i, (ax, tree) in enumerate(zip(axes.ravel(), forest.estimators_)) # estimators에 5개의 tree 값이 들어감.
표에 숫자를 매길때 enumerate을 써 zip을 감싸줌. 열거형으로 만들어줌. 열거형은 반복가능한 자료형으로 만들어준다. index넘버가 i에 들어가고 축axes.ravel이 ax에 들어가고 forest가 tree에 들어간다.
for i, (ax, tree) in enumerate(zip(axes.ravel(), forest.estimators_)):
ax.set_title('tree {}'.format(i))
mglearn.plots.plot_tree_partition(X, y, tree, ax=ax) #각각의 요소들이 어떻게 나눠져 있는지 쉽게 보여줌.
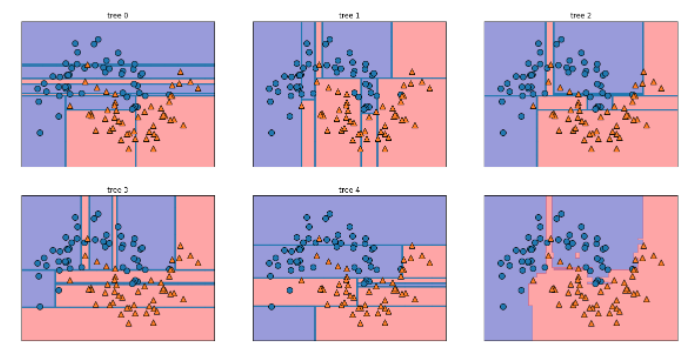
그럼 각각 그래프에 제목이 표시되고, tree형태로 출력되는 것을 알수 있다. (위에 랜덤 값 지정해서 새로 고침할때 마다 그래프가 다른게 나온다.)
마지막은 원래 비어 있는데 나머지 5개의 그래프를 합친게 표시되게 끔 할려면 아래와 같이 추가한다
mglearn.plots.plot_2d_separator(forest, X, fill=True, eps=0.5,ax=axes[-1,-1],
alpha=0.4)
#ax는 [-1,-1]란게 0뒤의 -1은 뒤로 돌아가서 3번째 순번이고, -1은 열의 마지막에서 돌아 가는 것이기 때문에 맨 마지막 순번인 5번째 칸이란 뜻이다. 여기서 알파값은 투명도. eps는 매게변수에서 0.5보다 낮은게 있으면 무시하는것.여기서 실제 데이터를 찍어보는 것을 추가해보면
mglearn.discrete_scatter(X[:,0], X[:,1], y)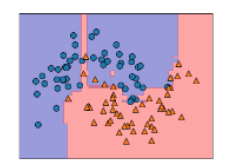
이렇게 나온다.
이걸 또 분석을 해보면 아래와 같이 나온다. 우선 쪼게고 난 뒤에 정확도를 본다.
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state=0)
forest = RandomForestClassifier(n_estimators=100, random_state=0)
forest.fit(X_train, y_train)print(forest.score(X_train, y_train))
print(forest.score(X_test, y_test))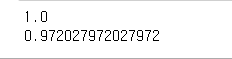
tree하나만 썼을때 보다 결과가 스무스하게 달라지는 것을 알수 있다.
이번엔 다른 tree 알고리즘을 배워보았다. GradientBoost알고리즘이다.
from sklearn.ensemble import GradientBoostingClassifier
X_train, X_test, y_train, y_test = train_test_split(cancer.data,
cancer.target,
random_state=0)gbrt = GradientBoostingClassifier(random_state=0)
gbrt.fit(X_train, y_train)
print(gbrt.score(X_train, y_train))
print(gbrt.score(X_test, y_test))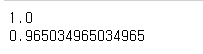
위의 다른 알고리즘보다 조금 떨어진다고 생각할 수 있다. 이때 제약을 달리해 보았다.
gbrt = GradientBoostingClassifier(random_state=0, max_depth=1)
gbrt.fit(X_train, y_train)
print(gbrt.score(X_train, y_train))
print(gbrt.score(X_test, y_test))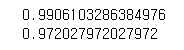
오히려 test정확도가 올라간 것을 볼수 있다.(학습정확도가 떨어짐) GradientBoost는 오차를 스스로 보정하는 옵션이 추가되어 있음. 그래서 depth를 깊게 안내려가도 보정이 되도록 되어있음. 적은 depth를 써도 정확도가 높게 나옴. 즉 적은 메모리를 써도 나온다는 말이다. 보통 4~5단계에서 해결이 가능하다. 5단계인데도 학습이 잘되면 단계를 낮춰야 한다.
다른 옵션을 추가했다.
gbrt = GradientBoostingClassifier(random_state=0, learning_rate=0.1) #학습량 자체제어
gbrt.fit(X_train, y_train)
print(gbrt.score(X_train, y_train))
print(gbrt.score(X_test, y_test))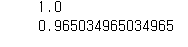
0.01을 했을때 학습정확도, 테스트정확도 모두 떨어졌다. 그래서 0.1 로 고쳤더니 원래와 같이 나왔다. learning_rate는 학습량의 복잡도를 제어할 수 있다.
정확히는 아래와 같다.
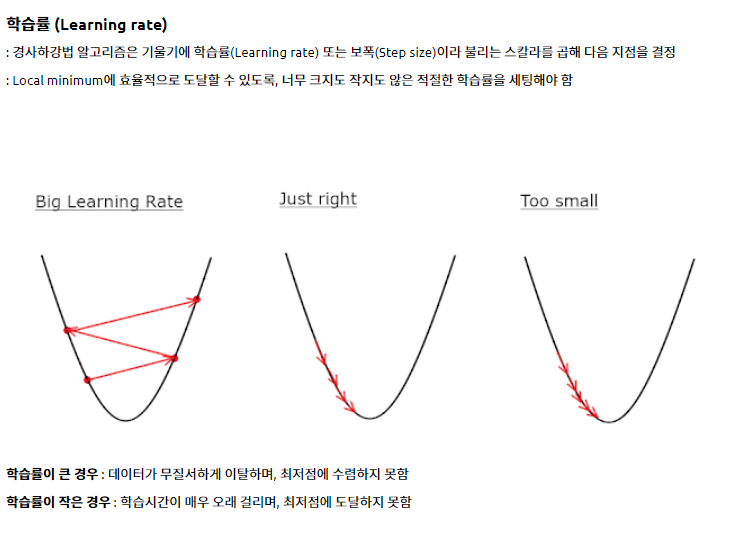
참고자료: https://bioinformaticsandme.tistory.com/130
2. 어려웠던 점 및 해결 방안
오늘도 전반적으로 어려웠지만, 설명을 놓친게 있어서 동영상을 다시 들었다.
마지막 부분이었는데 바로 아래와 같다.
X,y = make_blobs(centers=4, random_state=8) #centers:클러스터 수 혹은 중심, 기본값 3
y = y % 2
mglearn.discrete_scatter(X[:,0], X[:,1], y)
plt.xlabel('feature 0')
plt.xlabel('feature 1')여기서 4개의 그룹이 나오는데 원하는데로 2개씩 묶고 싶다 했을때, y =했을 때 홀수 짝수 끼리 묶는다 했을 때 2를 나눴을때 '0'이 되면(실제로 0이 안됨) 짝수가 된다. 파이썬에선 0나머지를 버린다는 의미에서 %을 쓴다. 그럼 아래와 같이 같은 그룹끼리 색을 나눠서 보여 준다.
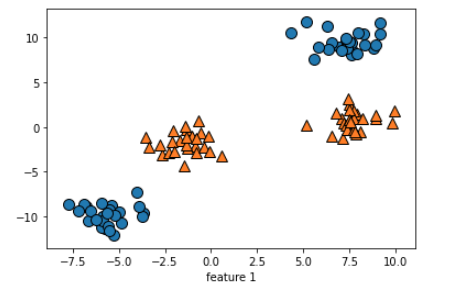
이걸 일반적인 linear regression로 나눌수 없기 때문에 다른 방법을 쓴다고 하셨다. 이걸 내일 배우게 된다.
3. 학습소감
이제 점점 인터넷에 찾아보지 않으면 모르는 것들이 늘어난다. 아직은 괜찮다고 되뇌이며 복습하고 있다. 끝까지 완주해보고 싶은 마음이 드는 하루였다.
