
🍒 AutoML을 활용해서 모델을 탐색해보자.
다양한 AutoML이 존재하지만 이번에는 PyCaret을 써보자!
- autogluon, pycare, supervised의 AutoML 등 다양한 것이 존재함
AutoML 만으로도 꽤나 좋은 성능을 보임
- 다양한 라이브러리와 모델을 비교하는 것이 포인트로 생각됨
01. 바로 써보기
!conda install pycaret
# !pip install pycaretfrom pycaret.regression import *
# data와 target 변수 전달
# 정규화와 변환 적용
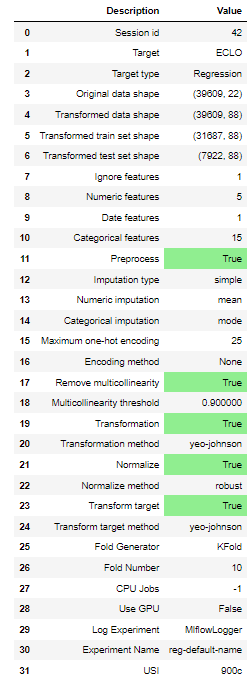
sol= setup(data = train_df[target_column],
session_id=42,
target = 'ECLO',
normalize = True,
transformation = True,
log_experiment = True,
remove_multicollinearity = True,
transform_target = True,
normalize_method='robust',
train_size = 0.8,
date_features = ['사고일시'],
ignore_features=['ID'],
)
# 변환된 데이터의 컬럼 확인
# 다중 공선성으로 인해 제거된 컬럼이 있을 수 있음
sol.X_train_transformed.columns모델 비교
best = compare_models()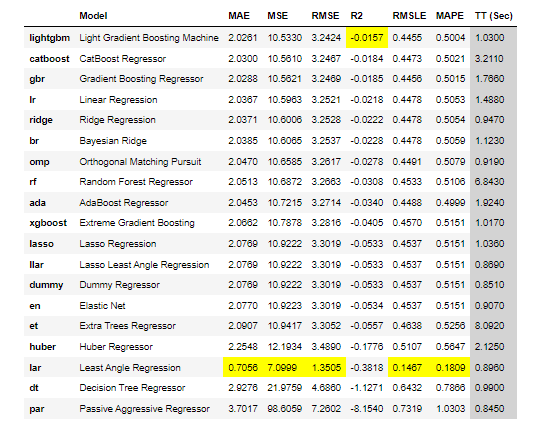
valid set 그래프 확인
plot_model(best,plot = 'learning')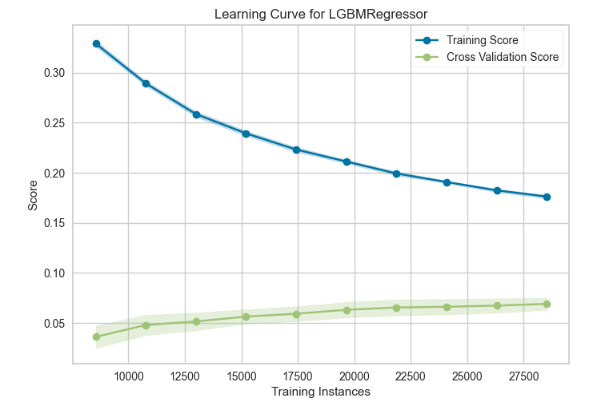
모델 생성 및 튜닝
model_RMSLE = tune_model(create_model('lightgbm'),optimize = 'RMSLE')
model_RMSLE 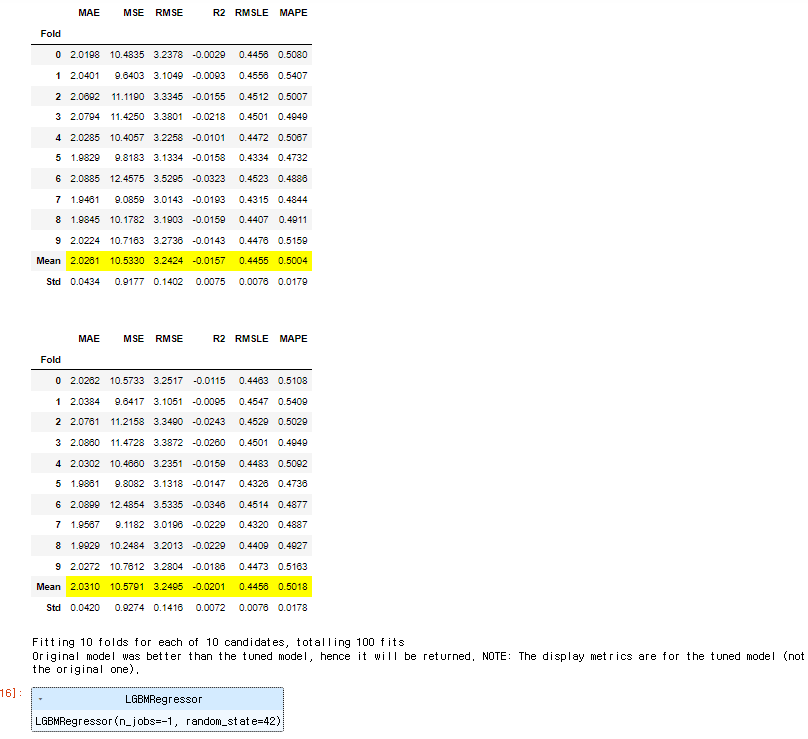
valid set 예측 결과
predict_model(model_RMSLE)
최종 모델 훈련
final_model = finalize_model(model_RMSLE)
final_model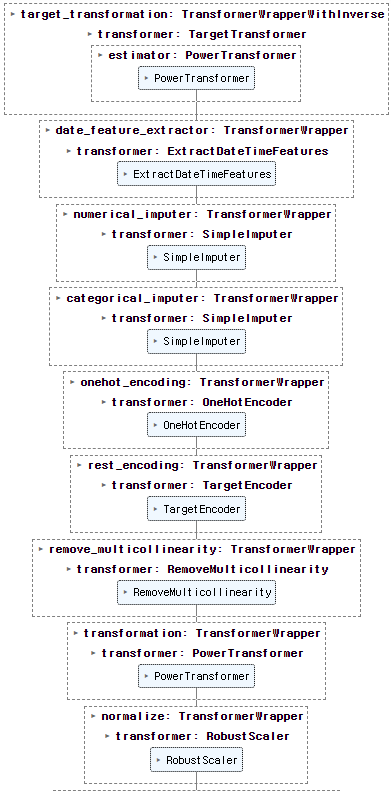
예측
predictions=predict_model(final_model,data = test_df[target_column[:-1]])
predictions.head()모델 저장
save_model(final_model, 'automl_final_model')평가 결과
-
lightgbm
- public 0.4399376221
- private 0.440819985
-
gradient boost
- public 0.4274680774
- private 0.4274374129
02. 공식문서로 함수 알아보기
- 공식문서 - QuickStart Regression
순서
- Setup - Compare Models - Analyze Model - Predictions - SaveModel
1) Setup
- 훈련 환경을 초기화하고 transformation pipeline을 생성하는 함수
- 다른 함수들을 실행하기 전에 반드시 실행되어야 함
- required : data, target
- optional : others
예를 들어MLFlow 환경 설정 같은 것들
- 다른 함수들을 실행하기 전에 반드시 실행되어야 함
예시
from pycaret.classification import *
clf1 = setup(data, target = 'Class variable', log_experiment = True, experiment_name = 'diabetes1')2 ) Compare Models
예시
- 다양한 옵션을 사용하여 모델을 비교할 수 있음
- 특정 모델만 비교, 특정 모델 제외 비교
- 베스트 n개 모델 선택
- 한정된 시간 내에 수행되는 모델만 비교
- Probability 임계점 설정
- Cross Validation 끄기 설정
# load dataset
from pycaret.datasets import get_data
diabetes = get_data('diabetes')
# init setup
from pycaret.classification import *
clf1 = setup(data = diabetes, target = 'Class variable')
# compare models
best = compare_models(include = ['lr', 'dt', 'lightgbm'])
best = compare_models(exclude = ['lr', 'dt', 'lightgbm'])
best = compare_models(n_select = 3)
best = compare_models(budget_time = 0.5)
best = compare_models(probability_threshold = 0.25)
best = compare_models(cross_validation=False)- 대규모 데이터셋으로 확장해서 사용할 수도 있음
로컬 Spark로 테스트 하려면 setup에서 n_jobs = 1로 설정해야 함
- 일부 모델은 이미 사용가능한 모든 코어를 사용하려고 시도할 수 있음
- 이런 모델을 병렬 실행하면 교착 상태가 발생할 수 있음
# load dataset
from pycaret.datasets import get_data
diabetes = get_data('diabetes')
# init setup
from pycaret.classification import *
clf1 = setup(data = diabetes, target = 'Class variable', n_jobs = 1)
# create pyspark session
from pyspark.sql import SparkSession
spark = SparkSession.builder.getOrCreate()
# import parallel back-end
from pycaret.parallel import FugueBackend
# compare models
best = compare_models(parallel = FugueBackend(spark))3 ) create_model
- 다양한 옵션을 사용하여 모델을 생성할 수 있음
- K-fold 횟수 지정
- custom parameter 전달
- 임계점 설정
등등
# load dataset
from pycaret.datasets import get_data
diabetes = get_data('diabetes')
# init setup
from pycaret.classification import *
clf1 = setup(data = diabetes, target = 'Class variable')
# train logistic regression
lr = create_model('lr')
lr = create_model('lr', fold = 5)
# train decision tree
dt = create_model('dt', max_depth = 5)
# access the scoring grid
dt_results = pull()
print(dt_results)
# train model without cv
lr = create_model('lr', cross_validation = False)
# train model without cv
lr = create_model('lr', return_train_score = True)
# train model with 0.25 threshold
lr = create_model('lr', probability_threshold = 0.25)
# train models in a loop
lgbs = [create_model('lightgbm', learning_rate = i) for i in np.arange(0.1,1,0.1)]참고 - 사용가능한 모델 확인
models()
custom model 추가하기
# load dataset
from pycaret.datasets import get_data
insurance= get_data('insurance')
# init setup
from pycaret.regression import *
reg1 = setup(data = insurance, target = 'charges')
# create custom estimator
import numpy as np
from sklearn.base import BaseEstimator
class MyOwnModel(BaseEstimator):
def __init__(self):
self.mean = 0
def fit(self, X, y):
self.mean = y.mean()
return self
def predict(self, X):
return np.array(X.shape[0]*[self.mean])
# create an instance
my_own_model = MyOwnModel()
# train model
my_model_trained = create_model(my_own_model)4 ) Optimize Model
a ) tune model
- 모델의 hyper parameter를 비교하여 fold 별 cross validation 성능을 반환
- 반복횟수 지정
n_iter = 50 - 최적화할 metric 지정
optimize = 'MAE' - 성능이 더 나은 모델 반환
choose_better = True- 때로는 hyper parameter 를 탐색한 결과가 더 나쁠 수 있음
- 이 때는 input model을 반환할 수 있도록 설정
- 반복횟수 지정
# load dataset
from pycaret.datasets import get_data
boston = get_data('boston')
# init setup
from pycaret.regression import *
reg1 = setup(data = boston, target = 'medv')
# train model
dt = create_model('dt')
# tune model
tuned_dt = tune_model(dt, n_iter = 50)
tuned_dt = tune_model(dt, optimize = 'MAE')
tuned_dt = tune_model(dt, choose_better = True)grid search할 범위를 전달할수도 있음
# define search space
params = {"max_depth": np.random.randint(1, (len(boston.columns)*.85),20),
"max_features": np.random.randint(1, len(boston.columns),20),
"min_samples_leaf": [2,3,4,5,6]}
# tune model
tuned_dt = tune_model(dt, custom_grid = params)search algorithm 변경도 가능
- default : RandomGridSearch
tune_model(dt) # default : RandomGridSearch
# tune model optuna
tune_model(dt, search_library = 'optuna')
# tune model scikit-optimize
tune_model(dt, search_library = 'scikit-optimize')
# tune model tune-sklearn
tune_model(dt, search_library = 'tune-sklearn', search_algorithm = 'hyperopt')tuning model 에 접근할 수 있음
# load dataset
from pycaret.datasets importh get_data
boston = get_data('boston')
# init setup
from pycaret.regression import *
reg1 = setup(boston, target = 'medv')
# train model
dt = create_model('dt')
# tune model and return tuner
tuned_model, tuner = tune_model(dt, return_tuner=True)이외에도
ensemble_model,blend_models,stack_models,optimize_threshold,calibrate_model같은 함수를 제공하므로 공식 문서 확인하기
5 ) Analys Model
a ) plot_model
- hod-out set에 대한 훈련된 모델의 성능을 시각화
- scale을 변경하거나
- plot을 저장할 수 있음
- Yellowbrick을 활용하므로 해당 인자로 custom 할 수 있음
- train data에 대한 시각화도 가능
- 다양한 시각화가 가능하므로 문서 확인하기
# load dataset
from pycaret.datasets import get_data
diabetes = get_data('diabetes')
# init setup
from pycaret.classification import *
clf1 = setup(data = diabetes, target = 'Class variable')
# creating a model
lr = create_model('lr')
# plot model
plot_model(lr, plot = 'auc')
plot_model(lr, plot = 'auc', scale = 3)
plot_model(lr, plot = 'auc', save = True)
plot_model(lr, plot = 'confusion_matrix', plot_kwargs = {'percent' : True})
plot_model(lr, plot = 'auc', use_train_data = True)이외에도 다양한 함수를 제공하므로 공식 문서 확인하기
6 ) Predict Model
a ) predict_model
a ) finalize_model
a ) save_model
이외에도 다양한 함수를 제공하므로 공식 문서 확인하기
