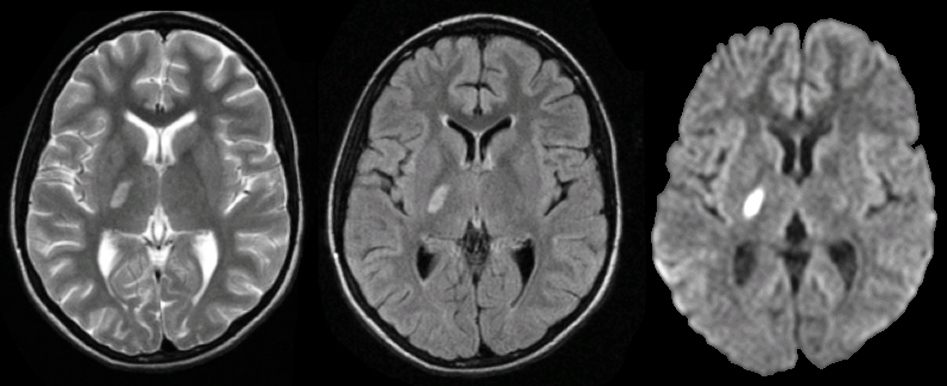
nnunet
nnunet 논문 링크
nnunet 논문 업데이트 버전 링크
nnunet은 제공된 데이터셋에 자동으로 적응하는 semantic segmentation 방법이다. U-Net 기반 segmentation pipeline이다.
semantic segmentation이 무엇인가?
=> 컴퓨터 비전분야에서 사용되는 기술로, 이미지에서 각 픽셀을 해당하는 객체 또는 클래스로 분할하는 작업을 말한다.
NIHSS 정의
National Institute of health stroke scale의 줄임말
stroke로 인한 신경학적 장애를 평가하는 임상 척도이며, 총 11개의 문항에 대한 답변을 기반으로 평가하며 0점~42점의 결과를 볼 수 있다. 점수가 높을수록 중증이다.
NIHSS score는 병변의 위치, 크기에만 영향을 받는것이 아니라 다양한 요인에 영향을 받으므로 단순히 병변의 크기만으로 NIHSS score를 판단하기는 어렵다.
Dataset
현재 우리의 데이터셋은 총
2019년 1월 1일부터 2021년 12월 31일까지 본원 응급실을 통해 신경과로 입원한 환자들 중에 뇌경색 진단명으로 등록된 환자, 18세 이상 90세 이하의 환자
=> 3년간 총 273명의 환자가 분류되었음
한 환자당 dataset은 이미지는 ADC,B0,B1000 nii.gz 영상으로 이루어져있고, lesion 영상도 있음
- 데이터 셋의 문제점
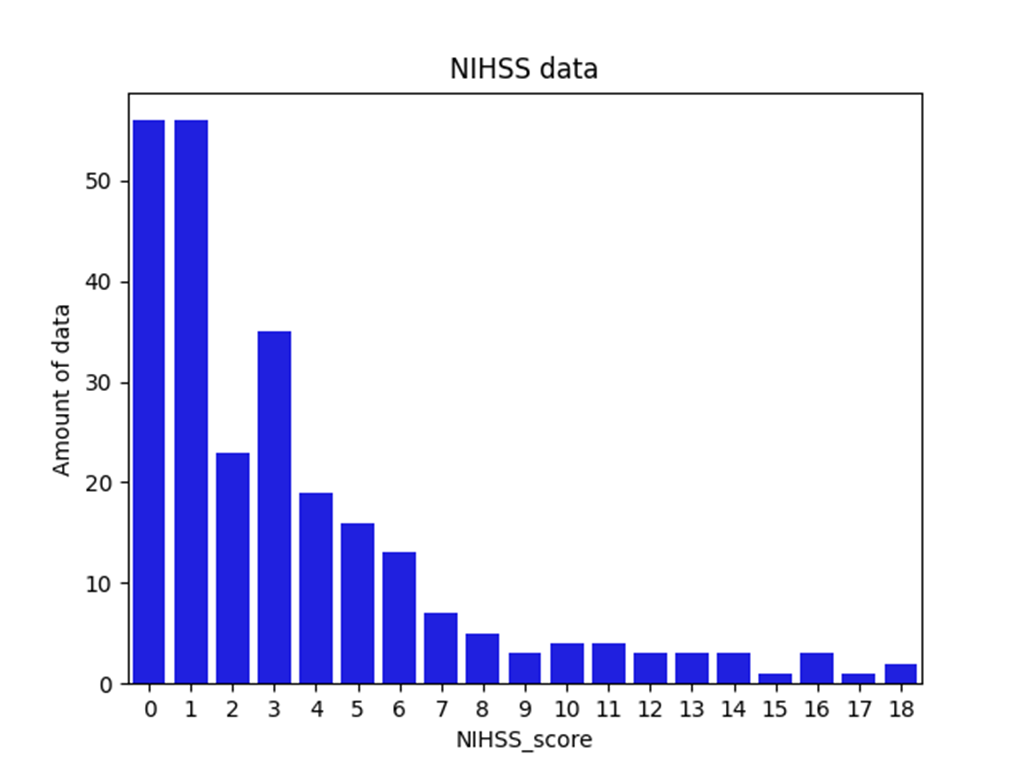
데이터 셋의 nihss score가 대부분 0~2에 분포되어 있는 class imbalance 문제가 있음
=> 5 fold cross validation 이용
학습
- framework
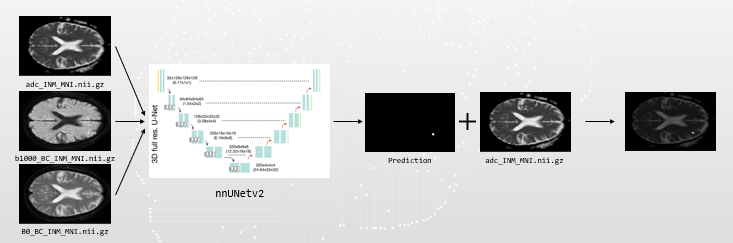
=> segmentation 결과
작은 lesion의 디테일 적인 부분을 전체적으로 예측하지 못함
(Lesion의 크기가 작으면 디테일 적인 부분이 떨어져 예측 값이 낮음)
Lesion의 모양이 매끈한 타원 모양이거나 크기가 크면 prediction이 잘 되는 경향을 보임

-
2d regression
train-> lesion이 제일 많은 depth 2개의 image를 추출하여 multi input으로 사용함

-
3d regression
train - lesion영상과 nii.gz 파일과 merge해서 사용
test - nnunetv2로 예측한 파일과 adc.nii.gz를 merge해서 사용
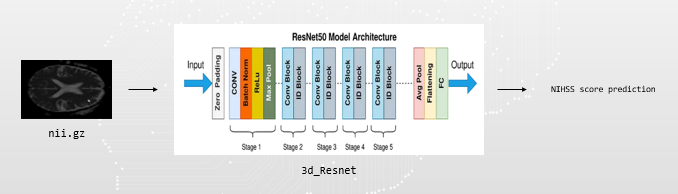
결과=> score가 큰 값을 예측할때 크게 벗어나는 경우가 있음
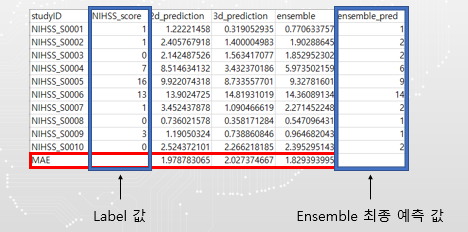
Ensemble
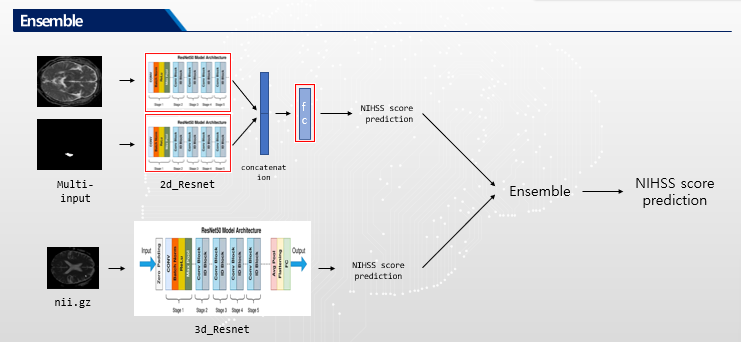
결과
Baseline
lesion1_MIN.nii.gz의 mask image를 ground truth로 사용
Epoch = 150
Fold 0~4 총 5번 학습을 함. (한 fold 당 대략 7시간이 걸림 총 35시간 정도 학습 진행)
Dataset의 마지막 10개를 제외한 NIHSS_S0001~263번을 학습하였으며 lesion이 없는 158, 212, 214, 226, 250, 258는 제외해 학습을 진행함
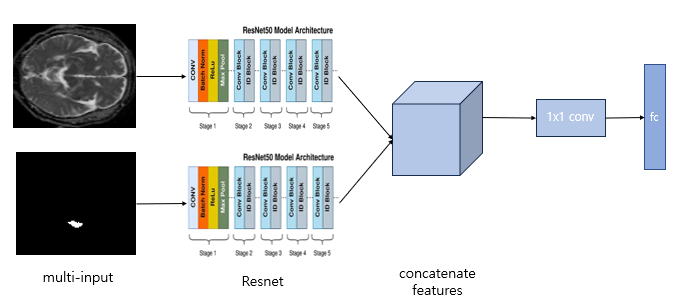
수정한 방식
기존 방식: brain image와 lesion image를 fc layer에 넣기 직전에 합쳐서 넣음.
수정한 방식: 공간적인 정보를 고려하기 위해 flatten 전에 두 이미지의 feature를 합치고 1x1conv 1개를 적용함
Transformer 기반의 모델 사용(ViT, DeiT)
Data augmentation 적용함
Epoch = 200
Batch size= 32
Image size = (182, 218)
Optimizer = SGD
Number of total data = 264
5-fold cross validation
성능: 전체 mae 사용
Transformer
Huggingface에 있는 모델과 transformers 라이브러리를 활용하여 image classification용 ViT와 DeiT를 regression 문제로 바꾸어 적용.
(image classification으로 풀면 성능이 안 좋았음)
ViT는 기본 모델인 “google/vit-base-patch16-224”를 사용.
DeiT는 MRI dataset으로 fine-tune한 “raedinkhaled/deit-base-mri”를 사용.
입력으로는 brain image와 lesion image를 각각 따로 넣어서 실험.
Epoch = 100
Batch size= 32
Image size = (224, 224)
Optimizer = Adamw
Number of total data = 264
5-fold cross validation
-
baseline

-
deit raw brain image
-
deit lesion image

-
vit raw brain image
-
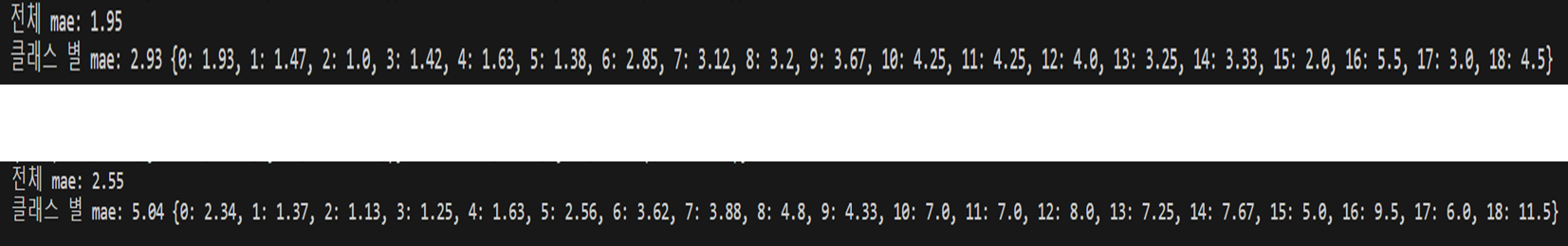
vit lesion image

=> 결과:
lesion image만 사용한 경우, ViT와 DeiT 모두 baseline을 뛰어넘는 성능을 보임.(baseline은 brain, lesion image 모두 사용)
lesion image의 경우 DeiT가 ViT 보다 성능이 좋지만, brain image만 사용하는 경우 오히려 ViT가 조금 더 좋음. (baseline 보단 낮음)
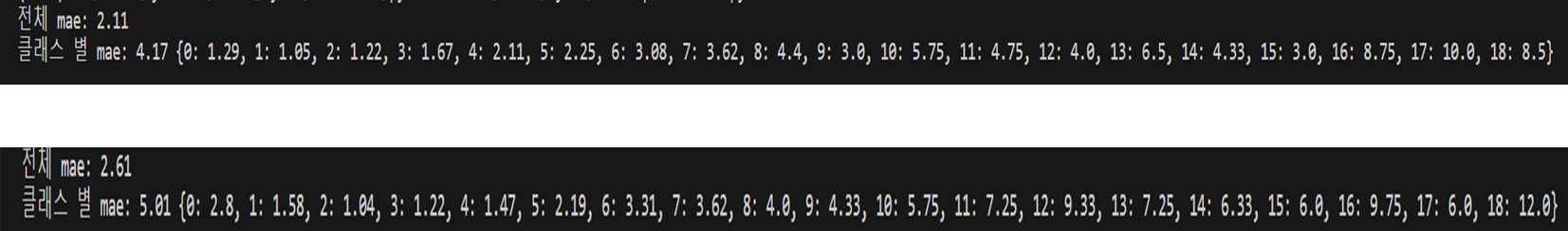
Data augmentation
일반적인 augmentation 방법(crop, flip, rotate)는 학습 데이터의 quality가 떨어질 수 있음
그래서 샘플수가 적은 score들에 대해서 환자의 3d data에 lesion이 있는 다른 layer들을 사용하였음
=> 전체 mae는 올라갔고 클래스별 mae는 떨어짐
결과
작은 lesion, 높은 score 예측 어렵
