Notebook
-
transcription
3250feats->532 feats using shap[LB: 0.436].ipynb
plt.style.use('seaborn')
- 현재 이런 방식으로 등록하는 것에 문제가 있어, 우선 사용하지 않는 것으로 코드 수정
import featuretools as ft
-
구글 코랩에서 기본 제공해주는 라이브러리가 아닌 문제
-
설치하여 사용
!pip install dask[dataframe] featuretools결측치 처리
edjefa와edjefe처리 &edjef특성 생성 & 데이터 불일치 수정 과정에서 TypeErrer 발생df_train[['edjefa', 'edjefe']]가 숫자형이 아닌 str형이어서 생긴 문제
- 숫자형으로 변경하여 해결
# edjefa와 edjefe를 숫자로 변환
df_train['edjefa'] = pd.to_numeric(df_train['edjefa'], errors='coerce')
df_train['edjefe'] = pd.to_numeric(df_train['edjefe'], errors='coerce')
df_test['edjefa'] = pd.to_numeric(df_test['edjefa'], errors='coerce')
df_test['edjefe'] = pd.to_numeric(df_test['edjefe'], errors='coerce')결측치 처리 후, missing value 존재 문제
-
결측치 처리를 동일하게 진행했으나, 여전히 결측치가 존재하는 문제 발생
-
위의 결측치 처리 전 단계에서 str을 int형으로 변경한 것에 영향이 있을 것으로 추측.
-
우선, 별도 조치를 하지 않고 그대로 진행
-
기존 노트북
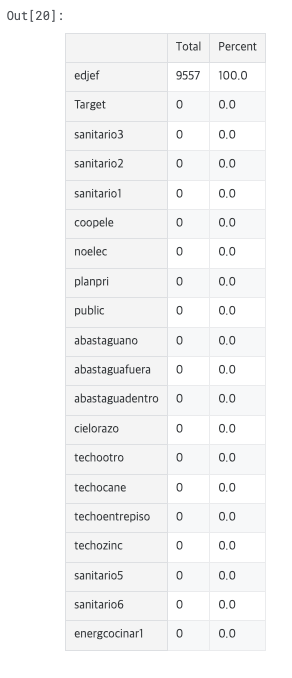
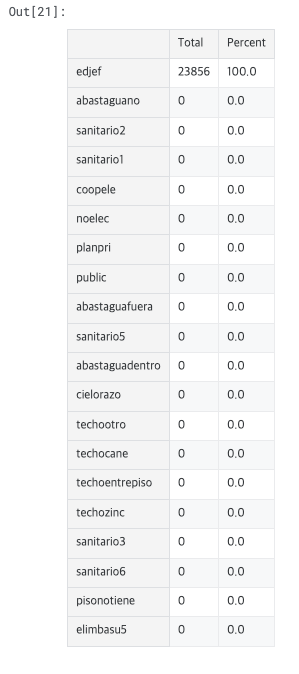
-
내 노트북

object형 피쳐가 다름
- 기존 코드에는 'edjefe', 'edjefa'도 있지만, 이전 단계에서 두 컬럼을 이미 수치형으로 바꿔줬기 때문에 다르게 나옴.
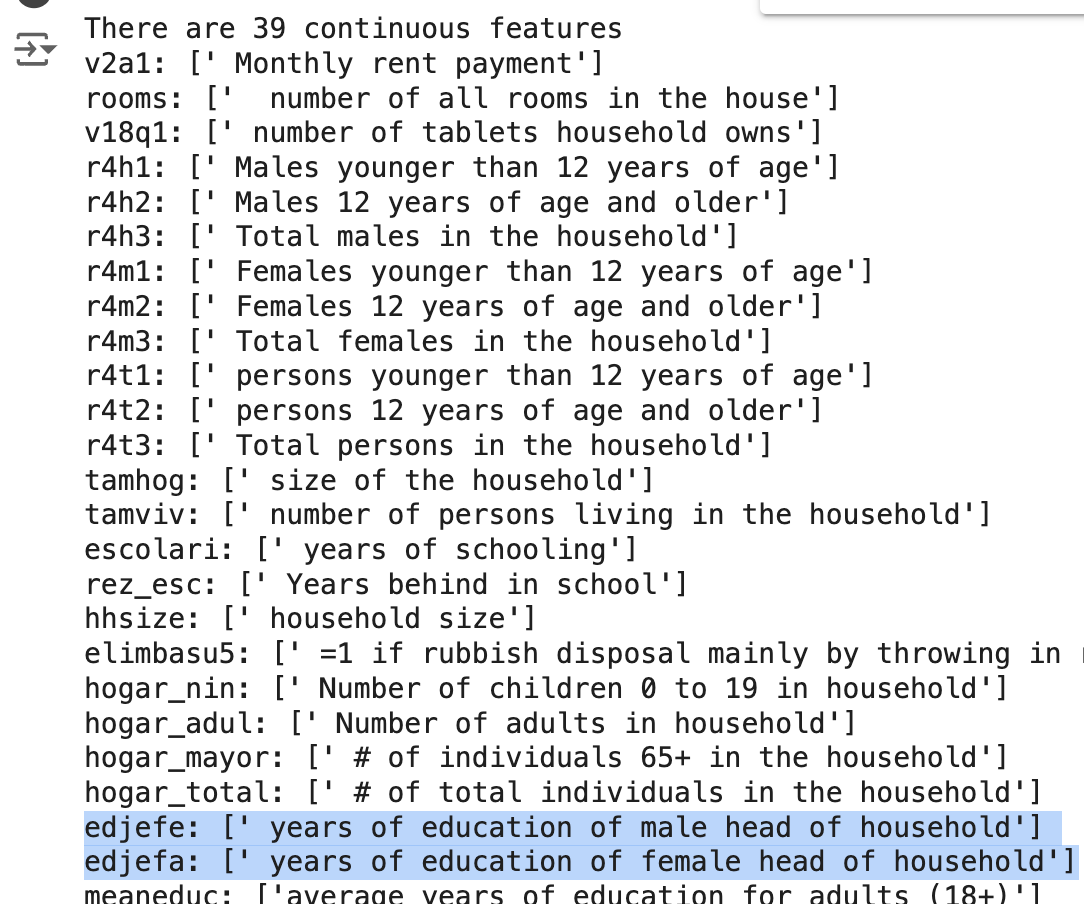
Make new features using continuous feature가 37개가 아닌 39개가 나옴
-
개수가 다르게 나오는 문제

-
이것 역시, 전처리 과정에서 다르게 처리했기 때문에 생기는 자연스러운 현상으로 파악
wall, roof, floor may be key factor
- wall, roof, floor : 중요 요소
- 이 특성들을 곱하여 새 특성 생성
- 이진 범주형이니 -> 곱하면 0, 1, 2, 3 등 새로운 범주형 값이 나오니까 이걸 활용하려는 것!
이진 범주형 데이터들을 곱해서 더 중요도가 높은 새 특성을 만들 수 있다는 것을 학습
특성을 무조건 곱하면?
- 하나라도 0이 있으면, 100개 이상을 가지고 있어도 값이 0이 될텐데, 이 부분은 의문

연결된 문제
TypeError: LGBMClassifier.fit() got an unexpected keyword argument 'early_stopping_rounds'
- 방법 1
from lightgbm import early_stopping설정- 해당 에러 위 코드의
clf.fit파트에서 -> 콜백 함수로 지정
- 해결하고 나니 다른 문제 발생
TypeError: LGBMClassifier.fit() got an unexpected keyword argument 'verbose'-
방법 2
- 해당 에러 위 코드의
clf.fit파트에서 -> verbose를 verbose_eval로 변경 - 여전히 해결되지 않아 다음 방법 수행 -> 실패
callbacks=[lgb.early_stopping(stopping_rounds=500), lgb.log_evaluation(period=500)], eval_train_metric = 'auc') - 해당 에러 위 코드의
-
방법 3
- 현재의 LightBGM에서는 사용되지 않는 함수들이기 때문에
eval_train_metric = 'auc'는 삭제하고 진행
- 현재의 LightBGM에서는 사용되지 않는 함수들이기 때문에
ValueError: Length of values (4) does not match length of index (3249)
-
shap_values를 DataFrame에 할당할 때 그 길이가 일치하지 않아서 발생
- shap_values는 (num_samples, num_classes, num_features)의 3차원 배열
- 클래스 차원에서 평균화
-
기존 코드
# SHAP 값 계산
shap_values = shap.TreeExplainer(clf.booster_).shap_values(X_train)
fold_importance_df = pd.DataFrame()
fold_importance_df['feature'] = X_train.columns
fold_importance_df['shap_values'] = abs(np.array(shap_values)[:, :].mean(1).mean(0))
fold_importance_df['feat_imp'] = clf.feature_importances_- 수정한 코드
# SHAP 값 계산
shap_values = shap.TreeExplainer(clf.booster_).shap_values(X_train)
# 다중 클래스의 경우 클래스 차원에서 SHAP 값을 평균화
shap_values_mean = np.mean(np.abs(shap_values), axis=0) # 클래스 차원에서 평균 (axis=0)
# 특성 중요도 계산
fold_importance_df = pd.DataFrame()
fold_importance_df['feature'] = X_train.columns
fold_importance_df['shap_values'] = shap_values_mean.mean(axis=0) # 샘플 차원에서 평균
fold_importance_df['feat_imp'] = clf.feature_importances_ValueError: Expected a 1D array, got an array with shape (3249, 4)
- 학습 파트 코드 수정
import numpy as np
import pandas as pd
import shap
import lightgbm as lgb
from sklearn.model_selection import StratifiedKFold
import time
import warnings
warnings.filterwarnings(action='ignore')
def extract_good_features_using_shap_LGB(params, SEED):
clf = lgb.LGBMClassifier(objective='multiclass',
random_state=1989,
max_depth=params['max_depth'],
learning_rate=params['learning_rate'],
silent=True,
metric='multi_logloss',
n_jobs=-1, n_estimators=10000,
class_weight='balanced',
colsample_bytree = params['colsample_bytree'],
min_split_gain= params['min_split_gain'],
bagging_freq = params['bagging_freq'],
min_child_weight=params['min_child_weight'],
num_leaves = params['num_leaves'],
subsample = params['subsample'],
reg_alpha= params['reg_alpha'],
reg_lambda= params['reg_lambda'],
num_class=len(np.unique(y)),
bagging_seed=SEED,
seed=SEED,
)
kfold = 5
kf = StratifiedKFold(n_splits=kfold, shuffle=True)
feat_importance_df = pd.DataFrame()
for i, (train_index, test_index) in enumerate(kf.split(train, y)):
print('='*30, '{} of {} folds'.format(i+1, kfold), '='*30)
start = time.time()
X_train, X_val = train.iloc[train_index], train.iloc[test_index]
y_train, y_val = y.iloc[train_index], y.iloc[test_index]
# callbacks 설정
callbacks = [
lgb.early_stopping(stopping_rounds=500), # early stopping
lgb.log_evaluation(period=100) # eval_metric 출력
]
# 모델 학습 (verbose는 제거)
clf.fit(X_train, y_train, eval_set=[(X_train, y_train), (X_val, y_val)],
eval_metric='multi_logloss', # 평가 지표 설정
categorical_feature=categorical_feats,
callbacks=callbacks) # 콜백 함수 설정
# SHAP 값 계산
shap_values = shap.TreeExplainer(clf.booster_).shap_values(X_train)
# 다중 클래스 문제에서 shap_values는 리스트로 반환됨
if isinstance(shap_values, list): # 다중 클래스 문제일 경우
# 각 클래스별 SHAP 값의 평균을 계산 (각 샘플에 대해 모든 클래스의 SHAP 값 평균)
shap_values_mean = np.mean(np.abs(np.array(shap_values)), axis=0) # 각 클래스에 대해 평균
else: # 이진 분류일 경우
shap_values_mean = np.abs(shap_values)
# 특성 중요도 계산
fold_importance_df = pd.DataFrame()
fold_importance_df['feature'] = X_train.columns
fold_importance_df['shap_values'] = shap_values_mean.mean(axis=0) # 샘플 차원에서 평균
fold_importance_df['feat_imp'] = clf.feature_importances_
# 모든 fold에서의 중요도 합치기
feat_importance_df = pd.concat([feat_importance_df, fold_importance_df])
# 실행 시간 출력
print_execution_time(start)
# SHAP 값 기반 특성 중요도 계산
feat_importance_df_shap = feat_importance_df.groupby('feature').mean().sort_values('shap_values', ascending=False).reset_index()
return feat_importance_df_shap🚨 해결되지 않아, 이 코드 다음의 코드부터는 실행하지 않음.
- 추후 해결 예정
