
이전 글에 이어서 작성된 게시물입니다.
이전 글 링크: NetCDF 의 특징과 라이브러리 활용 (with python)
목차
- 개발환경 세팅하기
- 예시 데이터 준비
- NetCDF 생성
- GeoTiff 파일 분석
- GeoTiff 파일로 NetCDF 생성
- CSV 파일로 NetCDF 생성
개발환경 세팅하기
저는 이 글에서 저의 개발환경 상에서 모든 작업을 합니다.
해당 개발환경 설정법은 docker 가 있다면 아주 쉽게 할 수 있습니다.
개발환경 설정법은 제가 이전에 작성한 게시물을 참고하시기 바랍니다.
예시 데이터 준비
💾 파일 다운로드
환격공간정보서비스 라는 사이트에 접속하고 로그인을 합니다.
(계정이 없다면 계정을 생성해주세요)
로그인이 완료된 후에 자료를 신청합니다.
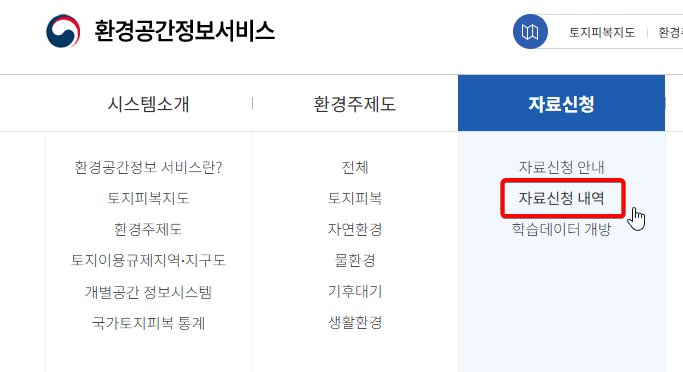
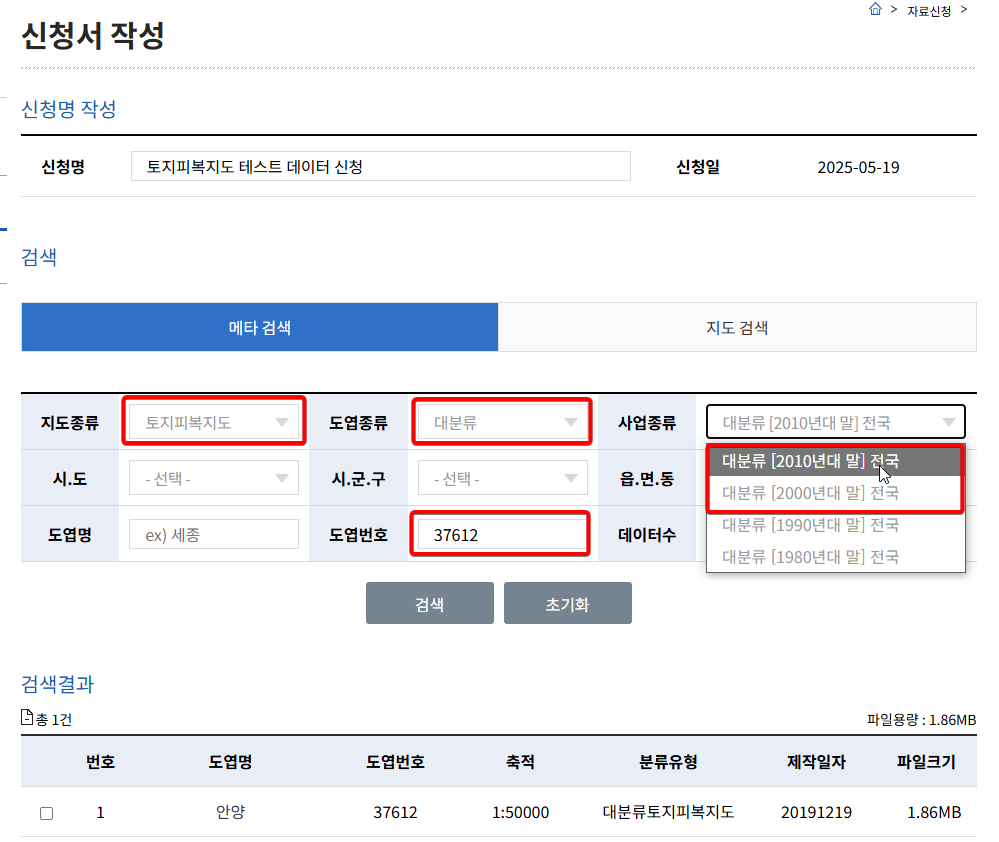
신청서에 보면 여러가지 검색 조건이 나옵니다.
검색조건을 다음과 같이 입력합니다.
- 지도 종류: 토지피복지도
- 도엽 종류: 대분류
- 도엽번호: 37612
이러면 안양의 토지피복도가 검색되는데,
사업종류 검색조건을 대분류 [2010년대 말] 전국, 대분류 [2000년대 말] 전국 2가지로 해서
2개의 토지피복도를 신청합니다.
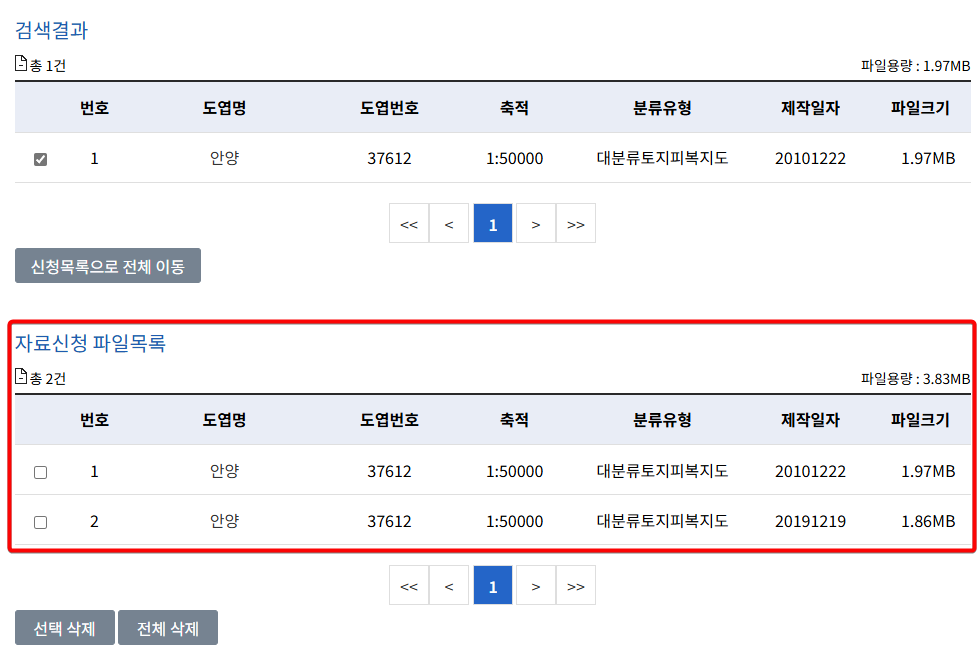
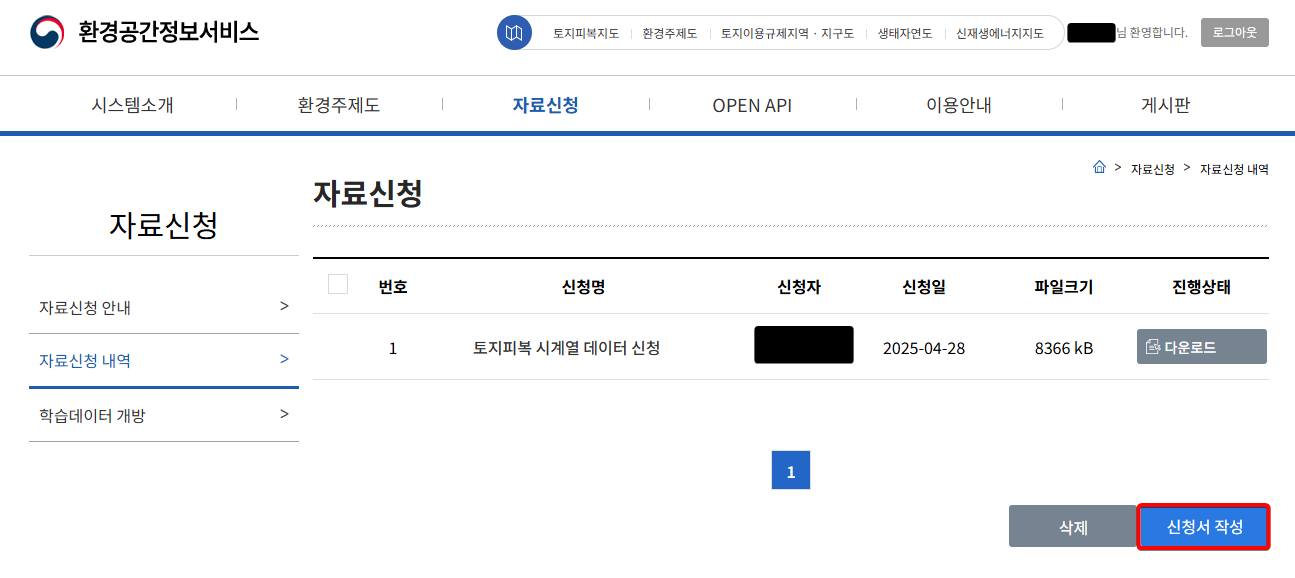
이후에 다시 자료신청 내역 메뉴에 들어가면
아래와 같이 목록이 하나 생길 겁니다.
다운로드 버튼을 클릭해서 다운로드 받으세요.
(참고로 무슨 파일 다운로드 프로그램(Innorix)을 설치하라고 나오는데,
그냥 설치하고 나서 신청한 파일을 다운로드 받아주세요)
이러면 2개의 파일을 다운로드 받게됩니다.(아래 그림참고)
각각의 zip 파일에는 다시 아래와 같이 geotiff 파일이 있습니다.
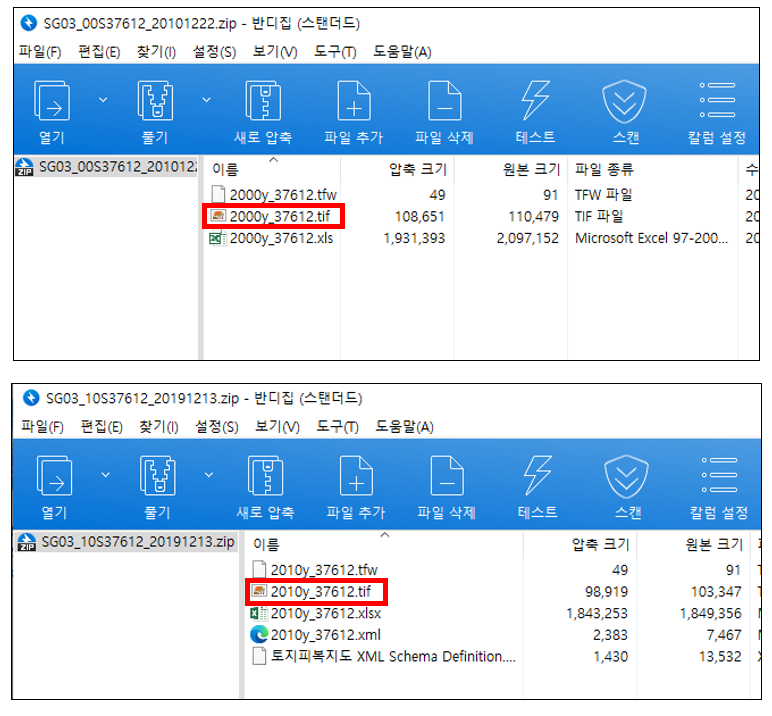
일단 저 2개의 geotiff 파일만 쏙 빼내서 다른 디렉토리에 잘 저장해둡시다.
참고로 해당 tif 파일을 QGIS 에 올려보면 다음과 같이 생겼습니다.
⚙ Geotiff 에서 CSV 추출하기
이 글에서는 geotiff 뿐만 아니라 CSV 로도 Netcdf 를 만들 것이기 때문에
앞서 다운로드 받은 geotiff 파일에서 csv 데이터를 미리 추출해놓겠습니다.
cli 환경에서 gdal 명령어를 통해서 아래와 같이 명령어를 입력하여
각 tif 파일에서 csv 를 추출합니다.
# 리눅스 환경에서 돌린 명령어들입니다.
gdal_translate -of XYZ 2000y_37612.tif 2000y_37612_tmp.csv
tr ' ' ',' < 2000y_37612_tmp.csv > 2000y_37612.csv
rm 2000y_37612_tmp.csv
gdal_translate -of XYZ 2010y_37612.tif 2010y_37612_tmp.csv
tr ' ' ',' < 2010y_37612_tmp.csv > 2010y_37612.csv
rm 2010y_37612_tmp.csv
이렇게 추출하고 나서 csv 파일을 까보면 다음과 같습니다.
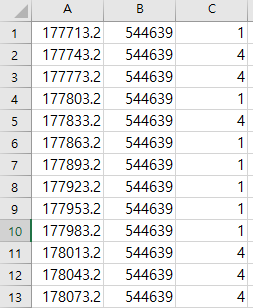
A 열은 지도 상의 X 축을 의미하고,
B 열은 지도 상의 Y 축을 의미합니다.
C 열은 각 X, Y 축에 위치한 cell 이 갖는 값을 의미합니다.
X, Y 축의 값은 row 가 내려감에 따라 작은 값에서 큰값(오름차순) 으로 바뀝니다.
참고로 추출된
X,Y열의 값은 모두GeoTiff의 각pixel의
중앙 위치(좌표) 값입니다. 각pixel의Top-Left꼭지점이 아닙니다!
QGIS의 배경지도CRS를EPSG:5186으로 맞춘 후에
Geotiff가장 좌측 꼭대기에 있는 픽셀의 중앙을 클릭해서 이를 확인할 수 있습니다.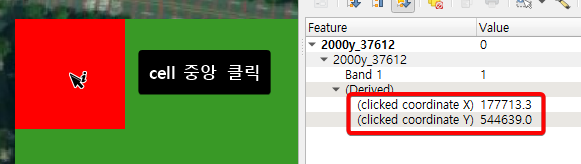
여기까지 해서 일단 샘플 데이터 준비는 끝났습니다. (고생하셨습니다!)
이제 이 GeoTiff (또는 CSV) 를 NetCDF 로 변환시켜 봅시다.
NetCDF 생성
먼저 필요한 python 라이브러리와 리눅스 패키지를 다운로드 받습니다.
# 파이썬 라이브러리 다운로드
pip install netCDF4 # netCDF 조작 라이브러리
pip install numpy # 복잡한 배열을 제어하기 위한 라이브러리
pip install rasterio # 이건 geotiff 분석용 라이브러리
pip install pandas # csv 조회를 위한 라이브러리
# 패키지 다운로드, ncdump 라는 명령어를 사용하기 위함입니다!
apt install -y netcdf-bin자~ 이제부터 본격적으로 시작해보겠습니다.
GeoTiff 파일 분석
GeoTiff 로 NetCDF 를 만들기 위해서는 일단 GeoTiff 파일의 메타정보를
읽는 것에서부터 시작합니다. 그러니 GeoTiff 파일을 분석하는 법부터 알아보죠.
아래처럼 코드를 작성해서 콘솔의 결과를 한번 스스로 관찰해봅시다.
의외로 중요한 정보들을 많이 뽑아낼 수 있습니다.
코드 :
import rasterio
import numpy as np
base_path = r'/bind_dir/netcdf_sample_inputs'
with rasterio.open(base_path + "/2000y_37612.tif", ) as src:
# width, height = src.width, src.height
# transform = src.transform
# xres, yres = src.res
print(f"pixel 갯수 (가로 x 세로) : [ {src.width} x {src.height} ]")
print(f"\npixel 갯수 (세로, 가로) : [ {src.shape} ]")
print(f"\n한 픽셀의 크기 (x, y) : {src.res}")
print(f"\n한 픽셀당 x 축 좌표 변화량 : {src.transform.a}")
print(f"\n한 픽셀당 y 축 좌표 변화량 : {src.transform.e}")
print(f"\n가장 좌측, 상단 픽셀의 TOP-LEFT 꼭지점의 x 값 : {src.transform.c}")
print(f"\n가장 좌측, 상단 픽셀의 TOP-LEFT 꼭지점의 y 값 : {src.transform.f}")
print("\nBounding Box :", src.bounds)
print("\n현재 GeoTiff 파일의 Band 수 : ", src.count)
data = src.read(1) # 가장 첫번째 band 를 읽습니다. 현재는 band 가 하나 밖에 없어서 1 만 가능합니다.
# read 메소드는 첫번째 요소를 조회하기 위해서 0 이 아닌 1 을 줘야합니다! 주의!
print("\n데이터(=2차원 배열, (y,x)) :", data) # (x,y) 가 아니라 (y,x) 라는 점 유의하세요!
print("\n밴드의 값 타입 :", data.dtype)
# print("\n밴드의 값 타입 :", src.dtypes[0]) # 이렇게도 조회할 수 있습니다
print("\n데이터가 없는 위치의 default 값: ", src.nodata)
# print("\n데이터가 없는 위치의 default 값: ", src.nodatavals[0]) # 이렇게도 조회할 수 있습니다
print("\nGeotiff 의 CRS => WTK 정보 :", src.crs.to_wkt())
print("\nGeotiff 의 CRS => EPSG CODE :", src.crs.to_epsg()) # None 일 수 있습니다!
print("\nGeotiff 의 CRS => proj4 :", src.crs.to_proj4())
print("\n\n======= Geotiff 의 전반적인 메타 정보 =======\n", src.meta)출력 결과 :
pixel 갯수 (가로 x 세로) : [ 747 x 934 ]
pixel 갯수 (세로, 가로) : [ (934, 747) ]
한 픽셀의 크기 (x, y) : (29.99999999999996, 30.0)
한 픽셀당 x 축 좌표 변화량 : 29.99999999999996
한 픽셀당 y 축 좌표 변화량 : -30.0
가장 좌측, 상단 픽셀의 TOP-LEFT 꼭지점의 x 값 : 177698.2205497106
가장 좌측, 상단 픽셀의 TOP-LEFT 꼭지점의 y 값 : 544654.0188778294
Bounding Box :
BoundingBox(
left=177698.2205497106,
bottom=516634.0188778294,
right=200108.22054971056,
top=544654.0188778294)
현재 GeoTiff 파일의 Band 수 : 1
데이터(=2차원 배열, (y,x)) :
[[1 4 4 ... 1 1 1]
[4 4 4 ... 1 1 1]
[4 4 4 ... 1 1 3]
...
[2 2 2 ... 4 4 4]
[2 2 2 ... 4 4 4]
[2 2 2 ... 4 4 4]]
밴드의 값 타입 : uint8
데이터가 없는 위치의 default 값: None
Geotiff 의 CRS => WTK 정보 :
PROJCS["Korea_2000_Korea_Central_Belt_2010",GEOGCS... <너무 길어서 생략> ]
Geotiff 의 CRS => EPSG CODE : None
Geotiff 의 CRS => proj4 :
+proj=tmerc +lat_0=38 +lon_0=127 +k=1 +x_0=200000 +y_0=600000 +ellps=GRS80 +units=m +no_defs=True
======= Geotiff 의 전반적인 메타 정보 =======
{
'driver': 'GTiff',
'dtype': 'uint8',
'nodata': None,
'width': 747,
'height': 934,
'count': 1,
'crs': ... <너무 길어서 생략>
}한 픽셀당 x 축 좌표 변화량,한 픽셀당 y 축 좌표 변화량라는 용어가 헷갈리죠?
이건 geotiff pixel 들이 정렬된 방향(+/-)와 각 픽셀의 크기를 곱한 값이라고 생각하면 됩니다.
GeoTiff 파일로 NetCDF 생성
GeoTiff 는 모든 pixel 들을 2차원 배열의 형태의 데이터로 담고 있습니다.
이러한 2차원 배열의 데이터를 NetCDF 의 변수(Variable) 로 넣을 생각입니다.
variable 의 dimension 은 (time, y, x) 로 지정할 예정이며,
(y,x) 는 geotiff 에서 뽑아내고,
time 은 각 geotiff 파일들의 기준연도(2010, 2000)를 기반으로 만들 겁니다.
그래서 파일 하나를 사용할 때는 그 time dimension 의 길이가 1 이고,
2 개를 넣을 때는 길이가 2입니다. 참고하세요.
GeoTiff 파일 하나만 사용하기
NetCDF 의 attribute 설정을 하지 않겠습니다.
코드가 너무 길어져서 안 했습니다.
import rasterio
import numpy as np
from netCDF4 import Dataset
base_path = r'/bind_dir/netcdf_sample_inputs'
with rasterio.open(base_path + "/2000y_37612.tif", ) as src:
data = src.read(1) # Read single band as 2D array
xres, yres = src.res
width = src.width # 747 , x 축 픽셀의 갯수
height = src.height # 934 , y 축 픽셀의 갯수
# netcdf 는 항상 좌표 배열을 쓰기는 하는데, 좌표가 픽셀위 중앙 위치 좌표값으로 세팅해줘야 한다.
transform = src.transform # 픽셀의 좌표값을 실제 좌표값으로 치환하기 위한 Affine 인스턴스
"""
참고:
transform.a # x pixel size (width of a cell)
transform.e # y pixel size (height, usually negative)
transform.c # x origin (top-left)
transform.f # y origin (top-left)
"""
#### 난해한 부분(1) , 밑에서 설명하니 모르겠으면 PASS ####
# X center coordinates for each column
x_coords = np.arange(width) * transform.a + transform.c + (transform.a / 2)
# Y center coordinates for each row
y_coords = np.arange(height) * transform.e + transform.f + (transform.e / 2)
#### 난해한 부분(2) , 밑에서 설명하니 모르겠으면 PASS ####
# integer data type can't have _FillValue as np.nan
val_type = src.dtypes[0] # 첫번째 밴드의 [dtypes] 조회
is_float=np.issubdtype(np.dtype(val_type), np.floating) # numpy 로 현재 value 의 타입을 확인
if is_float:
nodata = src.nodata if src.nodata is not None else np.nan
else:
nodata = src.nodata
try:
# Create Dimensions
nc = Dataset(base_path + "/from_geotiff.nc", "w", format="NETCDF4")
nc.createDimension("x", width)
nc.createDimension("y", height)
nc.createDimension("time", None)
# Create Coordinate variables
x_var = nc.createVariable("x", "float32", ("x",)) # "f4", np.float32 모두 가능
y_var = nc.createVariable("y", "float32", ("y",)) # "f4", np.float32 모두 가능
t_var = nc.createVariable("time", "int16", ("time",)) # "i4", np.int16
x_var[:] = x_coords
y_var[:] = y_coords
t_var[:] = [0] # 일단 한개의 geotiff 만 사용하니,
# time 배열은 길이가 하나짜리인 것으로 만들겠습니다.
# Create data variable
v = nc.createVariable(
"my_variable",
val_type, ("time", "y", "x"), zlib=True, fill_value=nodata)
v[0, :, :] = data # 결과적으로 time=0 에 GeoTiff 배열인 (y,x) 2차원 배열이 들어갑니다.
except ValueError as e:
print(f"ValueError!! - {e}")
raise
except BaseException as e:
print(f"something went wrong! - {e}")
raise
finally:
print("✅ NetCDF saved from GeoTIFF")
nc.close()아마 이전 게시물에서 netcdf 생성하는 방법을 직접해보신 분들에게는
익숙한 코드가 많아서 큰 어려움이 없을 수 있습니다.
다만 위 코드에서 좀 난해할 수 있는 부분이 있는데,
이와 관련해서만 설명을 덧붙이겠습니다.
난해한 부분(1)
x_coords = np.arange(width) * transform.a + transform.c + (transform.a / 2)
y_coords = np.arange(height) * transform.e + transform.f + (transform.e / 2)이 코드는 NetCDF 에서 사용하는 x,y 좌표값을 구하기 위함입니다.
이를 쉽게 이해하기 위해서 다음 그림을 봅시다.
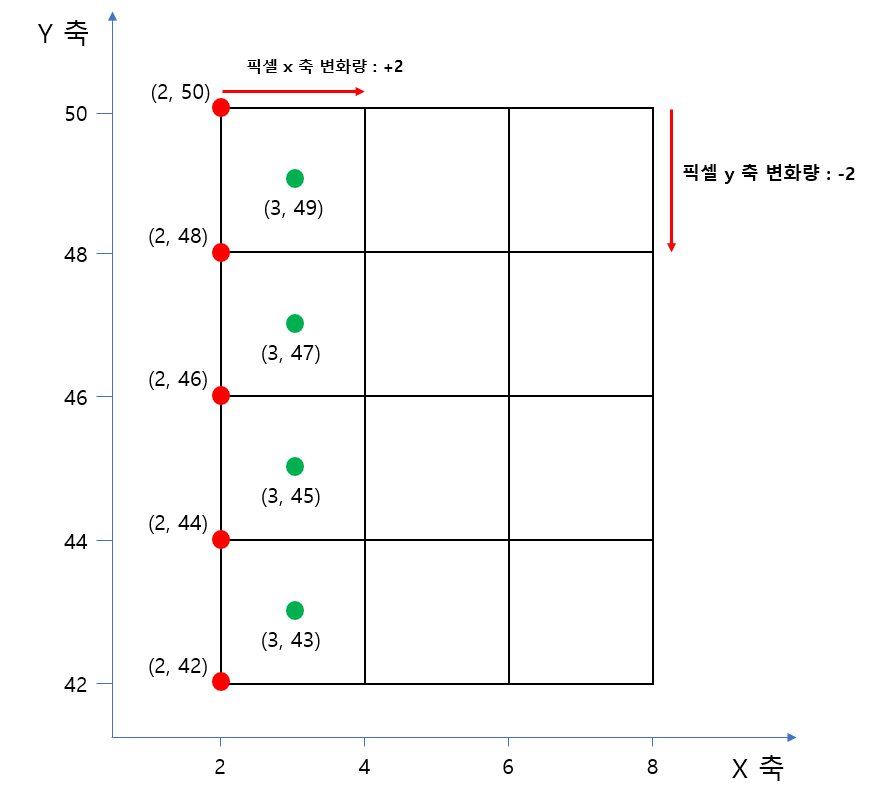
먼저 알아야 할 점이 NetCDF 는 GeoTiff 와 달리 좌표값을 설정할 때 pixel 의
꼭지점(빨간점)이 아니라, 픽셀의 중앙위치값(초록색 점)을 필요로 합니다.
그렇기 때문에 이와 관련해서 연산을 하는 부분이 바로 이 난해한 코드입니다.
이 코드의 이해를 위해서 위 그림이 실제 Geotiff 의 데이터 상태라고 가정해보겠습니다.
픽셀 하나의 크기는 (2 x 2) 이며, y 축의 변화량은 -2 인 것을 확인할 수 있습니다.
또한 전체 크기는 width: 3, height: 4 라는 것도 알 수 있네요.
자! 이제 y 축 하나로만 두고 천천히 생각해봅시다.
1. np.arange(height): [0, ..., height-1] 배열을 생성합니다.
==> np.arange(4) # [0, 1, 2, 3]
2. np.arange(height) * transform.e: 각 요소에 y축 변화량을 곱해줍니다.
==> [0, -2, -4, -6]
3. np.arange(height) * transform.e + transform.f
: 각 요소에 y 축의 origin (가장 Left, Top 의 좌표값) 을 더합니다.
: 현재 y 축 origin 은 50 입니다!
==> [50, 48, 46, 44] # 여기까지하면 GeoTiff 의 좌표값들이 연산된 겁니다.
4. np.arange(height) * transform.e + transform.f + (transform.e / 2)
: 마지막으로 y 축 변화량의 절반(`-2/2 = -1`)을 더해줍니다.
: 현재는 변화량이 음수니까 숫자가 작아진답니다.
==> [49, 47, 45, 43] 이렇게 해서 netcdf 에서 사용할 y 축의 좌표값이 연산된 겁니다.
x 축도 이와 마찬가지로 연산하게 됩니다.
난해한 부분(2)
그 다음으로 헷갈리는 부분이 아래 코드입니다.
val_type = src.dtypes[0] # 첫번째 밴드의 [dtypes] 지정함
is_float=np.issubdtype(np.dtype(val_type), np.floating)
if is_float:
nodata = src.nodata if src.nodata is not None else np.nan
else:
nodata = src.nodata이 코드는 데이터 타입에 따라 Nan 지정여부를 결정하는 코드입니다.
좀 풀어서 얘기해보겠습니다.
NetCDF 에서는 배열에 숫자형 데이터가 들어가고
이러한 숫자형 데이터들은 크게
정수형(integer, unsigned integer)실수형(float)
2가지로 나뉘게 됩니다.
여기서 주의할 점이 netcdf 는 _FillValue,
즉 배열에 중간에 값이 없을 때 사용되는 default 값으로 NaN 을 줄 수 있는데,
이 NaN 은 오로지 실수형(float) 타입에만 적용이 가능하다는 것입니다.
이러한 _FillValue 설정은 nc.createVariable 메소드 호출 시 적용되며,
이 메소드를 호출하기 전에 미리 float 형일 때만 NaN 이 적용되도록
하기 위해서 이 코드가 필요한 겁니다.
이 코드를 통해서 생성된 변수 nodata 는 추후 nc.createVariable 에서 사용됩니다.
# Create data variable
v = nc.createVariable(
"my_variable",
val_type, ("time", "y", "x"), zlib=True,
fill_value=nodata # 여기서 사용! 참고로 생략 가능.
)참고로
_FillValue를 지정 안하면(=생략하면),
각 타입에 따른 최대값을_FillValue로 간주합니다.
결과적으로 위 코드를 실행해서 netcdf 파일 하나를 생성했다면
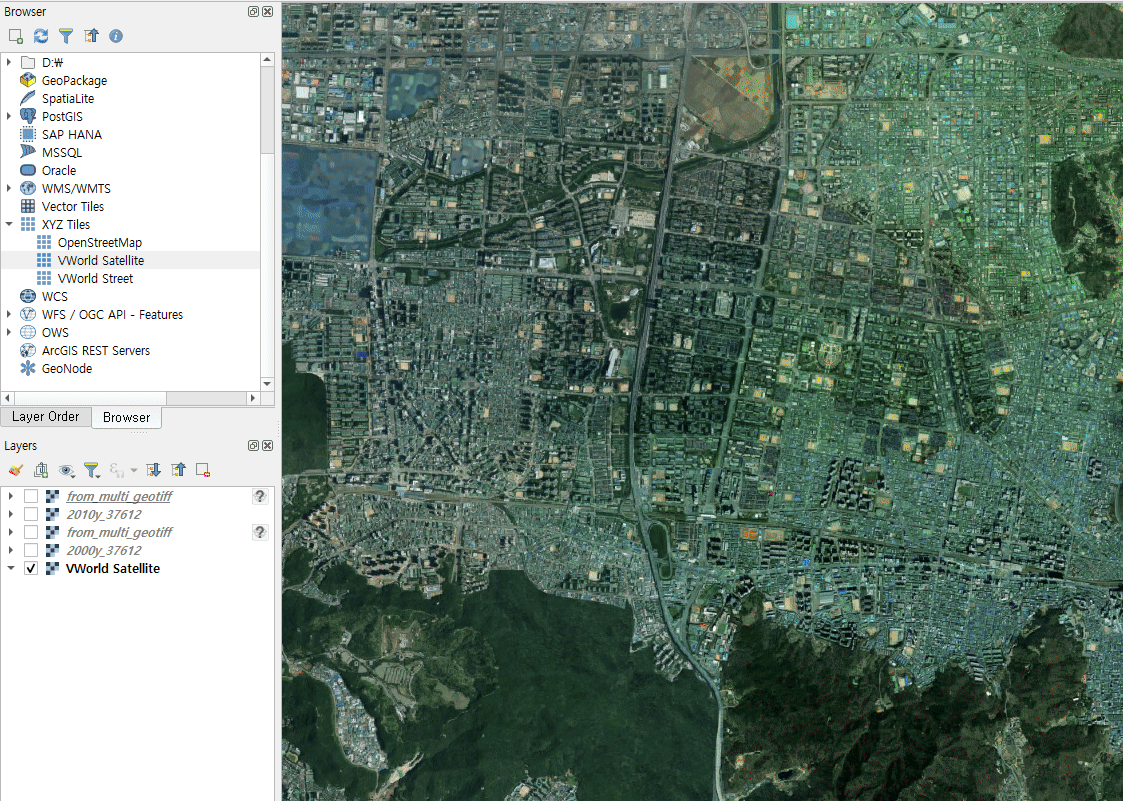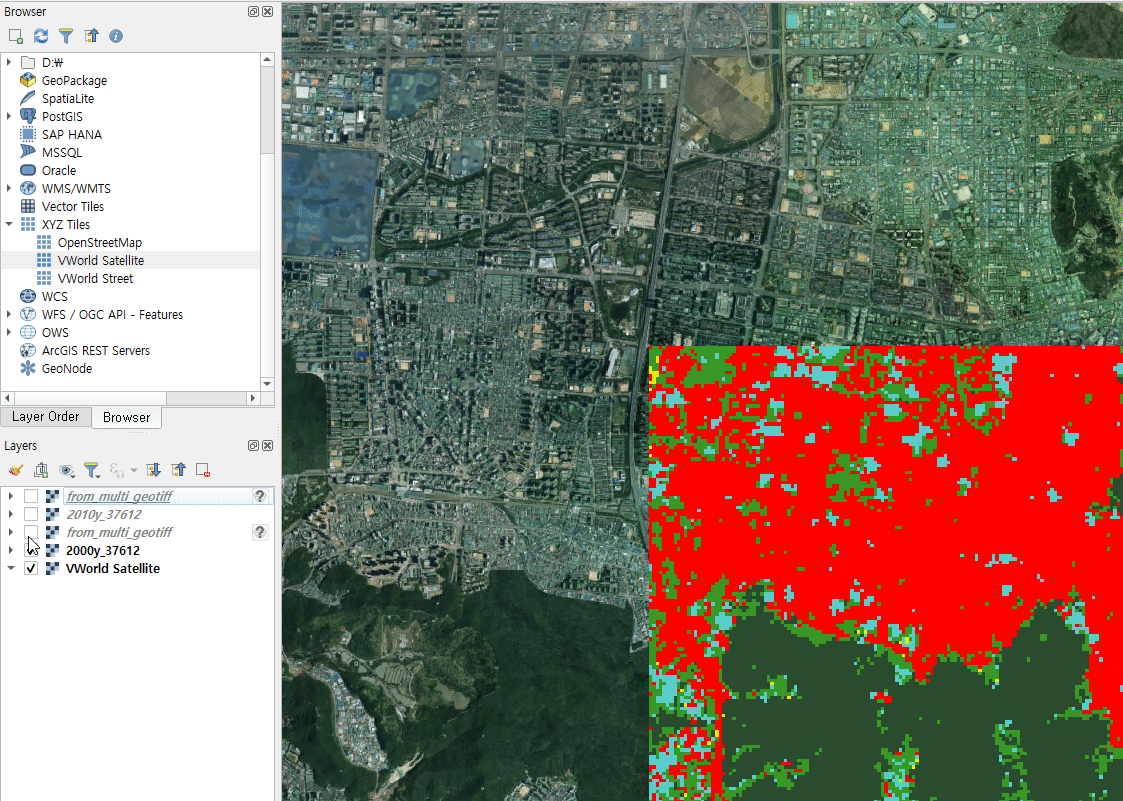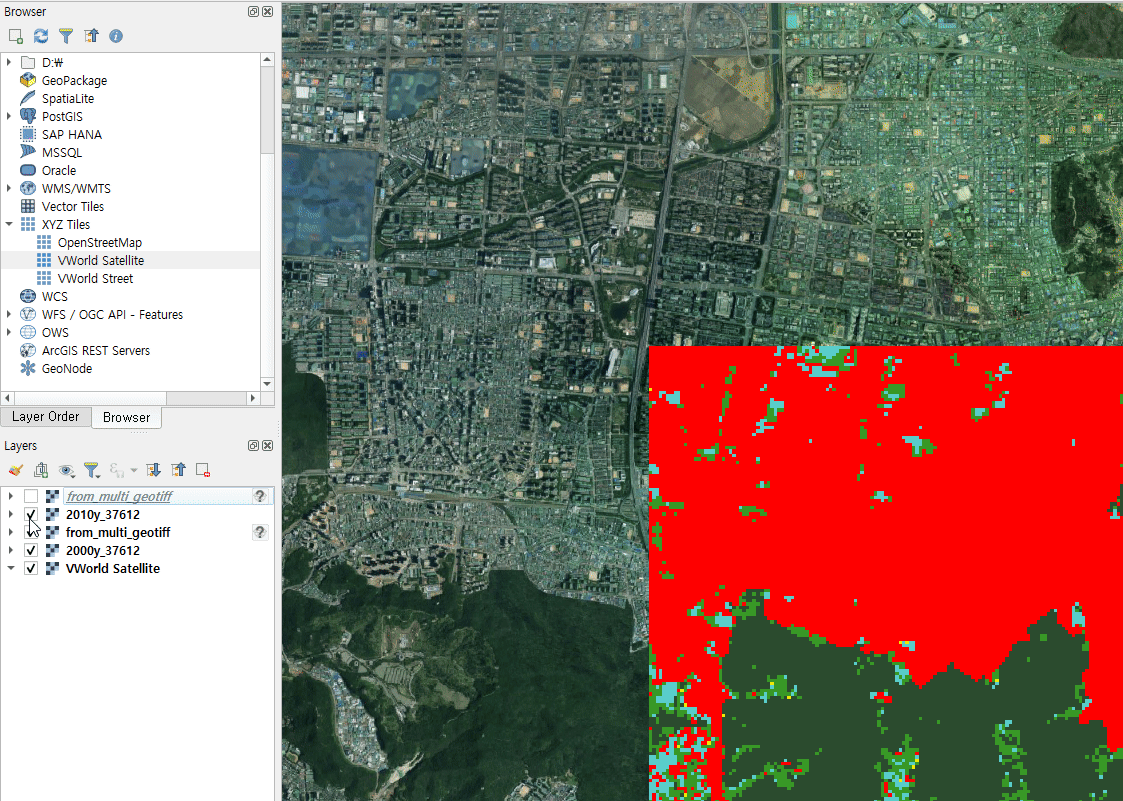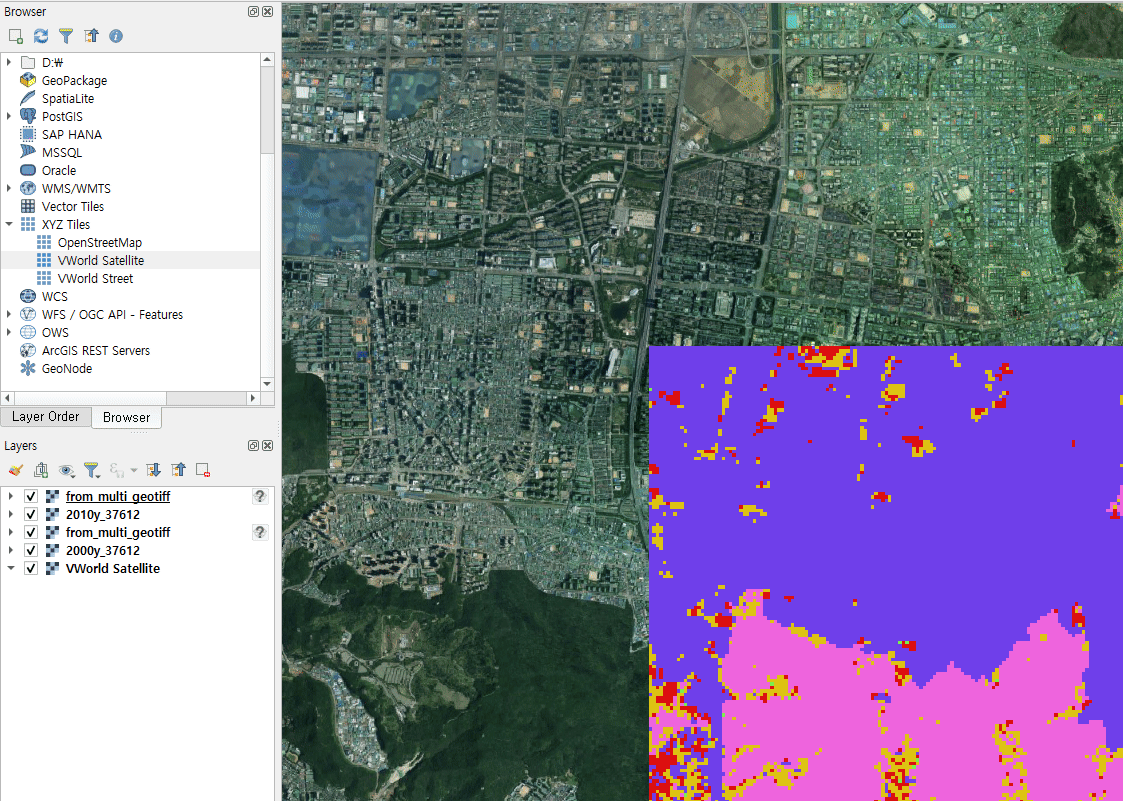
해당 파일을 QGIS 에서 가볍게 확인할 수 있습니다.
QGIS 에서 확인해보면 geotiff 파일과 netcdf 내용이 일치하는 것을 확인할 수 있습니다.
(QGIS 배경지도 CRS 를 EPSG:5186 으로 미리 맞춰놓은 상태입니다!)

-
from_geotiff가 nc 파일인데, 이 파일의 Layer Properties 에서
Render Type 을 Palatted 로 바꿔주고, Band 는 어짜피 하나밖에 없으니 그걸 선택하고,
Classify 버튼을 눌러서 각 지점마다 갖는 값에 색깔을 입힙니다. -
Layer Properties에서Band선택란은time dimension을 의미합니다.
현재는 저희가time dimension의 길이가 1 이므로 1개 밖에 안 나옵니다. -
2000y_37612 (=geotiff 파일 레이어)와from_geotiff (=nc 파일 레이어)와
색깔은 다르지만, 패턴이 똑같은 걸 확인할 수 있습니다.
정확히 데이터가 올바른 위치에 들어갔다는 의미죠.
GeoTiff 파일 여러개 사용하기
파일 하나만 사용해서 NetCDF 를 생성하는 것과 2개 이상을 하는 것에는 사실
큰 차이가 없습니다.
다만 여기서 조금 주의할 점이 있습니다.
여기서 사용되는 모든 GeoTiff 파일의 width, height, pixel size, x,y 축 변화량
이 모두 똑같으며, band 가 딱 한 개만 있다는 전제가 깔려있습니다. 유의해주세요.
저희가 받은 geotiff 파일은 모두 이에 부합하니 큰 문제가 되지 않습니다.
아무튼! 현재 저희는 2개의 geotiff 파일이 있는데,
아래와 같은 형태로 netcdf 파일에 데이터를 넣을 겁니다.
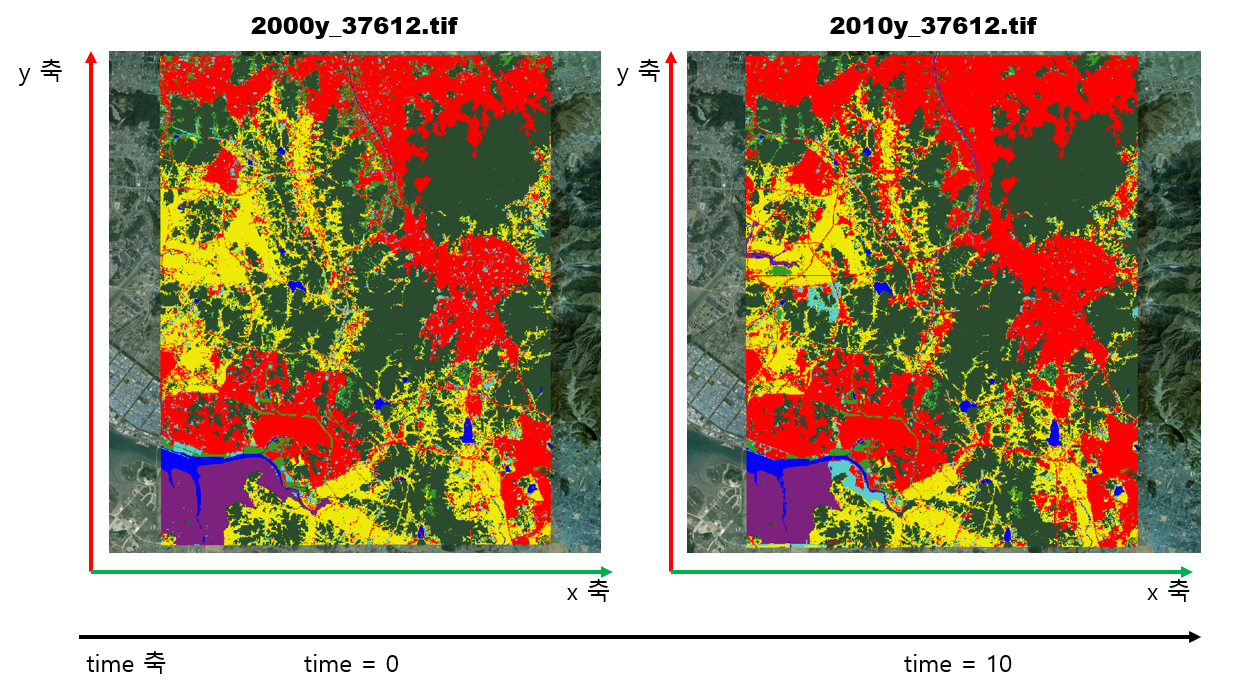
자 이제 코드로 보죠.
import rasterio
import numpy as np
from netCDF4 import Dataset
base_path = r'/bind_dir/netcdf_sample_inputs'
geotiff_files = [
'/bind_dir/netcdf_sample_inputs/2000y_37612.tif',
'/bind_dir/netcdf_sample_inputs/2010y_37612.tif',
]
t_axe_values = [0, 10]
t_var_length = len(t_axe_values) # 각 파일을 한 시간축에 존재하는 (y,x) 배열라고 가정한 겁니다.
### 참고로 모든 tif 파일은 똑같은 width, height, shape, resolution 을 갖고 있어야 합니다.
### 또한 모든 tif 가 single band 라는 전제도 깔려 있습니다.
### 안 그러면 정상적인 결과가 안 나올 수 있습니다.
### 저희가 다운로드 받은 예시 파일(tif)은 이를 모두 만족하므로 큰 문제가 없습니다.
### 이런 전제가 깔려 있으므로 아래 코드는 tif 파일 하나에 대해서만 메타정보를 조회합니다.
with rasterio.open(geotiff_files[0]) as sample_tif:
# data = sample_tif.read(1) # 여기서는 메타정보만 조회!
xres, yres = sample_tif.res
width = sample_tif.width # 747 , x 축 픽셀의 갯수
height = sample_tif.height # 934 , y 축 픽셀의 갯수
transform = sample_tif.transform
# X center coordinates for each column
x_coords = np.arange(width) * transform.a + transform.c + (transform.a / 2)
# Y center coordinates for each row
y_coords = np.arange(height) * transform.e + transform.f + (transform.e / 2)
# integer data type can't have _FillValue as np.nan
val_type = sample_tif.dtypes[0] # 첫번째 밴드의 [dtypes] 조회
is_float=np.issubdtype(np.dtype(val_type), np.floating) # numpy 로 현재 value 의 타입을 확인
if is_float:
nodata = sample_tif.nodata if sample_tif.nodata is not None else np.nan
else:
nodata = sample_tif.nodata
try:
# Create Dimensions
nc = Dataset(base_path + "/from_multi_geotiff.nc", "w", format="NETCDF4")
nc.createDimension("x", width)
nc.createDimension("y", height)
nc.createDimension("time", None)
# Create Coordinate variables
# 이번에는 타입 정보를 실제 데이터에서 읽어와 봤습니다.
x_var = nc.createVariable("x", np.array([xres]).dtype, ("x",))
y_var = nc.createVariable("y", np.array([yres]).dtype, ("y",))
t_var = nc.createVariable("time", np.array(t_axe_values).dtype, ("time",))
x_var[:] = x_coords
y_var[:] = y_coords
t_var[:] = t_axe_values
# Create data variable
v = nc.createVariable(
"my_variable",
val_type, ("time", "y", "x"), zlib=True, fill_value=nodata)
# t_var 배열의 시간축 배열값에 맞춰서 variable 에 배열 넣기
with rasterio.open(geotiff_files[0]) as tif1:
data1 = tif1.read(1) # 여기서는 메타정보만 조회!
v[0, : , :] = data1 # 1번째 time index 위치에 2차원 배열을 넣습니다.
with rasterio.open(geotiff_files[1]) as tif2:
data2 = tif2.read(1) # 여기서는 메타정보만 조회!
v[1, : , :] = data2 # 2번째 time index 위치에 2차원 배열을 넣습니다.
except ValueError as e:
print(f"ValueError!! - {e}")
raise
except BaseException as e:
print(f"something went wrong! - {e}")
raise
finally:
print("✅ NetCDF saved from Multiple GeoTIFF")
nc.close()QGIS 에서 확인하면 다음과 같습니다.
2000년도 geotiff,netcdf[time=0],2010년도 geotiff,netcdf[time=10]
순으로 한번씩 클릭해본 겁니다. (2개씩 패턴이 똑같죠?)
CSV 파일로 NetCDF 생성
geotiff 는 자기 자신이 이미 갖고 있는 메타정보가 많으므로
해당 정보들로 netcdf 만들 때 많은 부분 참조할 수 있습니다.
하지만 CSV 는 단순 텍스트 파일이기 때문에
어쩔 수 없이 수동으로 내용을 작성해줘야 합니다.
아래 코드를 보면 제가 직접 하드코딩해서 설정한 값들이 많다는 것을 확인할 수 있습니다.
import os
import zipfile
import pandas as pd
import numpy as np
from netCDF4 import Dataset
import json
base_path = r'/bind_dir/netcdf_sample_inputs'
csv_files = [
'/bind_dir/netcdf_sample_inputs/2000y_37612.csv',
'/bind_dir/netcdf_sample_inputs/2010y_37612.csv',
]
# === Step 1: Load one sample CSV to get x/y grid ===
import os
df_sample = pd.read_csv(csv_files[0], header=None)
print(df_sample.columns) # Index([0, 1, 2], dtype='int64')
x_vals = df_sample[0].unique()
y_vals = df_sample[1].unique()
t_vals = [0, 10] # 2개의 csv 파일은 각각 하나의 time index 를 갖는다.
print("len(x_vals)", len(x_vals)) # len(x_vals) 747
print("len(y_vals)", len(y_vals)) # len(y_vals) 934
x_len, y_len, t_len = len(x_vals), len(y_vals), len(t_vals)
try:
# === Step 2: Create NetCDF file ===
nc = Dataset(base_path + "/from_multiple_csv.nc", "w", format="NETCDF4")
# === Step 3: Create dimensions ===
nc.createDimension("x", x_len)
nc.createDimension("y", y_len)
nc.createDimension("time", None)
x_var = nc.createVariable("x", np.float32, ("x",))
y_var = nc.createVariable("y", np.float32, ("y",))
t_var = nc.createVariable("time", np.int8, ("time",))
x_var[:] = x_vals
y_var[:] = y_vals
t_var[:] = t_vals
# Create variable
v = nc.createVariable(
"my_variable",
np.int8,
("time", "y", "x"),
fill_value=0
)
# 3번째 column 에 값들이 들어 있으므로, 해당 열의 값들을 읽어서 numpy 배열로 만든다.
values1 = pd.read_csv(csv_files[0], header=None)[2].to_numpy(dtype=np.int8)
grid1 = values1.reshape((y_len, x_len)) # 1차원 numpy 배열을 reshape 을 통해서 2차원 배열로 접근할 수 있는 view 를 생성한다.
v[0, :, :] = grid1
values2 = pd.read_csv(csv_files[1], header=None)[2].to_numpy(dtype=np.int8)
grid2 = values2.reshape((y_len, x_len))
v[1, :, :] = grid2
except ValueError as e:
raise # re-throw error
finally:
print("[multiple csv -> single netcdf] Transform FINISH!")
nc.close()보충
attribute 설정법
이미 이전 글에서 많이 다뤄서 여기에는 쓰지 않았지만,
그냥 복습한다고 생각하고 추가 작성합니다.
(CSV 로 Netcdf 를 생성하는 코드 일부를 수정했습니다)
# === Step 2: Create NetCDF file ===
nc = Dataset(base_path + "/from_multiple_csv.nc", "w", format="NETCDF4")
## Global Attribute 설정
nc.setncattr("project", "My Blogging Sample Project")
# === Step 3: Create dimensions ===
nc.createDimension("x", x_len)
nc.createDimension("y", y_len)
nc.createDimension("time", None)
x_var = nc.createVariable("x", np.float32, ("x",))
y_var = nc.createVariable("y", np.float32, ("y",))
t_var = nc.createVariable("time", np.int8, ("time",))
x_var[:] = x_vals
x_var.setncattr("standard_name", "projection_x_coordinate")
x_var.setncattr("unit", "m")
y_var[:] = y_vals
y_var.setncattr("standard_name", "projection_y_coordinate")
y_var.setncattr("unit", "m")
t_var[:] = t_vals
t_var.setncattr("standard_name", "time")
t_var.setncattr("unit", "years since 2000-01-01")
# Create variable
v = nc.createVariable(
"my_variable",
np.int8,
("time", "y", "x"),
zlib=True,
fill_value=0
)
# 3번째 column 에 값들이 들어 있으므로, 해당 열의 값들을 읽어서 numpy 배열로 만든다.
values1 = pd.read_csv(csv_files[0], header=None)[2].to_numpy(dtype=np.int8)
grid1 = values1.reshape((y_len, x_len)) # 1차원 numpy 배열을 reshape 을 통해서 2차원 배열로 접근할 수 있는 view 를 생성한다.
v[0, :, :] = grid1
values2 = pd.read_csv(csv_files[1], header=None)[2].to_numpy(dtype=np.int8)
grid2 = values2.reshape((y_len, x_len))
v[1, :, :] = grid2
v.setncattr("standard_name", "my_sample_data")
v.setncattr("unit", "Unitless")이후 ncdump 로 확인해보면...
# 명령어: ncdump -h from_multiple_csv.nc
netcdf from_multiple_csv {
dimensions:
x = 747 ;
y = 934 ;
time = UNLIMITED ; // (2 currently)
variables:
float x(x) ;
x:standard_name = "projection_x_coordinate" ;
x:unit = "m" ;
float y(y) ;
y:standard_name = "projection_y_coordinate" ;
y:unit = "m" ;
byte time(time) ;
time:standard_name = "time" ;
time:unit = "years since 2000-01-01" ;
byte my_variable(time, y, x) ;
my_variable:_FillValue = 0b ;
my_variable:standard_name = "my_sample_data" ;
my_variable:unit = "Unitless" ;
// global attributes:
:project = "My Blogging Sample Project" ;
}주의! "_FillValue" 속성 중복 설정
variable 의 attribute 설정할 때 조금 주의할 게 하나 있는데,
그게 바로 _FillValue 속성입니다.
_FillValue 속성은 nc.createVariable 을 호출한 순간 무조건 지정됩니다.
그렇기 때문에 nc.createVariable 호출 후에, 다시 세팅을 하려고 하면 에러가 납니다!
에러 예시:
# Create variable
v = nc.createVariable(
"my_variable",
np.int8,
("time", "y", "x"),
zlib=True,
fill_value=0 # 변수 생성 시점부터 FillValue 가 설정하는 것을 확인할 수 있다.
# (주의) fill_value 인자를 직접적으로 안 줘도 FillValue 는 설정됩니다!
)
v.setncattr("_FillValue", 1)
# 에러!
# AttributeError:
# _FillValue attribute must be set when variable is created
# (using fill_value keyword to createVariable)이상으로 글을 마치겠습니다.

