Abstract
- annotatated된 샘플을 최대한 활용하여 학습하는 기법 제안
- contracting path(capture context)+symmetric expanding path(precise localization)으로 구성
Motivation
- 의료분야는 annotation이 있는 데이터를 만들기가 특히 어려움 (비용+개인정보 문제)
- 명확한 경계를 구분하기 어려운 경우가 많음 (ex. 세포 단위의 작업 등)
Contribution
- encoder가 확장함에 따라 채널 수를 1024까지 증폭 좀 더 고차원에서 정보를 매핑
- 각기 다른 계층의 encoder 출력을 decoder와 결합, 이전 레이어의 정보를 효율적으로 활용
구조 및 특징
구조
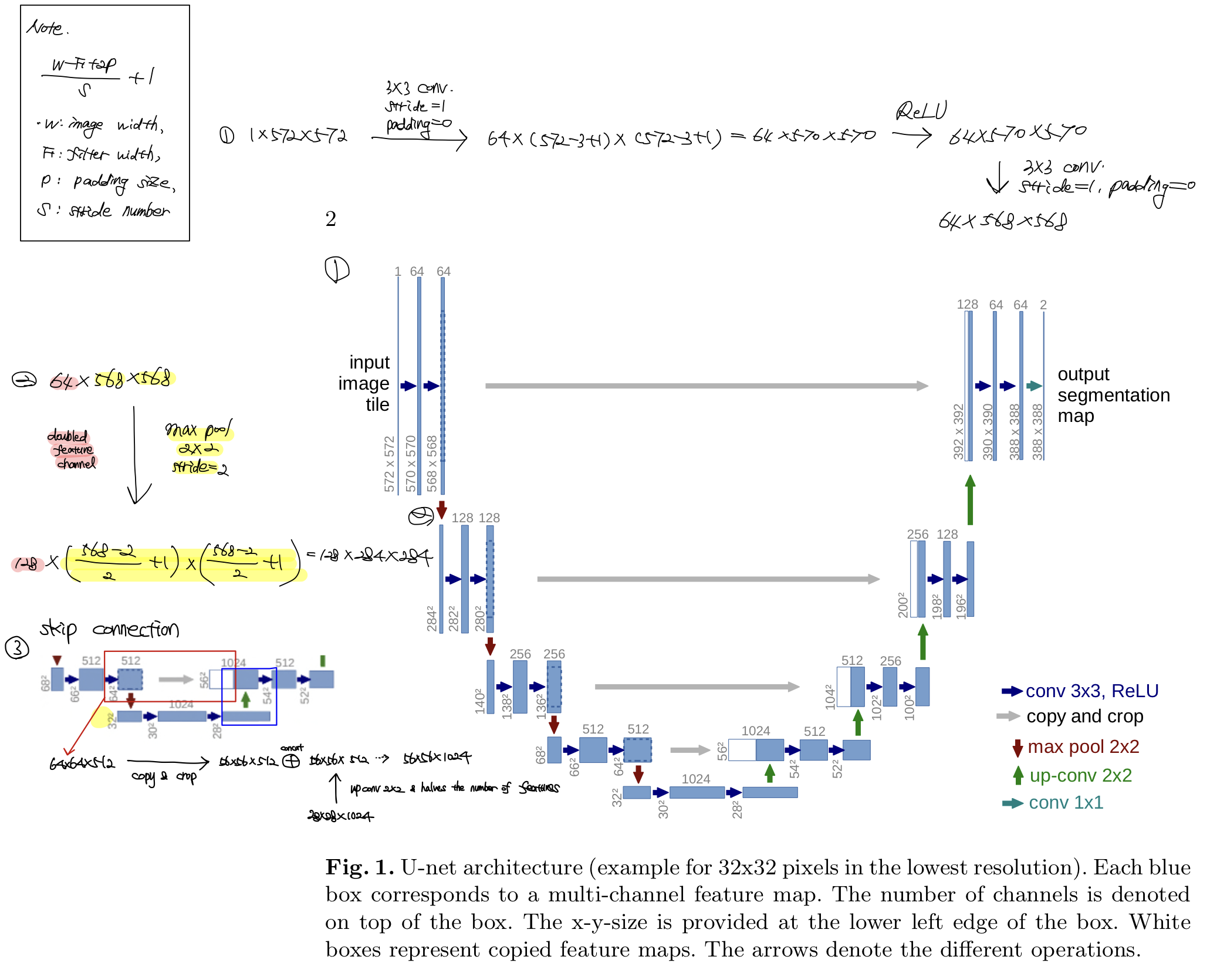
| contracting path | expanding path |
|---|---|
| 입력 이미지의 전반적인 특징을 추출 | localization |
| max-pooling 과정을 통해 down-sampling | 차원을 늘리는 up-samling |
- contracting path
- (3x3 컨볼루션+ReLU)*2
- 2x2 max pooling with stride 2을 적용하여 down sampling을 수행, 이 때 채널 수를 2배로 늘림
- expanding path
- 2x2 컨볼루션을 통해 up sampling을 수행하고 채널 수를 절반으로 줄임, 이 때, contracting path로부터의 특성 맵(feature map)을 사이즈에 맞게 크롭하여 이어붙임
- 컨불루션 연산을 수행하면 각 경계에 대한 정보를 조금씩 잃게 되므로, 이를 보완하고자 크롭을 수행
- (3x3 컨볼루션+ReLU)*2
Random elastic deformation 증강 기법
동일한 세포여도 사람에 따라 형태가 매우 다르므로, 형태를 다양하게 만들고자 유도한 증강 기법
Pixel-wise weight을 계산하기 위한 weight map 생성
같은 클래스를 가지는 인접한 셀을 분리하기 위해 경계부분에 가중치를 제공
- : 클래스의 빈도를 조정하기 위한 weight map
- : 가장 인접한 셀의 경계까지의 거리
- : 두번째로 인접한 셀의 경계까지의 거리
세로 중앙의 픽셀이라면, 값이 큼 exp 값은 작아짐 중앙의 픽셀일수록 weight값이 작음 (경계에 위치할수록 weight이 큼!) 경계를 잘 맞추면 loss가 더 작음
한계점
- 깊이가 4로 고정되어 있으므로, 데이터셋에 따라서 성능 차이가 많이 날 수 있음
- 단순한 skip connection 구조를 가지고 있어 보다 다양한 표현을 학습하지 못함
코드
class DoubleConv(nn.Module):
"""(3x3 컨볼루션+batch normalization+ReLU)*2 수행"""
def __init__(self, in_channels, out_channels, mid_channels=None):
super().__init__()
if not mid_channels:
mid_channels = out_channels
self.double_conv = nn.Sequential(
nn.Conv2d(in_channels, mid_channels, kernel_size=3, padding=1, bias=False),
nn.BatchNorm2d(mid_channels),
nn.ReLU(inplace=True),
nn.Conv2d(mid_channels, out_channels, kernel_size=3, padding=1, bias=False),
nn.BatchNorm2d(out_channels),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.double_conv(x)
class Down(nn.Module):
"""2x2 max pooling with stride 2을 적용하여 down sampling을 수행+double conv"""
def __init__(self, in_channels, out_channels):
super().__init__()
self.maxpool_conv = nn.Sequential(
nn.MaxPool2d(2),
DoubleConv(in_channels, out_channels)
)
def forward(self, x):
return self.maxpool_conv(x)
class Up(nn.Module):
"""2x2 컨볼루션을 통해 up sampling을 수행+double conv"""
def __init__(self, in_channels, out_channels, bilinear=True):
super().__init__()
# if bilinear, use the normal convolutions to reduce the number of channels
if bilinear:
self.up = nn.Upsample(scale_factor=2, mode='bilinear', align_corners=True)
self.conv = DoubleConv(in_channels, out_channels, in_channels // 2)
else:
self.up = nn.ConvTranspose2d(in_channels, in_channels // 2, kernel_size=2, stride=2)
self.conv = DoubleConv(in_channels, out_channels)
def forward(self, x1, x2):
x1 = self.up(x1)
# input is CHW
diffY = x2.size()[2] - x1.size()[2]
diffX = x2.size()[3] - x1.size()[3]
x1 = F.pad(x1, [diffX // 2, diffX - diffX // 2,
diffY // 2, diffY - diffY // 2])
# if you have padding issues, see
# https://github.com/HaiyongJiang/U-Net-Pytorch-Unstructured-Buggy/commit/0e854509c2cea854e247a9c615f175f76fbb2e3a
# https://github.com/xiaopeng-liao/Pytorch-UNet/commit/8ebac70e633bac59fc22bb5195e513d5832fb3bd
x = torch.cat([x2, x1], dim=1) #concatenation 수행!
return self.conv(x)
class OutConv(nn.Module):
def __init__(self, in_channels, out_channels):
super(OutConv, self).__init__()
self.conv = nn.Conv2d(in_channels, out_channels, kernel_size=1)
def forward(self, x):
return self.conv(x)
class UNet(nn.Module):
def __init__(self, n_channels, n_classes, bilinear=False):
super(UNet, self).__init__()
self.n_channels = n_channels
self.n_classes = n_classes
self.bilinear = bilinear
self.inc = (DoubleConv(n_channels, 64))
self.down1 = (Down(64, 128))
self.down2 = (Down(128, 256))
self.down3 = (Down(256, 512))
factor = 2 if bilinear else 1
self.down4 = (Down(512, 1024 // factor))
self.up1 = (Up(1024, 512 // factor, bilinear))
self.up2 = (Up(512, 256 // factor, bilinear))
self.up3 = (Up(256, 128 // factor, bilinear))
self.up4 = (Up(128, 64, bilinear))
self.outc = (OutConv(64, n_classes))
def forward(self, x):
x1 = self.inc(x)
x2 = self.down1(x1)
x3 = self.down2(x2)
x4 = self.down3(x3)
x5 = self.down4(x4)
x = self.up1(x5, x4)
x = self.up2(x, x3)
x = self.up3(x, x2)
x = self.up4(x, x1)
logits = self.outc(x)
return logits참고문헌
