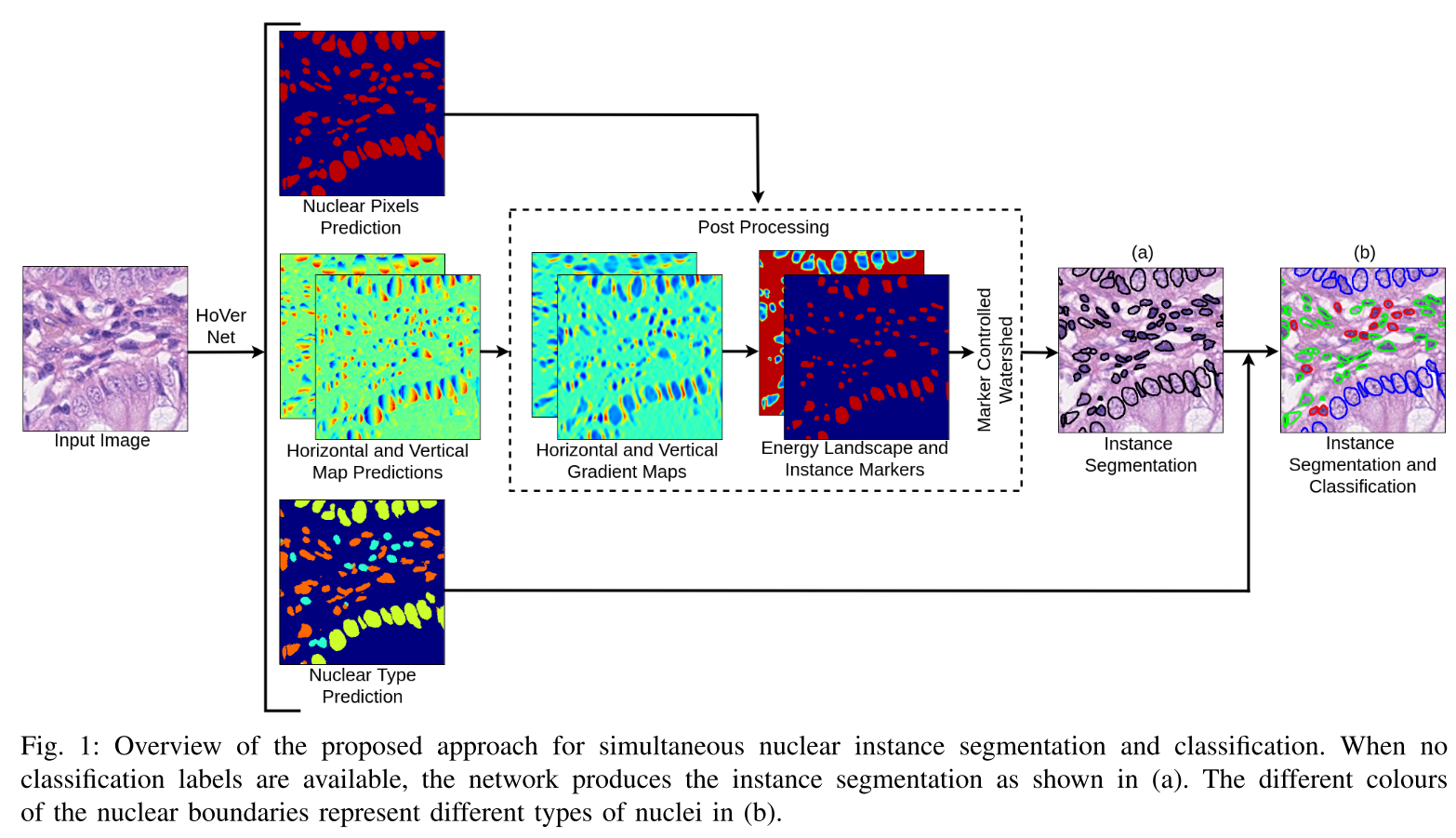
[2019 MIA] Hover-Net: Simultaneous segmentation and classification of nuclei in multi-tissue histology images
Paper Review
2019년 다양한 H&E 염색 이미지 데이터셋에서 SOTA를 달성한 nuclei segmentation 알고리즘
Main Contribution
- 인접한 nuclei를 구분하기 위해 cell의 center point에서 horizontal 방향(+x)의 distance 맵과 vertical 방향(+y)의 distance 맵 활용하는 DL 알고리즘 제안
- DICE와 AJI의 맹점을 논하며 Panoptic Quality 처음 제안함
- CoNSeP dataset을 처음 제안함
Method
Network Architecture and Loss
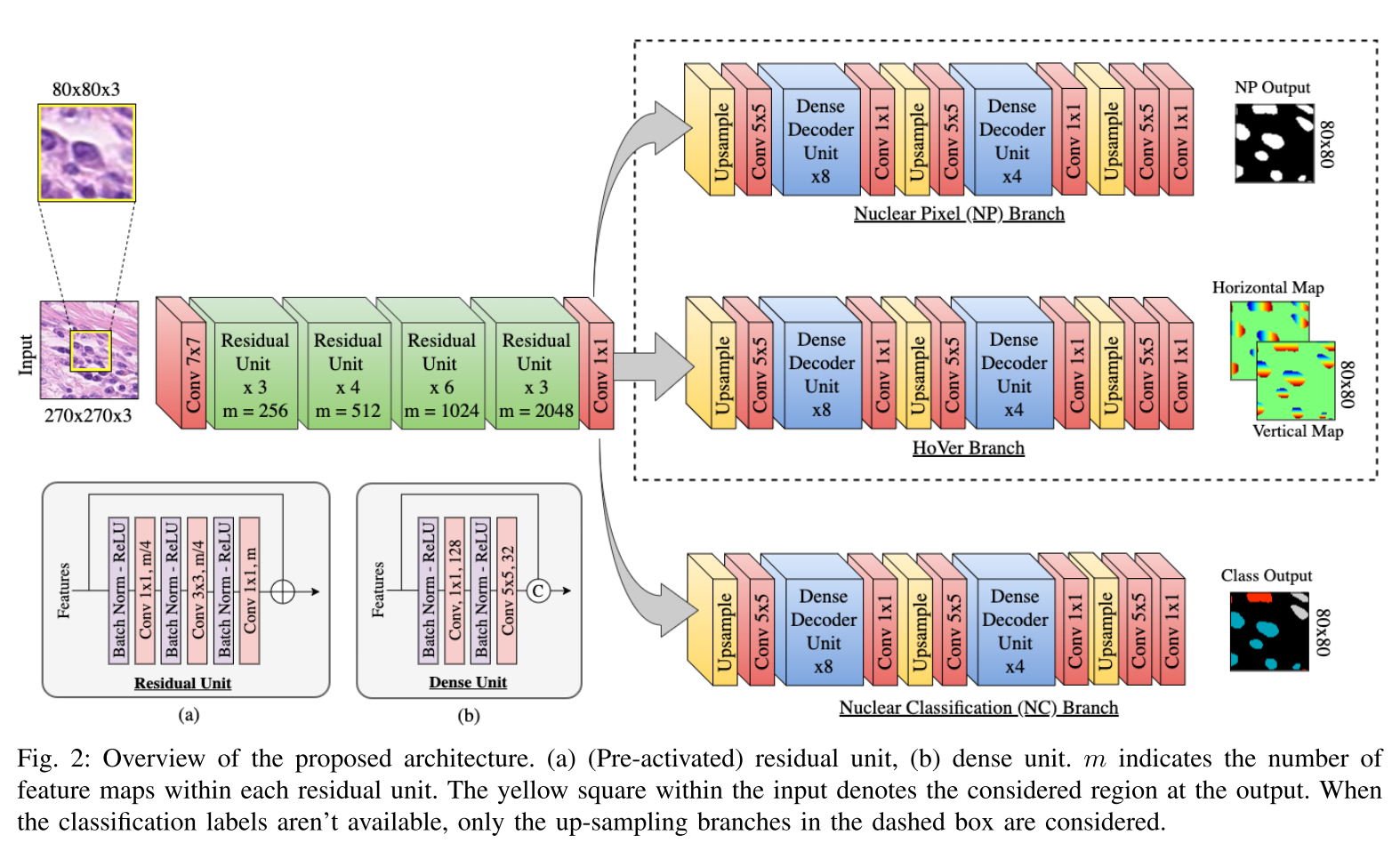
크게 보면 1 enconder + 3 decoder branches
- nuclear 유무만 예측하는 Nuclear Pixel (NP) branch
- horizontal map과 vertical map을 예측하는 HoVer branch
- nuclear의 class를 예측하는 Nuclear Classification branch
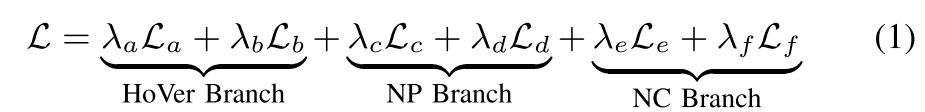
HoVer branch 빼고는 각각에 대한 CE loss + Dice loss의 조합
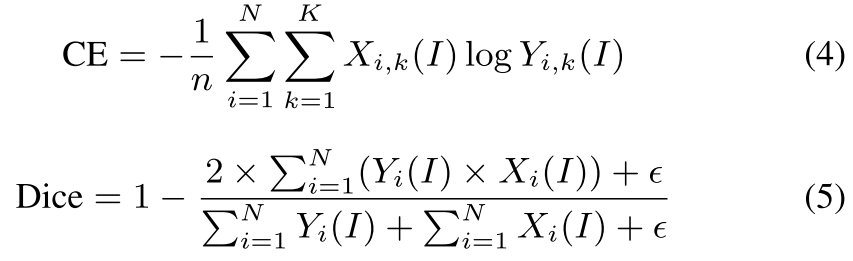
HoVer branch를 알아보자

horizontal 방향 (+x)과 vertical 방향 (+y)으로 center에서 distance를 만들면, cell이 인접한 방향에 대해서 자연스럽게 값의 차이가 커진다
그림에서 화살표는 경계에서 값의 차이가 확 커지는 경우를 설명해준다
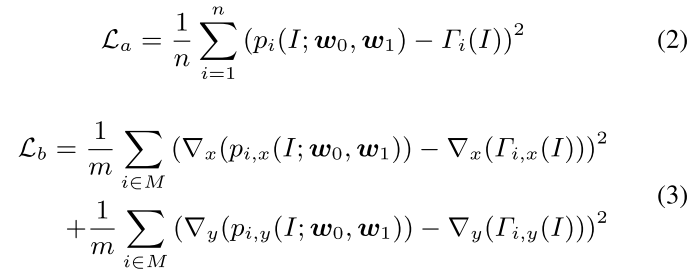
이를 MSE로 map을 그냥 regression하는 것도 있지만, 이 경우 gradient를 각 방향으로 취해 그 gradient를 regression해주는 loss를 추가로 제안한다
경계 부분에서 차이가 커져서 인접한 방향에 대해서 gradient가 크게 나타나는 gradient map을 상상해보면 될 것 같다
이를 통해 인접한 nuceli를 separate하는 데에 사용한다
Post-processing
각 방향의 gradient 값은 인접한 nuclei 사이에 높은 값이 나타나게 하는데, pixel 값 중에 유의미한 (significant) 차이를 이와 같이 정의함
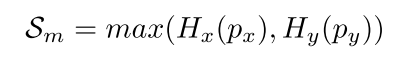
p_x, p_y: Horizontal and Vertical Map Predictions
H_x, H_y: 각 방향의 sobel kernel로 gradient를 근사화해서 구해주는 function
인접한 nuclei 사이에 유의미한 값의 차이를 통해 marker와 energy landscape를 정의한다
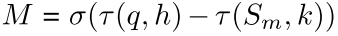
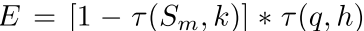
q: 해당 pixel에 NP branch의 Nuclear가 있을 확률값
h, k: 성능이 제일 잘 나오는 걸로 골랐다고 함
: 음수는 0으로 만드는 rectifier
: a가 b보다 크면 1, 작으면 0
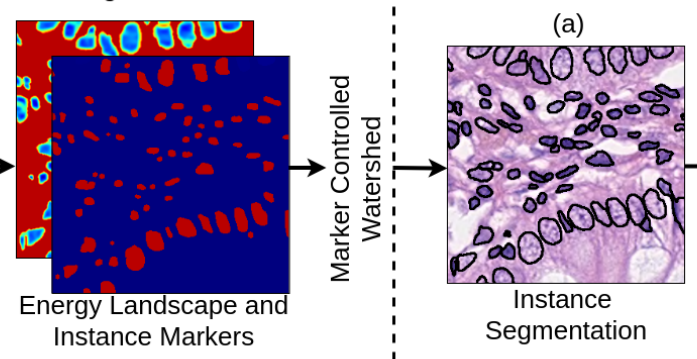
marker 는 주어진 energy landscape 에서 를 어떻게 split할 지 결정하는 marker-controlled watershed의 marker로 사용된다.
뒤의 실수 값이 남아있는 Instance Marker를 통해 Marker Controlled Watershed를 한 건 그림으로 이해가 되는데, 주어진 energy landscape에서 를 어떻게 split한다는 건지는 설명이 더 나와있지 않아 이해가 잘 안 간다.
Metric
Nuclear Instance Segmentation Evaluation

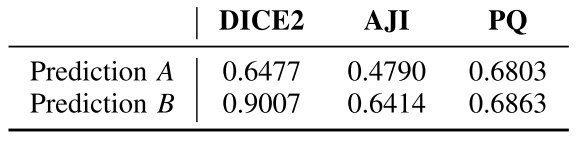
DICE2와 AJI (Aggregated Jaccard Index) 의 경우, Prediction B의 성능이 더 높게 나오고 Prediction A의 성능이 너무 낮게 나온다.. DICE2의 경우 그 차이가 더 심하다
Prediction A처럼 인접한 instance의 misclassification된 부분이 over-penalisation되는 경향이 있다는 것이다
DICE2는 '2x(겹치는 영역)/(각 영역의 크기의 합)'을 각 nuclei마다 구하니 Prediction A의 경우, cyan 색의 nuclei말고도 blue 색의 nuclei에 대해서도 penalisation을 받게 된다.
AJI는 (교집합의 합/합집합의 합)으로 계산되어서 그나마 덜하지만, 역시 인접한 부분에 대해서 침범하는 쪽에 대한 예측 성능이 penalisation되는 경향이 있다.
그래서 이 논문에서 Panoptic Quality를 제안한다
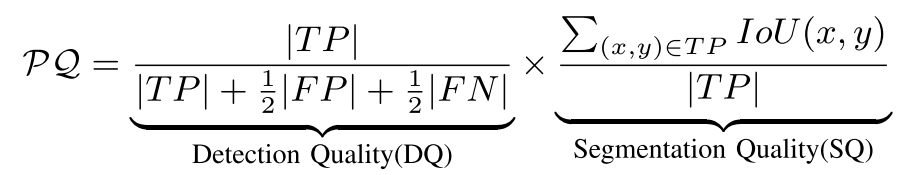
해당 sample에 대한 detection quality를 추정하는 term과 segmentation quality를 추정하는 term인데, segmentation quality의 경우 true positive가 있는 영역에 한해서 IoU를 계산해 ratio를 계산해준다
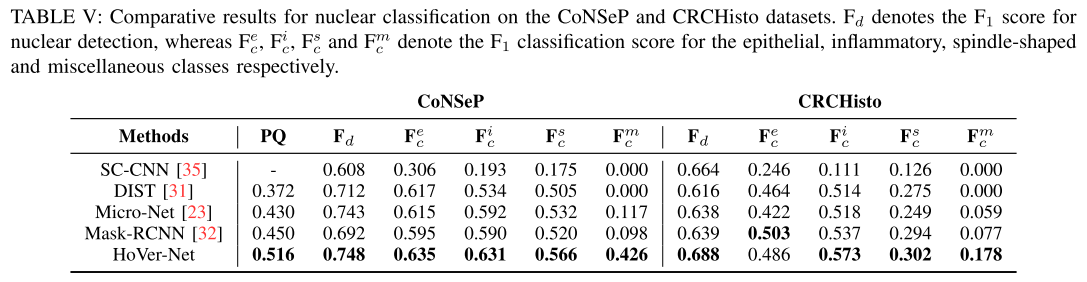
Nuclear Classification Evaluation
특정 type t의 F_c score를 다음과 같이 정의함. , 로 줬는데, nuclear classification을 좀 더 강조하기 위해
TP_c나 TP_d나 같다고 보면 됨
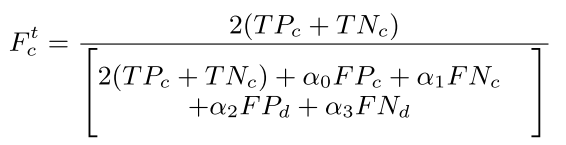
전체 Type T에 대한 overall F_c score는 다음과 같이 정의함

Experiments
Datasets
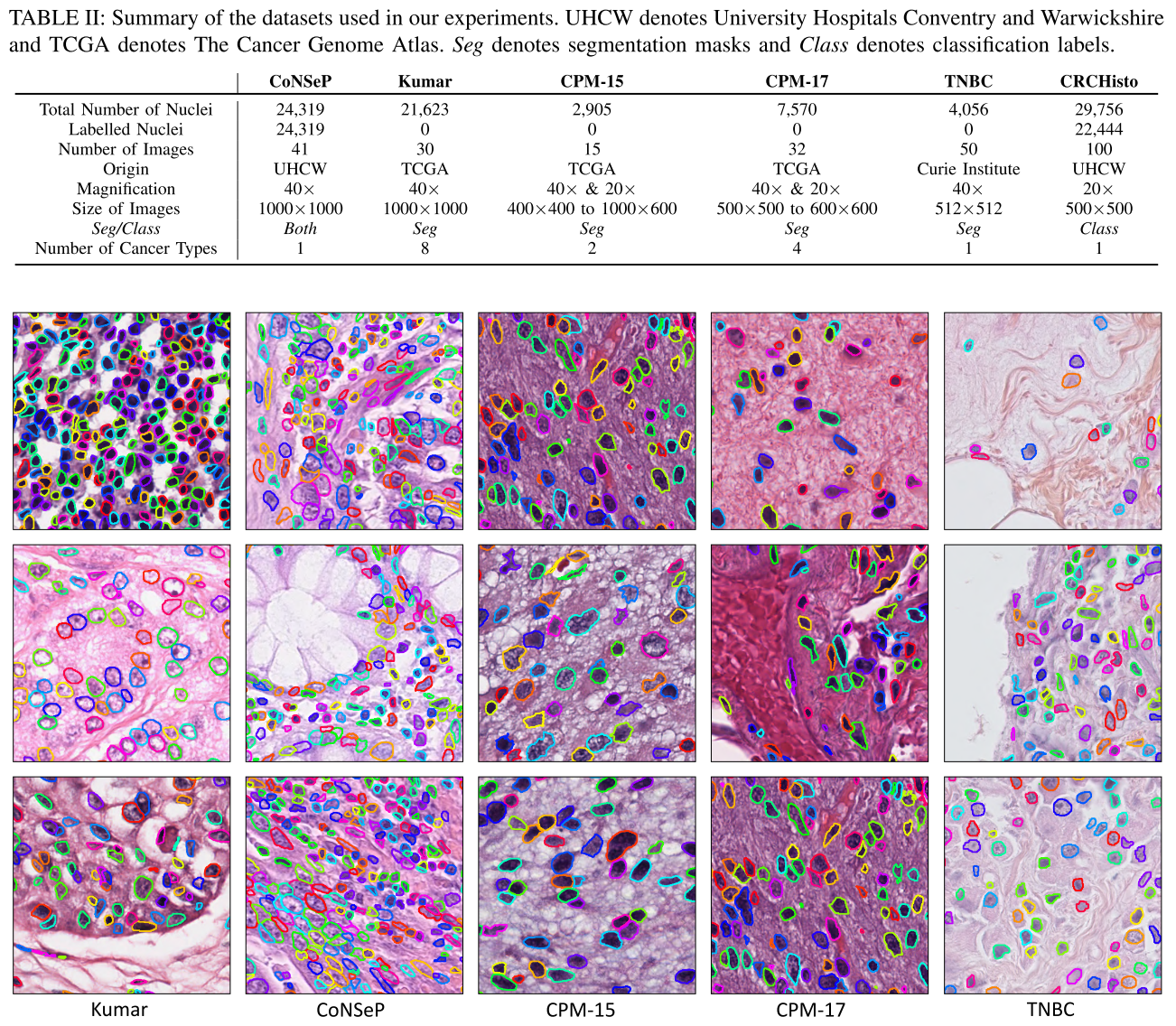
CoNSeP dataset는 이 논문에서 새롭게 소개하는 데이터셋
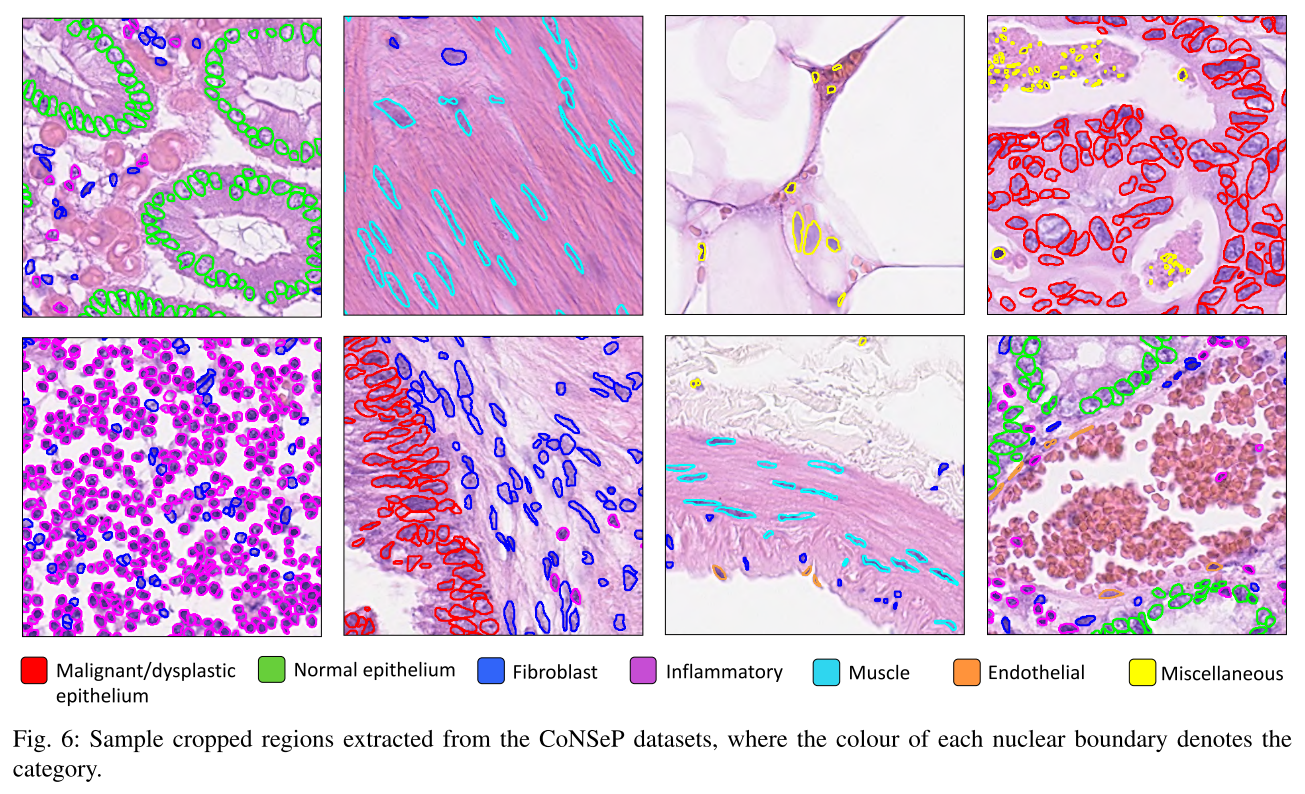
Training
input size: 252 X 252 ~ 270 X 270 (due to the use of valid convolutions)
augmentation: flip, rotation, Gaussian blur, median blur
network: ImageNet에서 사전훈련된 weight으로 model을 초기화
two stage: only decoder (50 epoch) -> encoder + decoder (50 epoch, fintuning)
optim: Adam
learning rate: 1e-4로 시작해서 25 epoch 이후엔 1e-5 (각 stage마다 반복)
batch size: only decoder (82) -> encoder + decoder (42)
두 개의 NIVDIA GeForece 1080 Ti GPU로 stage마다 120분, 260분 총 380분 정도 걸렸다
Results
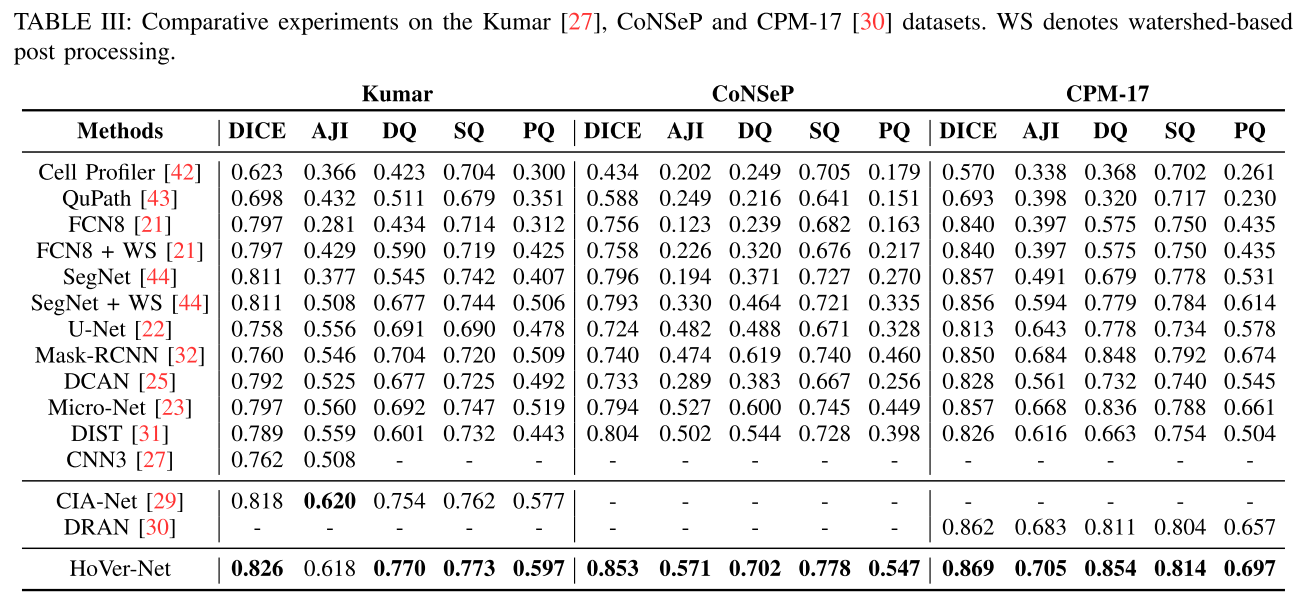
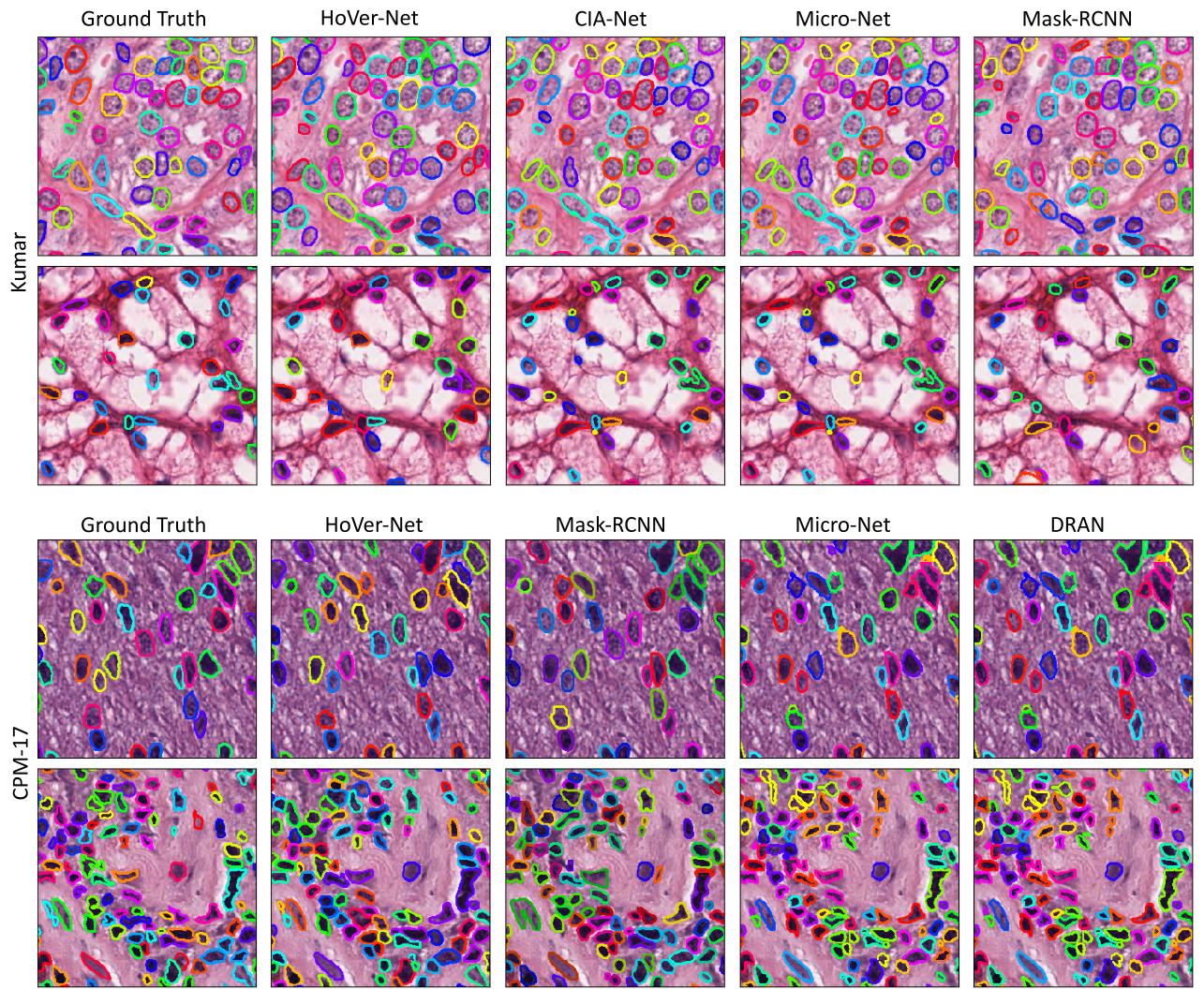
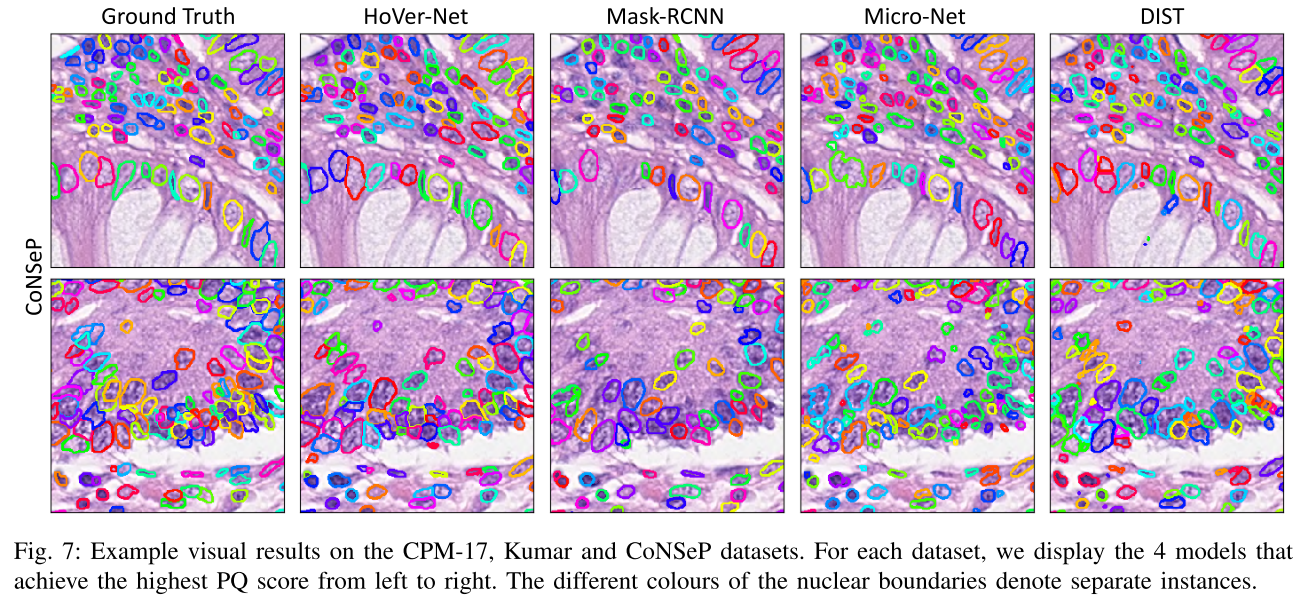
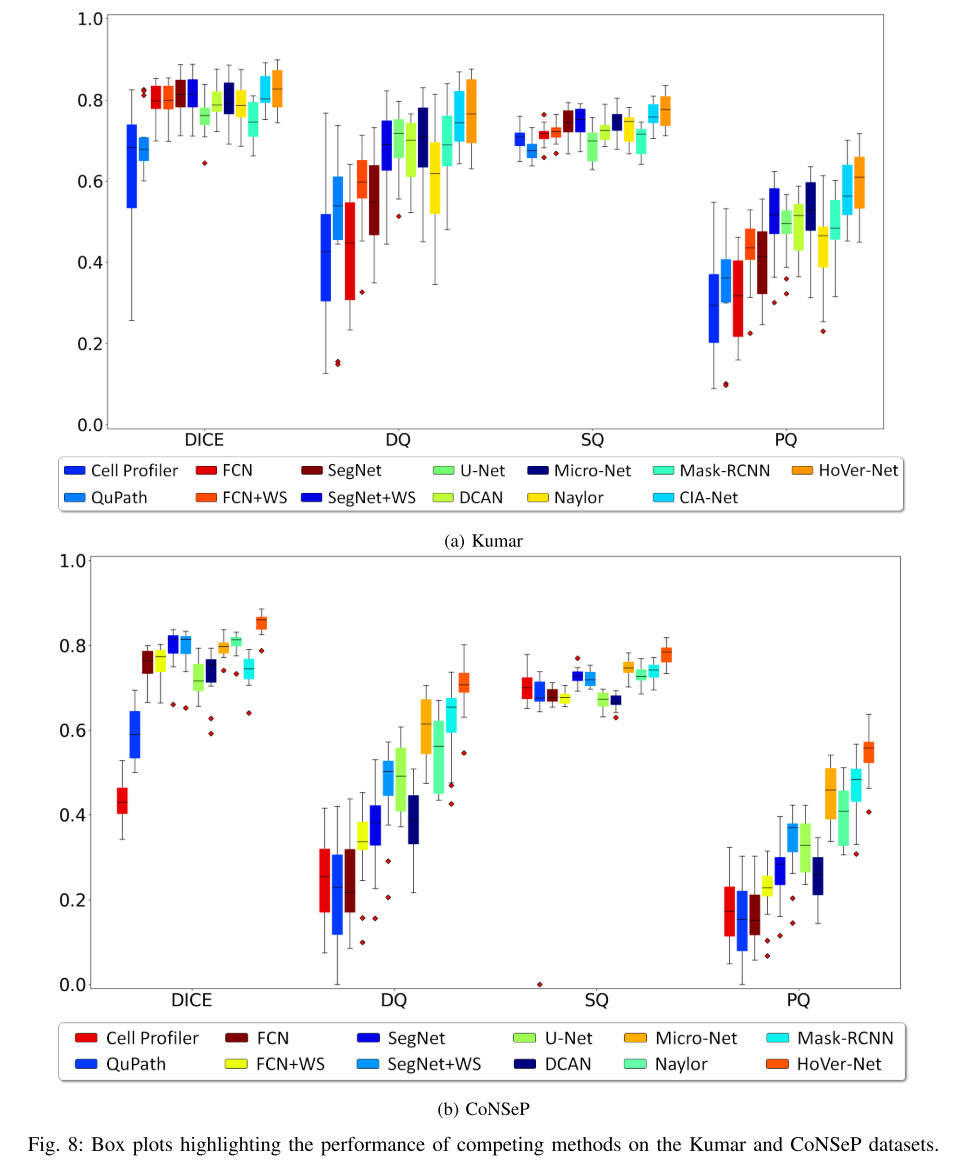
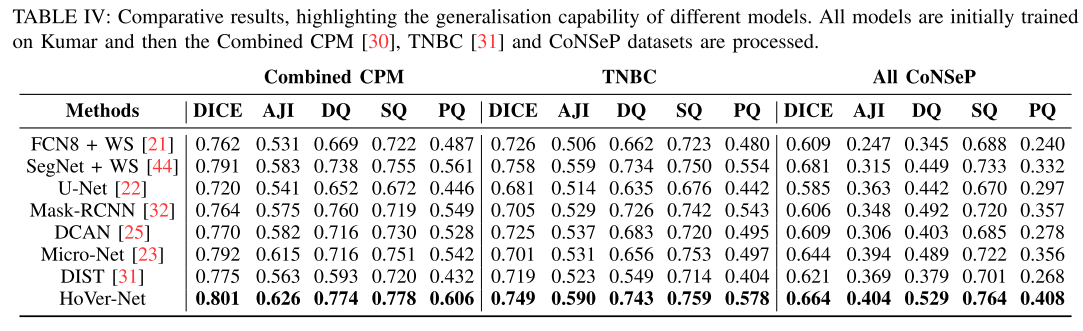
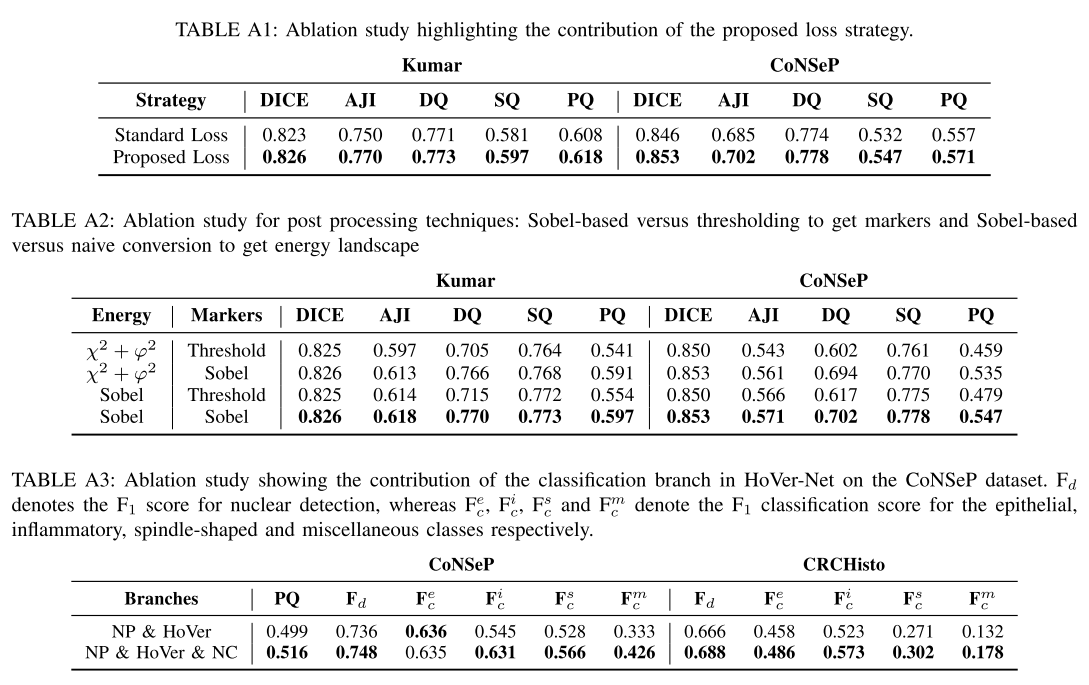
Ablation Study
Conclusion & Discussion
- distance 기반의 horizontal map과 vertical map을 예측하고 활용하는 알고리즘으로 다양한 데이터셋, 당대 최신 모델과 extensive한 실험결과로 SOTA를 입증한 것은 물론, loss와 post-processing, NC branch 등에 대해서 ablation study를 통해 효과를 입증했다
- paper-with-code를 보면 여전히 Kumar에선 SOTA를 달성하고 있다. paper-with-code nuclei segmentation benchmark가 많이 등록 안 되있어서 tracking하려면 논문을 다 봐야함
- distance를 이용한 관점에서 StarDist와 유사하지만, StarDist는 star-convex polygon이라는 모형을 가정하고 instance의 boundary가 주어지면 32개의 방향으로 거리를 재서 instance이 존재할 확률이 instance의 중앙에서 커지고 경계로 갈수록 값이 작아지는 원리를 쓴 거고
- HoVer-Net은 수직, 수평 2방향에 대해서만 +/- 값의 distance로 map을 만들어 인접한 경계에서 큰 값을 가질 수 있도록 gradient까지 활용한 것으로 좀 더 간단하면서도 똑똑하게 접근했다고 생각된다.
