Introduction
붗꽃의 품종을 분류해보자. (데이터 셋 다운로드)
문제 정의
붗꽃은 꽃잎 모양과 길이를 기준으로 품종을 구분한다.

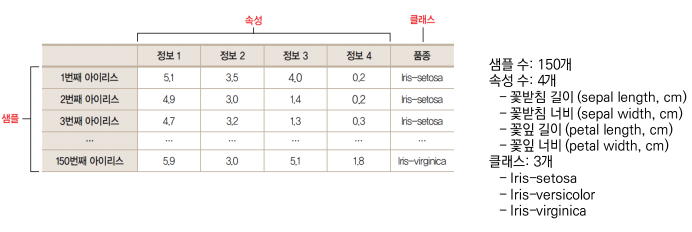
데이터 세트 확인
다음과 같이 총 4개의 속성으로 이루어짐을 확인할 수 있다.
상관도 그래프
클래스간 상관도를 알아보자(상관도 == 유사도) 얼마나 각 데이터들이 서로 상관되어있는지를 확인한다.
뚜렷하게 구분하기 위해서 이러한 상관도는 낮을수록 좋다.(많이 다르므로 구분하기 좋음)
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
df = pd.read_csv(my_data, names = ["sepal_length", "sepal_width",
"petal_length", "petal_width", "species"])
sns.pairplot(df, hue='species');
plt.show()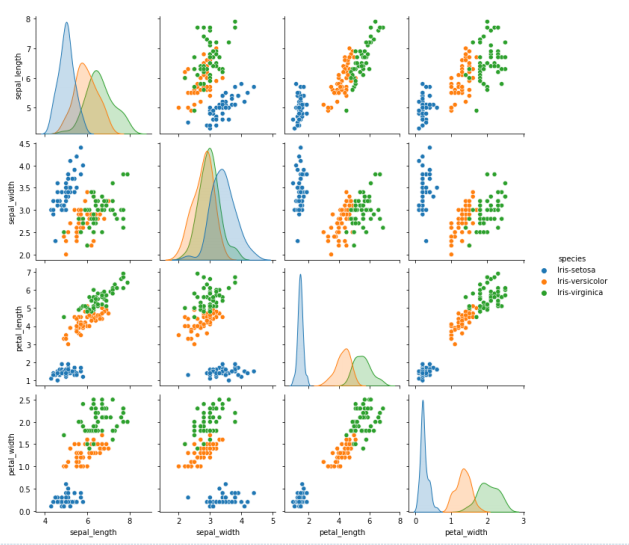
원-핫 인코딩
컴퓨터가 이해할 수 있는 언어로 레이블을 변경해준다.
from sklearn.preprocessing import LabelEncoder
e = LabelEncoder()
e.fit(Y_obj)
Y = e.transform(Y_obj)
Y_encoded = tf.keras.utils.to_categorical(Y)모델 정의 및 컴파일
model = Sequential()
model.add(Dense(16, input_dim=4, activation='relu'))
model.add(Dense(3, activation='softmax’))
model.compile(loss='categorical_crossentropy',
optimizer='adam',
metrics=['accuracy’])
model.fit(X, Y_encoded, epochs=50, batch_size=1)해당 코드가 이해가 잘 되지 않는다면 딥러닝 모델 설계를 참고하라. 간단한 내용이다.
제공되는 데이터 세트
⚫ Scikit-lean 제공 데이터 세트
https://scikit-learn.org/stable/datasets.html
