본 논문은 OOD문제를 self-supervised learning을 통한 representation learning에 기반하여 문제를 해결한다.
We ask the following question: what training information is required to design an effective outlier/out-of-distribution (OOD) detector, i.e., detecting samples that lie far away from the training distribution?
논문의 저자는 다음과 같은 질문을 던진다. 어떤 훈련 정보가 효과적인 OOD를 디자인하는데 필요할까?
즉, 훈련된 분포로부터 멀리 떨어진 샘플들에 대해 탐지하는 것에 필요한 훈련 정보가 무엇이 있을까? 라는 질문이다.
그에 대한 해답으로, 본 논문에서는 SSD 라는 Framework를 제시한다. 이는 라벨이 없는 in-distribution data(unlabeld in-distribution data)로만 학습을 진행한다. 그리고 특징공간(feature space)에서 Mahalanobis distance에 기반한 탐지를 따르는 self-supervised representation learning기법을 사용한다. 다시 말해서, 어떤 distance metric을 통한 self-supervised learning을 통해 OOD를 해결하고자 하였다. 더불어, 두가지 확장 실험을 진행하였는데,
첫번째로 few-shot OOD detection이라고 하는 하나에서 다섯개의 샘플을 각 클래스에서 꺼내 사용하는 방법과
그리고 두번째로, 만약 가능하다면(label이 있다면) 훈련 과정에서 데이터의 label을 포함시킨 framework를 제시한다.
INTRODUCTION
딥러닝의 경우 많은 발전이 있었음에도 불구하고, 훈련 데이터 분포(in-distribution)이 아닌 test inputs이 들어갔을때, 이것이 training distribution에서 나왔는지, 아니면 분포에서 멀리 떨어져 있는 처음 보는 데이터(Out-of-distribution)인지에 대해 구분하는 것이 실패하는 경우가 많이 있다. 이러한 취약점은 많은 outlier detector의 발전에 대한 동기를 부여했다. 그러나 하나의 이해해야만 하는 중요한 질문이 있는데, 어떤 훈련 정보(training information)가 outlier detection에 있어서 주요한 역할을 하는가 ? 에 대한 질문이다. Detector가 세부적인 훈련데이터에 대한 annotation label을 필요로 하거나 훈련과정에서 outliers의 일부가 필요하다거나 하는 주요한 정보들에 대한 것.
결국 논문 저자들이 하고 싶은 말은 만약에 비지도학습이 in-distribution의 주요한 특징들을 담을 수 있다면, 이러한 특징이 없는 outlier의 경우 특징 공간(feature space)상에서 멀리 떨어져 있을 것이라고 주장한다. 따라서 이는 쉽게 구분할 수 있게 된다는 의미이다. 저자들은 학습을 위해 Mahalanobis distance에 기반한 cluster-conditioned framework를 제안한다.
REF:https://darkpgmr.tistory.com/41
REF:https://gaussian37.github.io/ml-concept-mahalanobis_distance/
여기서 마할라노비스 거리(Mahalanobis distance)는 평균과의 거리가 표준편차의 몇 배인지를 나타내는 값이다.
예를들어, 일일 교통량의 평균이 20이고, 표준편차가 3일 경우를 생각하자.
평균적으로 하루에 20대 정도 차가 지나가는데 들쑥 날쑥한 정도가 평균적으로 3이라는 의미이다.
즉, 17대가 지나갈 수도, 23대가 지나갈 수도 있다는 말이다.
만약, 어느날 차가 26대가 지나갔다고 하자. 이 때, Mahalanobis distance는 (26-20)/3 = 2이다.
즉, 표준적인 편차의 2배 정도의 오차가 있는 값이라는 것이다.
Mahalanobis distance는 어떤 값이 얼마나 일어나기 힘든 값인지, 또는 얼마나 이상한 값인지를 수치화하는 한 방법이다. 예를 들어서, 1년 내내 매일 매일 차가 정확히 20대만 지나갔었는데 어느날 보니 차가 21대가 지나갔다. 얼마나 이상한가? 이 경우 Mahalanobis distance는 굉장히 큰 값을 가질 것이다. 그런데, 차가 어느날은 10대, 다음날은 30대, 또 다른날은 24대, ... 이와 같이 들쑥 날쑥한 경우에 21대가 지나간 것은 전혀 이상한 일이 아닐 것이다. 그래서 이 경우 Mahalanobis distance는 굉장히 작은 값을 가진다.
Mahalanobis distance는 어떤 데이터가 가짜 데이터인지, 아니면 진짜 데이터인지를 구분하는 용도로 주로 사용된다. 예를 들어, 일일 교통량에 대한 평균을 내고자 하는데 어느날 갑자기 정말 이상한 데이터가 들어왔다면 이걸 포함해서 평균을 내는 것 보다는 이것은 이상한 놈으로 치고 정상적인 것으로 판단되는 데이터들만 이용해서 평균을 구하는 것이 보다 합리적일 수 있다.
우리가 고등학교 수학(확통) 표준정규분포로 바꾼 후에 z 값을 구하는 것이 바로 Mahalanobis distance를 구하는 과정이다. 그리고 우리가 통상적으로 사용하는 자로 잰 거리를 유클리디언 거리(Euclidean distance)라고 부른다.
(마할라노비스 거리)
단변수에서 z-score를 구하듯이, covariance matrix의 inverse matrix를 곱해서 거리를 잰다. 이렇게 구하게 되면 변수들 간의 correlation등 분포를 고려해서 거리를 재게 된다.
여기에 추가적으로 만약에 일부분의 outlier에 대한 정보를 가지고 있을 경우에, 이 데이터를 활용하게 되면 추가적으로 성능을 향상시킬 수 있다고 주장한다.(당연한 얘기)
KEY CONTRIBUTIONS
SSD for unlabeld data
저자들은 SSD, outlier를 탐지하는 비지도학습 기반 프레임워크를 제안한다. SSD는 현존하는 비지도학습 기반 outlier detector를 큰 폭으로 뛰어넘었으며, 어떤 경우는 지도학습 기반의 프레임워크를 뛰어넘기도 한다.
Extention of SSD
덧붙여, 저자들은 few-shot OOD detection에 대해 제안한다.
들어가기에 앞서서 관련된 연구들이 어떤 것들이 진행되었는지 간단하게 summary 한 것은 다음과 같다.
1. OOD detection with unsupervised detectors
2. OOD detection with supervised learning
3. Access to OOD data at training time
4. In conjunction with supervised training
5. Anomaly detection
이 때 unsupervised OOD detection은 세가지 그룹으로 나뉜다.
(1) Reconstruction-error based detection using Auto-encoders
(2) Classification based detection
(3) Probabilistic based detection
SSD: Self-Supervised Outlier/Out-of-Distribution Detection
이제부터 SSD에 대해 설명해보려고 한다. 먼저 outlier/out-of-distribution 기본 배경에 대해 설명하고 SSD를 설명한다. 마지막으로 SSD를 좀 확장해서 label이 있는 데이터를 일부 사용하는 것도 실험했다고 함.
: input space
: label space
: in-distribution with label
: in-distribution without label
: feature extractor where
, g is parameterized by a shallow neural network, geneally a linear classifier.
Problem Formulation : OOD detection
일단 주어지는 문제의 입력은 다음에서 샘플링 될 것이다.
: in-distribution과 out-of-distribution 중 무작위로 하나의 샘플을 가져올 것.
supervised OOD 방법에 의하면,을 가지고 신경망을 훈련시켜서 OOD detection을 진행한다. (일반적으로 학습된 in-distribution의 confidence값이 out-of-distribution보다 높게 나오기 때문에 이 둘의 차이를 가지고 학습을 진행한다. (논문 참조 : Hendrycks & Gimpel(2017), ODIN(Liang(2018)))
Unsupervised OOD detector는 오직 만을 가지고 학습을 진행한다.
(참고사항)Contrastive self-supervised representation learning
unlabeled training data를 가지고 feature extractor를 훈련시키는 방법 중에는 self-supervsied learning 중 많이 쓰이는 contrastive loss 방법이 있다.
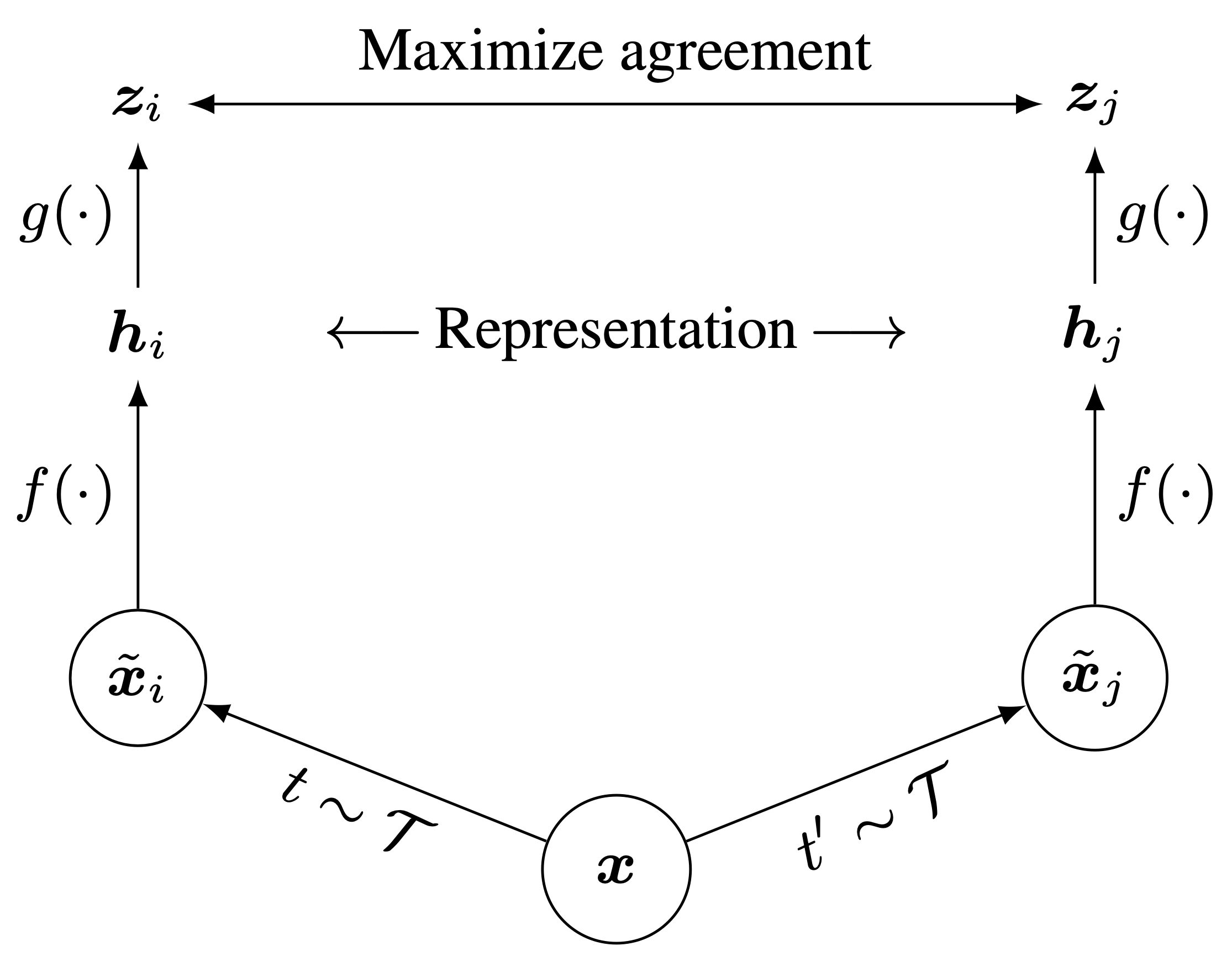
이 방법은 위 그림처럼, 어떤 입력값에 대해 transformation을 적용한다. 만약 N배치사이즈의 이미지를 모두 transformation을 적용하면 N개의 원래 데이터와 N개의 transform된 데이터가 나올 것이다. 여기서, 자기 자신을 변환한 하나의 이미지만을 positive pair로 두고, 그 positive pair를 제외한 모든 이미지들은 멀리 떨어지도록 학습한다(밑에 보이는 temperature scaled Softmax함수에서 positive pairs 둘의 cosine similarity만 loss가 됨.).
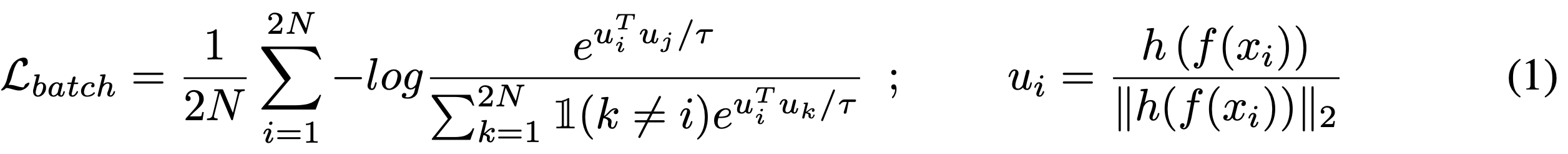
Leveraging contrastive self-supervised training
데이터 라벨이 없는 SSD 는 두가지 스텝으로 구분된다.
1) Training a feature extractor using unsupervised representation learning (위에서 설명한 Contrastive learning을 활용한 SSL을 이용)
2) Developing an effective OOD detector based on hidden features which isn't conditioned on data labels.
Cluster-conditioned detection
데이터의 labels이 없는 상태에서, 저자들은 cluster-conditioned detection method를 개발했다.
- 먼저 훈련 데이터를 m개의 클러스터로 나눈다. (k-means clustring)
- 각각의 클러스터별로 독립적인 features를 으로 나타낸다.
- 를 각 테스트 입력에 대해 구한다.
여기서 은 특징공간에서의 거리 metric이다.
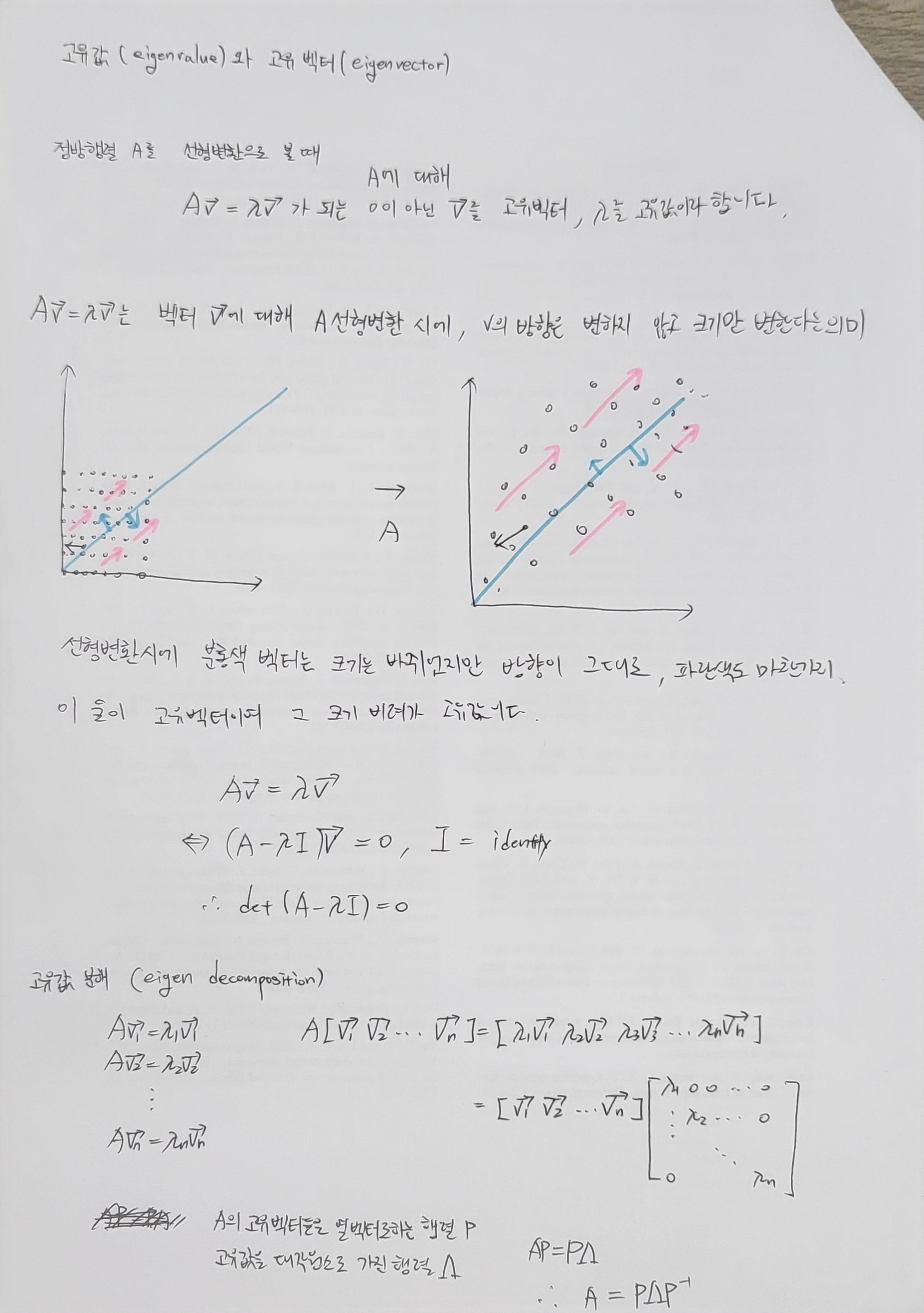
Choice of distnace metric : Mahalanobis distance
위 공식에서 과은 in-distribution training data의 feature 의 sample mean과 sample covariance를 나타낸다.
본 저자들이 마할라노비스 거리를 쓴 이유는 다음과 같다.

고유값 분해를 통해 sample covariance를 분해하면, 과 같고, 이는 eigenspace에서 eigenvalues로 스케일된 유클리디안 거리와 같다.(???)
그니까 eigenspace에서 eigenvalue로 어떤 의미를 줌 즉, 밑에 그림처럼, eigenvalue값들에 대한 정보들이 들어가고, 이는 일반적인 공간의 유클리디안 거리가 아니라, eigenvalue값들에 대한 정보가 들어간 유클리디안 거리이기 때문에, feature space의 분포에 대한 특성을 좀 더 잘 반영한다 라고 생각(.. 완전 그냥 자유 해석)
그래서 만약에 스케일링이 없는 유클리디안 거리면 높은 eigenvalues을 가지는 요소들이 더 큰 weight을 가지고, 이는 차이가 거의 없다. 스케일링된 eigenvalues은 이러한 편향을 제거하고, Mahalanobis distance가 더욱 효과적으로 특징공간에서 outlier을 잘 잡도록 해준다.

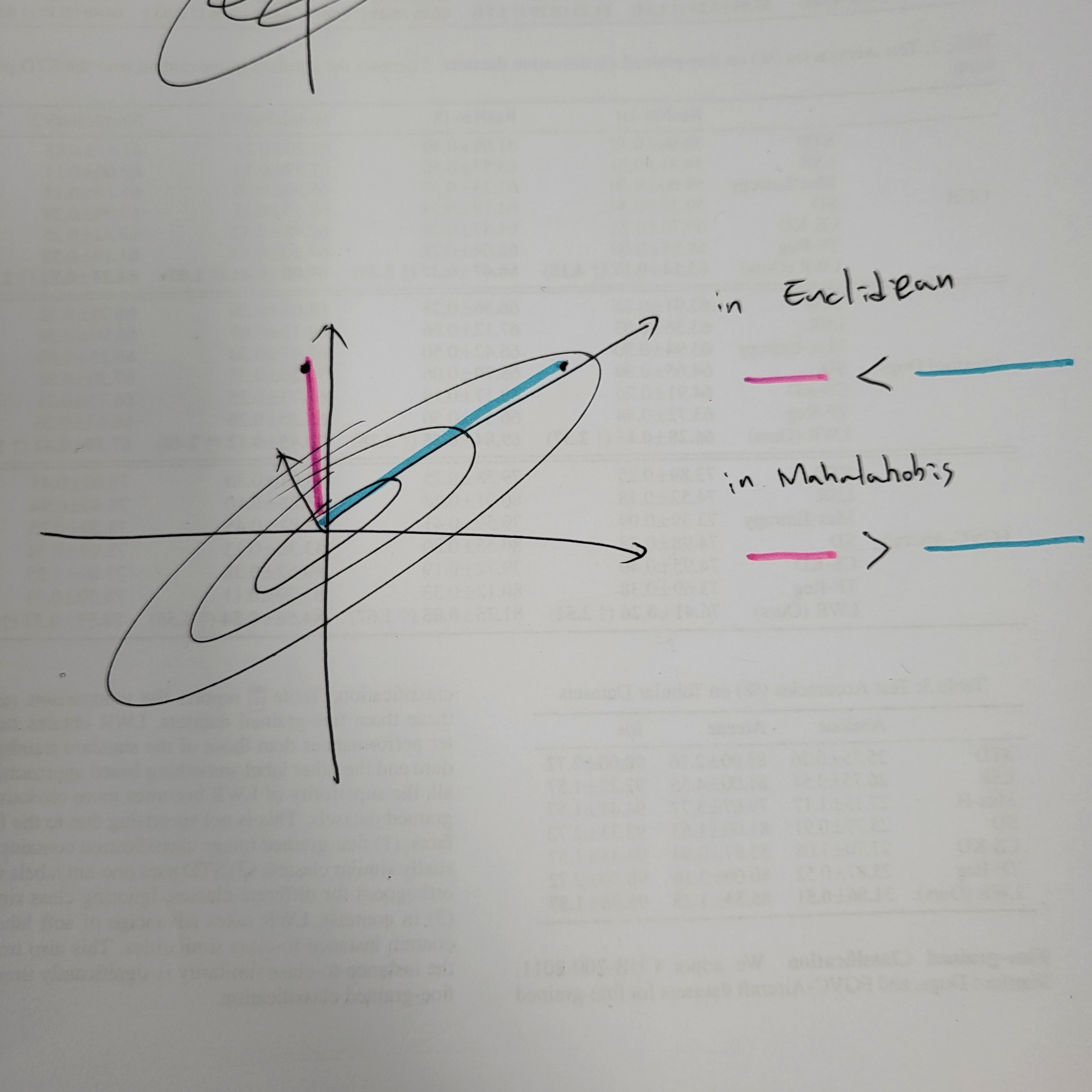
대충보면 이런 느낌일 것 같다.
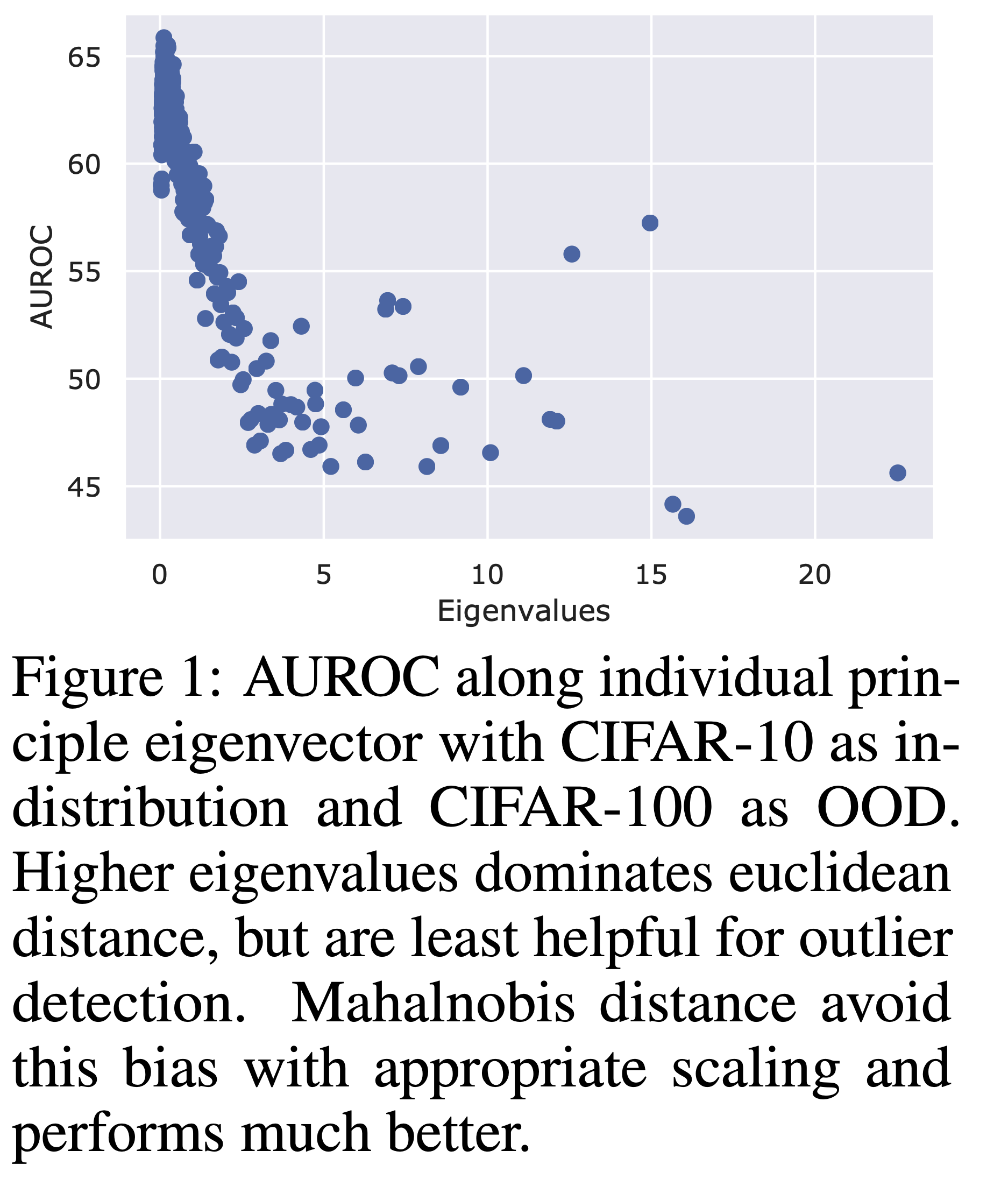
결과를 보더라도, 각각의 principle eigenvector들을 따르는 AUROC값들을 보여주는데, 높은 eigenvalues를 가질수록, euclidean distance와 별반 다를바 없게 되고 이는 별로 outlier distection에 도움이 되지 않는다.(???), 이 실험은 데이터를 각각의 principal eigenvector에 대해 데이터를 구분한 것이다. 즉 각각의 principal eigenvector들 특히 eigenvalues이 높은 값을 가지는 Eigenvector들에 대해 상당히 성능이 낮아짐을 알 수 있다. 이러한 현상은 유클리디안 디스턴스를 사용했을 때 높은 eigenvalues에 높은 weight를 부여하기 때문이며, 이를 해결하기 위해 마할라노비스 거리를 이용한 것 같다.
마할라노비스 거리의 경우 이러한 편향을 없애주고, 적절한 스케일링을 통해 성능이 잘 나온다고 한다.
이 외에도 조금 라벨이 있거나, ood에 대한 정보가 조금 있을 때를 가정하여 실험을 하였는데, 이에 관련된 내용들은 나중에 채워보겠다.

일단 여기서 self-supervised 방법을 제외하고는 보지 않을 것이다.
: return
: return
Parition in training set () and calibration set
# train feature extractor
여기까진 위에서 설명한 그대로다.
Train feature extractor (f) by minimizing over ;
# outlier score
인퍼런스 코드를 분석해보자.
is an outlier if ( threshold at TPR = )
eval_ssd.py
# create model
if args.training_mode in ["SimCLR", "SupCon"]:
model = SSLResNet(arch=args.arch).eval()
elif args.training_mode == "SupCE":
model = SupResNet(arch=args.arch, num_classes=args.classes).eval()
else:
raise ValueError("Provide model class")
model.encoder = nn.DataParallel(model.encoder).to(device)
# load checkpoint
ckpt_dict = torch.load(args.ckpt, map_location="cpu")
if "model" in ckpt_dict.keys():
ckpt_dict = ckpt_dict["model"]
if "state_dict" in ckpt_dict.keys():
ckpt_dict = ckpt_dict["state_dict"]
model.load_state_dict(ckpt_dict)
# dataloaders
train_loader, test_loader, norm_layer = data.__dict__[args.dataset](
args.data_dir,
args.batch_size,
mode=args.data_mode,
normalize=args.normalize,
size=args.size,
)
features_train, labels_train = get_features(
model.encoder, train_loader
) # using feature befor MLP-head
features_test, _ = get_features(model.encoder, test_loader)
print("In-distribution features shape: ", features_train.shape, features_test.shape)
ds = ["cifar10", "cifar100", "svhn", "texture", "blobs"]
ds.remove(args.dataset)
for d in ds:
_, ood_loader, _ = data.__dict__[d](
args.data_dir,
args.batch_size,
mode="base",
normalize=args.normalize,
norm_layer=norm_layer,
size=args.size,
)
features_ood, _ = get_features(model.encoder, ood_loader)
print("Out-of-distribution features shape: ", features_ood.shape)
fpr95, auroc, aupr = get_eval_results(
np.copy(features_train),
np.copy(features_test),
np.copy(features_ood),
np.copy(labels_train),
args,
)
logger.info(
f"In-data = {args.dataset}, OOD = {d}, Clusters = {args.clusters}, FPR95 = {fpr95}, AUROC = {auroc}, AUPR = {aupr}"
)
일단 모델을 만든다. 그 모델은 SimCLR을 가지는(label이 없기 때문) SSLResNet을 가지고 온다.
class SSLResNet(nn.Module):
def __init__(self, arch="resnet50", out_dim=128, **kwargs):
super(SSLResNet, self).__init__()
m, fdim = model_dict[arch]
self.encoder = m()
self.head = nn.Sequential(
nn.Linear(fdim, fdim), nn.ReLU(inplace=True), nn.Linear(fdim, out_dim)
)
def forward(self, x):
return F.normalize(self.head(self.encoder(x)), dim=-1)
여기서 arch는 resnet50을 가져오고, 그때 리스트인 [resnet50, 2048]을 가져온다. resnet50을 encoder로 하고 head부분은 nn.Linear(2048,2048)을 ReLU를 통과시키고, out_dim으로 128차원의 feature를 내뱉는다.
def get_features(model, dataloader, max_images=10 ** 10, verbose=False):
features, labels = [], []
total = 0
model.eval()
for index, (img, label) in enumerate(dataloader):
if total > max_images:
break
img, label = img.cuda(), label.cuda()
features += list(model(img).data.cpu().numpy())
labels += list(label.data.cpu().numpy())
if verbose and not index % 50:
print(index)
total += len(img)
return np.array(features), np.array(labels)get_features는 이미 훈련된 모델(eval_ssd)을 가져와서 feature랑 label을 뽑는 역할을 한다. 그리고 여기서 가져온 feature들을
features_train, features_test, features_odd, labels_train
def get_eval_results(ftrain, ftest, food, labelstrain, args):
"""
None.
"""
# standardize data
ftrain /= np.linalg.norm(ftrain, axis=-1, keepdims=True) + 1e-10
ftest /= np.linalg.norm(ftest, axis=-1, keepdims=True) + 1e-10
food /= np.linalg.norm(food, axis=-1, keepdims=True) + 1e-10
m, s = np.mean(ftrain, axis=0, keepdims=True), np.std(ftrain, axis=0, keepdims=True)
ftrain = (ftrain - m) / (s + 1e-10)
ftest = (ftest - m) / (s + 1e-10)
food = (food - m) / (s + 1e-10)
dtest, dood = get_scores(ftrain, ftest, food, labelstrain, args)
fpr95 = get_fpr(dtest, dood)
auroc, aupr = get_roc_sklearn(dtest, dood), get_pr_sklearn(dtest, dood)
return fpr95, auroc, aupr여기서 feature들을 모두 정규화를 하고, get_scores로 점수를 구한다. (질문: 왜 norm으로 나눈 다음에 평균이랑 분산 구해서 다시 정규화함?)
def get_scores(ftrain, ftest, food, labelstrain, args):
if args.clusters == 1:
return get_scores_one_cluster(ftrain, ftest, food)
else:
if args.training_mode == "SupCE":
print("Using data labels as cluster since model is cross-entropy")
ypred = labelstrain
else:
ypred = get_clusters(ftrain, args.clusters)
return get_scores_multi_cluster(ftrain, ftest, food, ypred)cluster는 10개이기 떄문에 else로 간다.
그러면 training mode가 ssl 이기 때문에, get_clusters를 통해 feature train의 클러스터를 가져오고, 그 예측값을 ypred에 저장한 뒤에, multi-cluster에 해당하는 스코어를 리턴으로 가져온다.
def get_clusters(ftrain, nclusters):
kmeans = faiss.Kmeans(
ftrain.shape[1], nclusters, niter=100, verbose=False, gpu=False
)
kmeans.train(np.random.permutation(ftrain))
_, ypred = kmeans.assign(ftrain)
return ypredget_cluster는 간단한 Kmeans 클러스터를 통해서, 학습하고 feature에 대해 y값들을 다 할당해준다. 그렇게 나온 y pred값은
def get_scores_multi_cluster(ftrain, ftest, food, ypred):
xc = [ftrain[ypred == i] for i in np.unique(ypred)]
din = [
np.sum(
(ftest - np.mean(x, axis=0, keepdims=True))
* (
np.linalg.pinv(np.cov(x.T, bias=True)).dot(
(ftest - np.mean(x, axis=0, keepdims=True)).T
)
).T,
axis=-1,
)
for x in xc
]
dood = [
np.sum(
(food - np.mean(x, axis=0, keepdims=True))
* (
np.linalg.pinv(np.cov(x.T, bias=True)).dot(
(food - np.mean(x, axis=0, keepdims=True)).T
)
).T,
axis=-1,
)
for x in xc
]
din = np.min(din, axis=0)
dood = np.min(dood, axis=0)
return din, dood위에 들어가게되고, feature train, feature test, feature odd와 y_pred값을 넣어주면서, 일단 ftrain이 kmeans로 분리될 때, ypred값에 해당하는 [0,1,2,---,10]클러스터 값들에 해당하는 ftrain들의 값들을 넣어줍니다.
