# EDA
1. seaborn pairplot
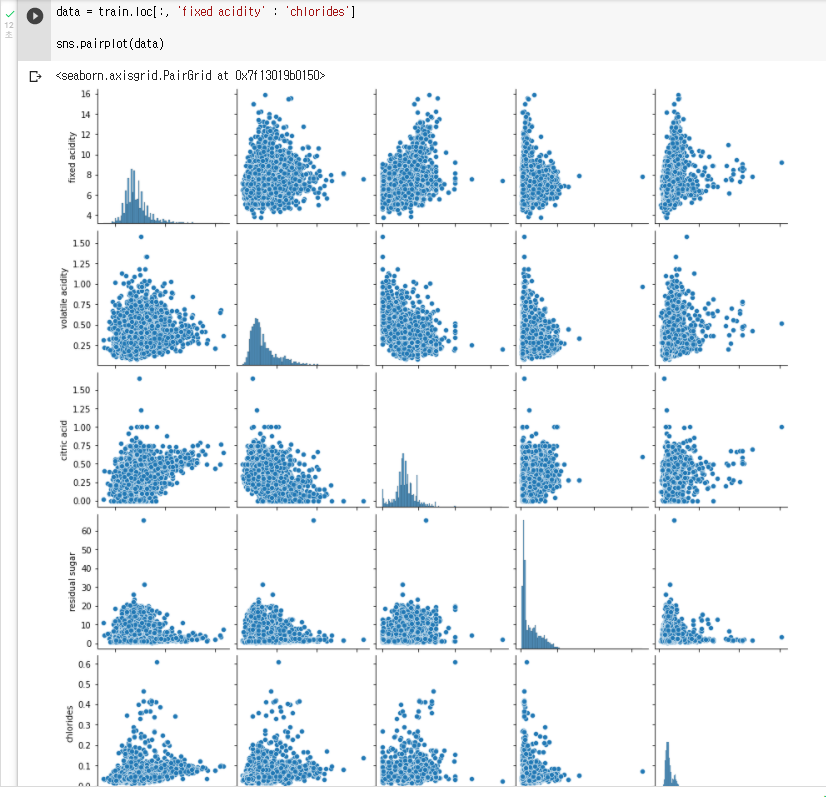
: 3차원 이상의 데이터는 pairplot 함수를 사용해 분포도를 그리면 손쉽게 모든 변수간의 상관관계를 얻을 수 있다.
: pairplot은 grid(격자) 형태로 각 집합의 조합에 대해 히스토그램과 분포도를 그린다.
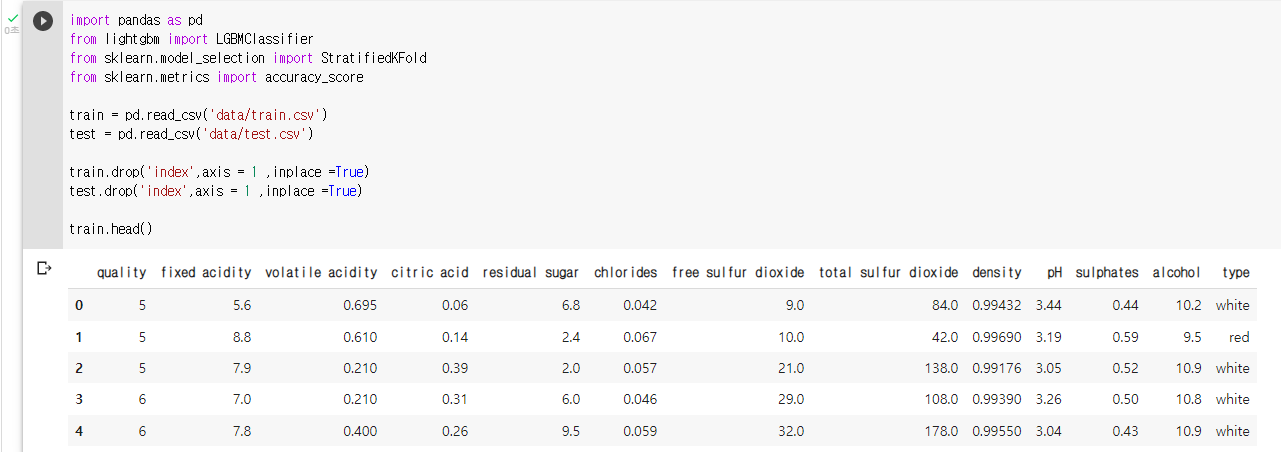
: 데이터 다운로드 링크로 데이터를 코랩에 불러온 뒤 라이브러리 불러오기
: "data"라는 변수에 train의 "fixed acidity"부터 "chlorides"까지의 변수를 저장하기data = train.loc[:, 'fixed acidity' : 'chlorides']: data의 pairplot을 그려보기
sns.pairplot(data)
2. seaborn distplot
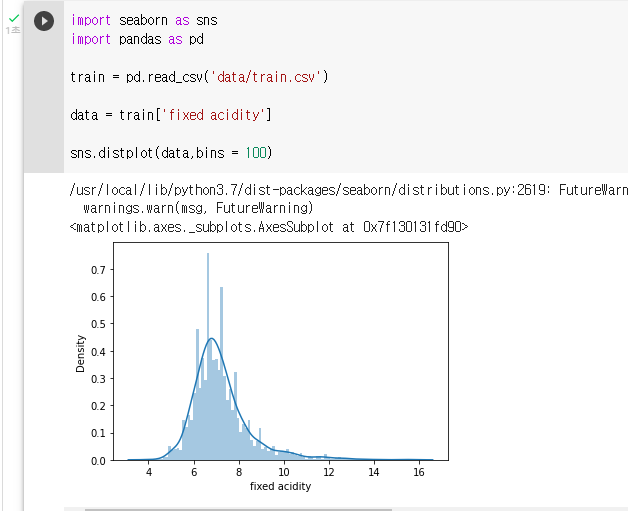
: seaborn의 distplot 함수는 데이터의 히스토그램을 그려주는 함수이다.
: 히스토그램이란 수치형 데이터 분포를 정확하게 표현해주는 시각화 방법이다.
: 변수를 여러 개의 bin으로 자르고(사용자 지정) bin당 관측수를 막대그래프로 표현한다.
: "data"라는 변수에 train의 "fixed acidity" 변수를 저장하기
data = train['fixed acidity']: data의 distplot을 그려보기(bins = 100)
sns.distplot(data,bins = 100)
3. seaborn | heatamp
Heat Map
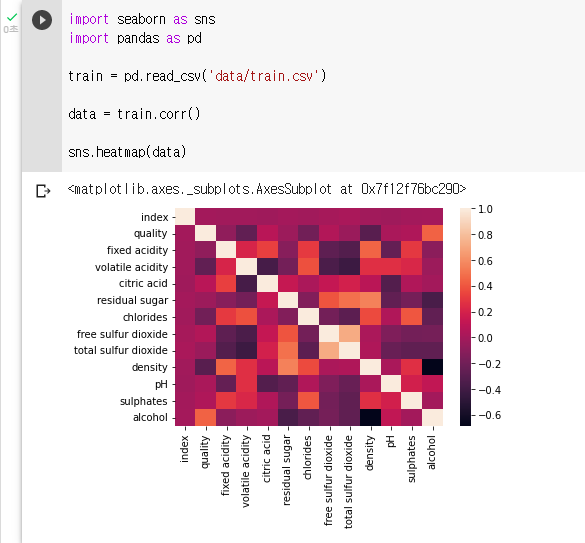
: 두개의 범주형(Categorical) 변수에 대한 반응변수의 크기를 색깔의 변화로 표현하는 것이다.
ex) 매일 온도를 히트맵으로 표현하여 일 별 온도 변화의 추이를 볼 수 있다.
: 데이터 분석 과정에서는 변수별 상관관계를 확인 할때 히트맵 그래프를 많이 사용한다.
: 상관관계를 파악하는 이유는 바로 다중공선성 때문이다.
: 히트맵 그래프를 그릴 변수 지정 (train.corr() )
: corr() 함수는 데이터의 변수간의 상관도를 출력하는 함수 이다.data = train.corr(): seaborn 의 heatmap 함수를 이용해 히트맵 그래프를 그린다.
sns.heatmap(data)
4. 다중공선성 Scatter plot
다중공선성
: 상관관계가 높은 독립변수들이 동시에 모델에 포함될 때 발생한다.
: 만약 두 변수가 완벽하게 다중공선성에 걸려있다면, 같은 변수를 두 번 넣은 것이므로 모델이 결괏값을 추론하는 데 방해가 될 수 있다.
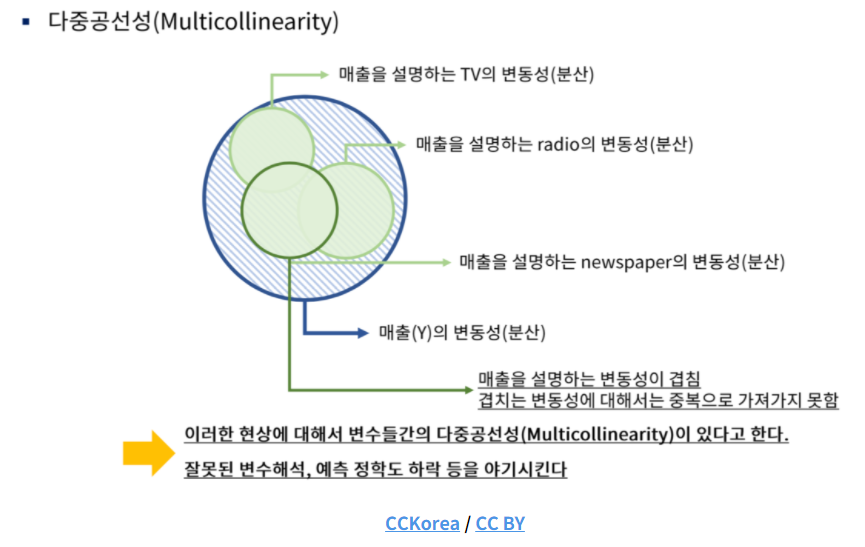
다중공선성 확인
- Scatter plot을 통한 확인
- Heatmap 그래프를 통한 확인
- VIF(Variance Inflation Factors, 분산팽창요인)을 통한 확인
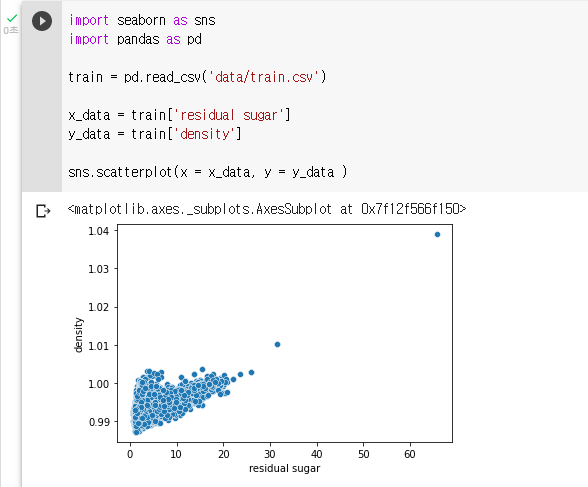
: Scatter Plot(산점도 그래프)는 두 개의 연속형 변수에 대한 관계를 파악하는데 유용하게 사용할 수 있다.
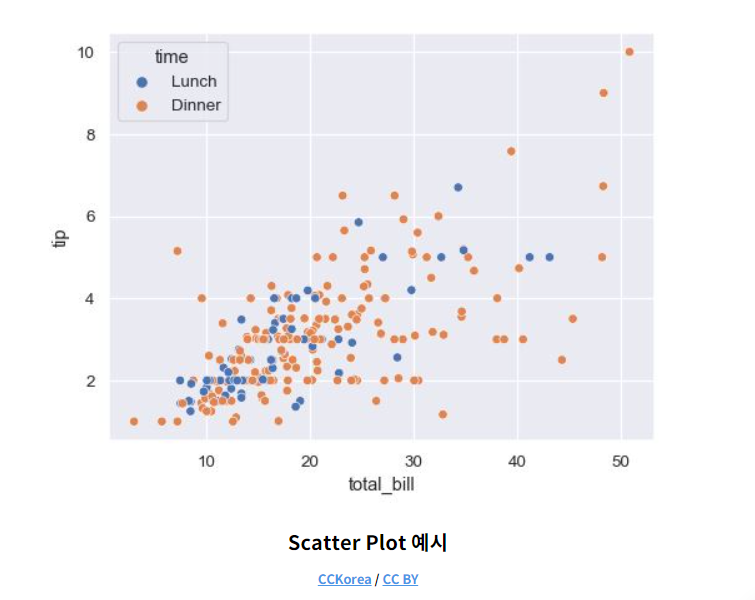 : 예시처럼 x 데이터가 증가함에 따라 y 데이터가 증가하는 경향을 보이는데, 이럴 경우 두 변수의 상관도가 높다고 해석 할 수 있다.
: 예시처럼 x 데이터가 증가함에 따라 y 데이터가 증가하는 경향을 보이는데, 이럴 경우 두 변수의 상관도가 높다고 해석 할 수 있다.
: Scatter Plot을 그릴 변수 지정 (x_data 에는 residual sugar변수, y_data 에는 density 변수)
x_data = train['residual sugar'] y_data = train['density']: seaborn 의 scatterplot함수를 이용해 그래프를 그린다.
sns.scatterplot(x = x_data, y = y_data )
5. 다중공선성 VIF(분산 팽창 요인)
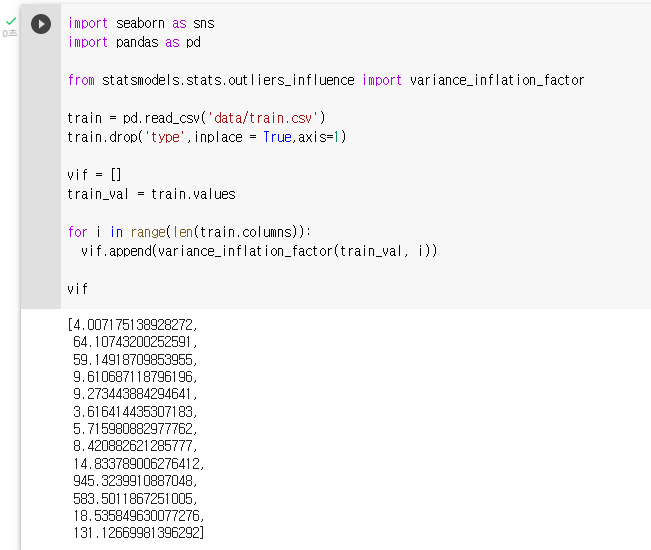
: VIF는 변수간의 다중공선성을 진단하는 수치이며 범위 1부터 무한대이다.
: 통계학에서는 VIF 값이 10이상이면 해당 변수가 다중공선성이 잇는 것으로 판단한다.
: VIF를 구하는 수식은 VIFk = 1 / (1 - Rj2) 이다.
: VIFk(아래첨자) 는 k번째 변수의 VIF 값을 의미하고, Rj2(지수) 는 회귀분석에서 사용하는 결정계수이다.
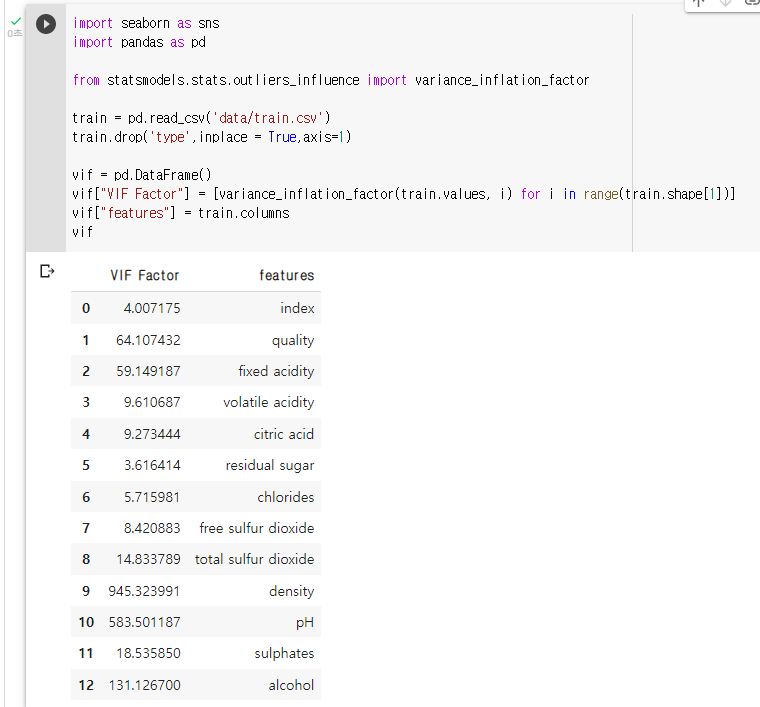
: VIF기능을 제공하는 라이브러리 불러오기 및 범주형 변수 제거(type)
: 결과값을 저장할 VIF라는 이름의 리스트 생성vif = []: values atribution(속성) 이용해 train 데이터의 값만 추출
train_val = train.values: variance_inflation_factor 함수에 인자로 train_val (train 데이터의 값)과 인덱스 번호 지정 - for 반복문 이용
for i in range(len(train.columns)): vif.append(variance_inflation_factor(train_val, i))
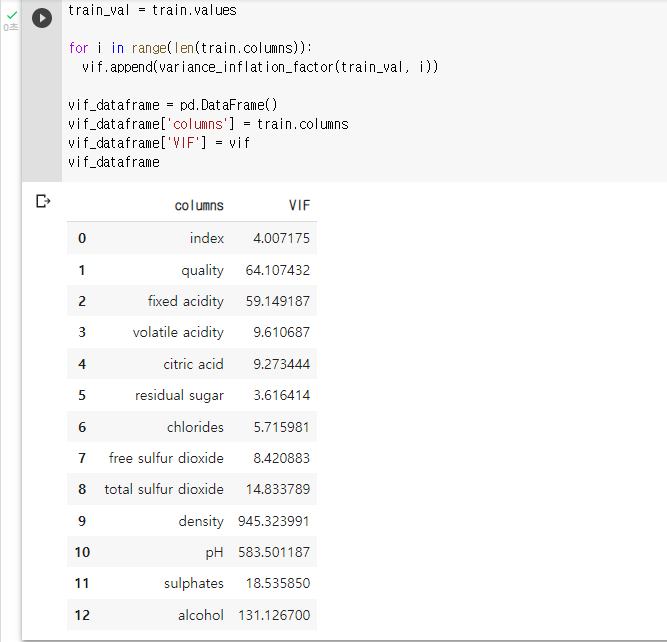
: 데이터 프레임 형식으로 바꿔 가독성 높이기! (보너스 문제)vif_dataframe = pd.DataFrame() vif_dataframe['columns'] = train.columns vif_dataframe['VIF'] = vif vif_dataframe
# 전처리
1. 다중공선성 해결 | 변수 정규화
다중공선성 해결
- 변수 정규화 (ㅇ)
- 변수 제거
- PCA(주성분 분석)
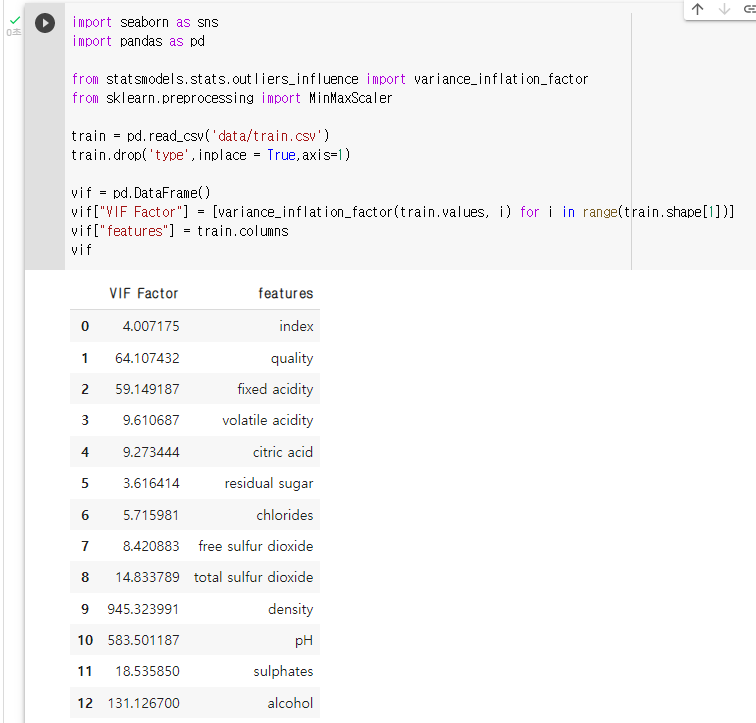
: 정규화를 적용하기 전 분산 팽창 요인(VIF)를 확인하고 정규화를 적용한 후 분산 팽창 요인을 확인해 서로 비교
: sklearn 의 MinMaxScaler 라이브러리 불러오기
: train 데이터의 VIF 계수 출력vif = pd.DataFrame() vif["VIF Factor"] = [variance_inflation_factor(train.values, i) for i in range(train.shape[1])] vif["features"] = train.columns vif
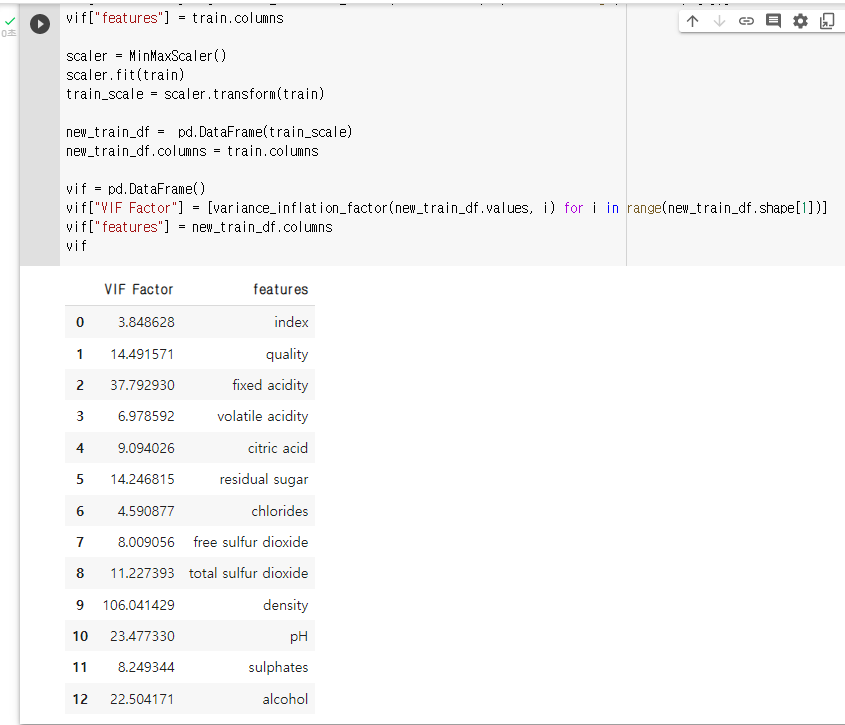
: MinMaxScaler를 통해 변수 변환
: MinMaxScaler를 "scaler"라는 변수에 지정할 것scaler = MinMaxScaler(): "scaler"를 train 으로 학습시키기
scaler.fit(train): "scaler"를 통해 train의 수치들을 변환 시키고 train_scale에 저장하기
train_scale = scaler.transform(train): Sclaer 를 통해 변환된 데이터의 VIF 확인
new_train_df = pd.DataFrame(train_scale) new_train_df.columns = train.columns vif = pd.DataFrame() vif["VIF Factor"] = [variance_inflation_factor(new_train_df.values, i) for i in range(new_train_df.shape[1])] vif["features"] = new_train_df.columns vif
2. 다중공선성 해결 | 변수 제거
다중공선성 해결
- 변수 정규화
- 변수 제거 (ㅇ)
- PCA(주성분 분석)
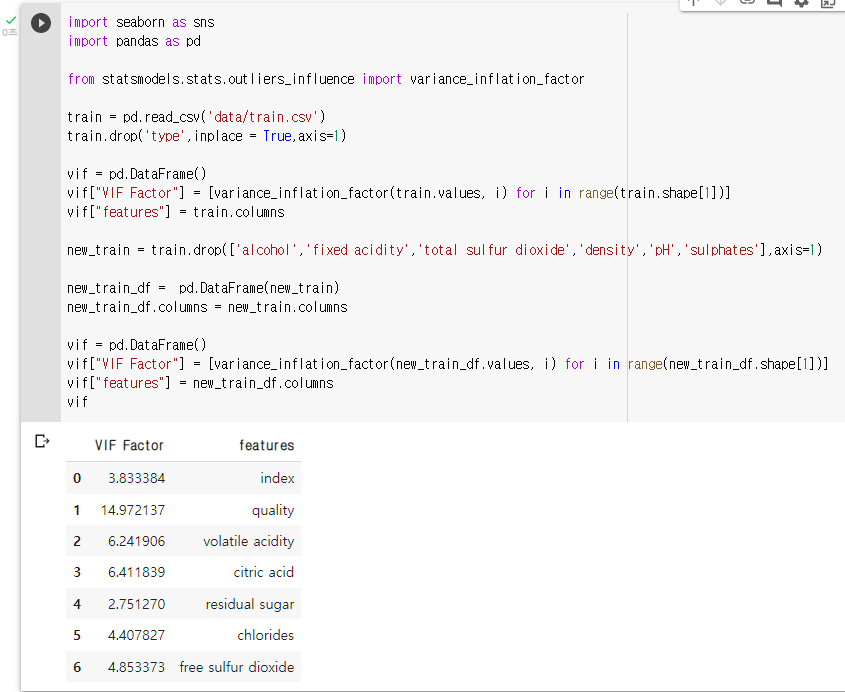
: VIF 기준 10이상의 변수들을 제거
: train 데이터의 VIF 계수 출력
vif = pd.DataFrame() vif["VIF Factor"] = [variance_inflation_factor(train.values, i) for i in range(train.shape[1])] vif["features"] = train.columns vif
: 종속변수인 quality를 제외 한 vif 10이상 변수 제거new_train = train.drop(['alcohol','fixed acidity','total sulfur dioxide','density','pH','sulphates'],axis=1): VIF 10이상의 변수 제거 후 VIF 다시 확인
new_train_df = pd.DataFrame(new_train) new_train_df.columns = new_train.columns vif = pd.DataFrame() vif["VIF Factor"] = [variance_inflation_factor(new_train_df.values, i) for i in range(new_train_df.shape[1])] vif["features"] = new_train_df.columns vif
3. 다중공선성 해결 - PCA (1)
다중공선성 해결
- 변수 정규화
- 변수 제거
- PCA(주성분 분석) (ㅇ)
: PCA 를 통한 해결
: PCA를 이해하기 위해서는 먼저 차원축소의 개념을 이해해야 한다.
차원 축소
: 많은 피처로 구성된 다차원 데이터 셋의 차원을 축소해 새로운 차원의 데이터 셋을 생성하는 것이다.
: 일반적으로 차원이 증가할수록 데이터 포인트 간의 거리가 기하급수적으로 멀어지게 되고, 희소(sparse)한 구조를 가지게 된다.
: 수백 개 이상의 피처로 구성된 데이터 셋의 경우 상대적으로 적은 차원에서 학습된 모델보다 예측 신뢰도가 떨어진다.
: 또한 피처가 많은 경우 개별 피처 간의 상관관계가 높을 가능성이 크다.
: 선형 회귀와 같은 선형 모델에서는 입력 변수 간의 상관관계가 높을 경우, 이로 인한 다중 공선성 문제로 모델의 예측 성능이 떨어진다.
: 그리고 수십 개 이상의 피처가 있는 데이터의 경우 이를 시각적으로 표현해 데이터의 특성을 파악하기는 불가능 하다.
: 이 경우 3차원 이하의 차원 축소를 통해서 시각적으로 데이터를 압축해서 표현할 수 있다.
: 또한 차원 축소를 할 경우 학습 데이터의 크기가 줄어들어서 학습에 필요한 처리 능력도 향상 시킬 수 있다.
: 일반적으로 차원 축소는feature selection 과 feature extraction으로 나눌 수 있다.
: feature selection 은 말 그대로 특정 피처에 종속성이 강한 불필요한 피처는 아예 제거하고, 데이터의 특징을 잘 나타내는 주요 피처만 선택하는 것이다.
: feature extraction은 기존 피처를 저 차원의 중요 피처로 압축해서 추출하는 것이다.
: 새롭게 추출된 중요 특성은 기존의 피처가 압축된 것이므로 기존의 피처와는 완전히 다른 값이 된다.
: 다중공선성을 해결하는 방법 중 하나 인 PCA의 경우 feature extraction의 기법 중 하나이다.
feature Extraction
: 기존 피처를 단순 압축이 아닌, 함축적으로 더 잘 설명할 수 있는 또 다른 공간으로 매핑 해 추출하는 것이다.
: 학생을 평가하는 다양한 요소로 모의고사 성적, 종합 내신 성적 , 수능성적 등 관련된 여러 가지 피처로 되어있는 데이터 셋이라면 이를 학업 성취도, 커뮤니케이션 능력,문제 해결력과 같은 더 함축적인 요약 특성으로 추출할 수 있다.
4. 다중공선성 해결 - PCA (2)
PCA
: 차원 축소 기법 중 가장 대표적인 기법인 PCA는 여러 변수 간에 존재하는 상관관계를 이용해 이를 대표하는 주성분을 추출해 차원을 축소하는 기법이다.
: 기존 데이터의 정보 유실 최소화를 위해 가장 높은 분산을 가지는 데이터 축을 찾아 해당 축으로 차원을 축소한다.
: 키와 몸무게 2개의 피처를 가지고 있는 데이터셋이 다음과 같이 구성 되어 있다고 가정
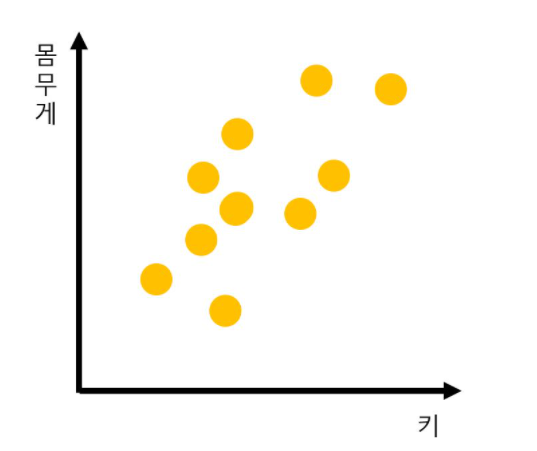 : 이 2개의 피처를 한개의 주성분을 가진 데이터 셋으로 차원축소하는 과정은 다음과 같다.
: 이 2개의 피처를 한개의 주성분을 가진 데이터 셋으로 차원축소하는 과정은 다음과 같다.
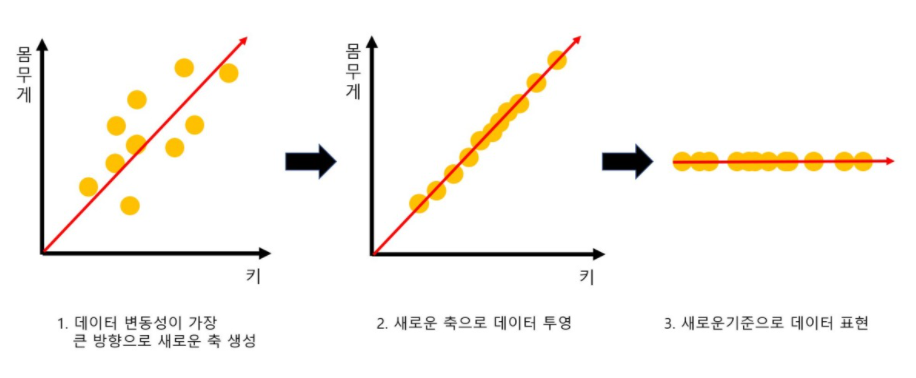 : PCA는 제일 먼저 가장 큰 데이터 변동성을 기반으로 첫 번째 벡터 축을 생성한다.
: PCA는 제일 먼저 가장 큰 데이터 변동성을 기반으로 첫 번째 벡터 축을 생성한다.
: 두 번째 축은 이 벡터 축에 직각이 되는 벡터(직교 벡터)를 축으로 한다.
: 세 번째 축은 다시 두 번째 축과 직각이 되는 벡터를 설정하는 방식으로 축을 생성한다.
: 이렇게 생성된 벡터 축에 원본 데이터를 투영하면 벡터 축의 개수 만큼의 차원으로 원본 데이터가 차원 축소 된다.
: PCA는 많은 속성으로 구성된 원본 데이터를 그 핵심을 구성하는 데이터로 압축한 것이다.
5. 다중공선성 해결 - PCA (3)
실습 순서
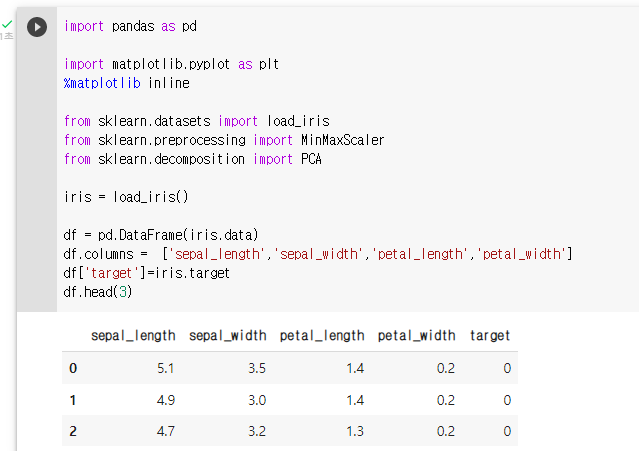
- iris 데이터 로딩
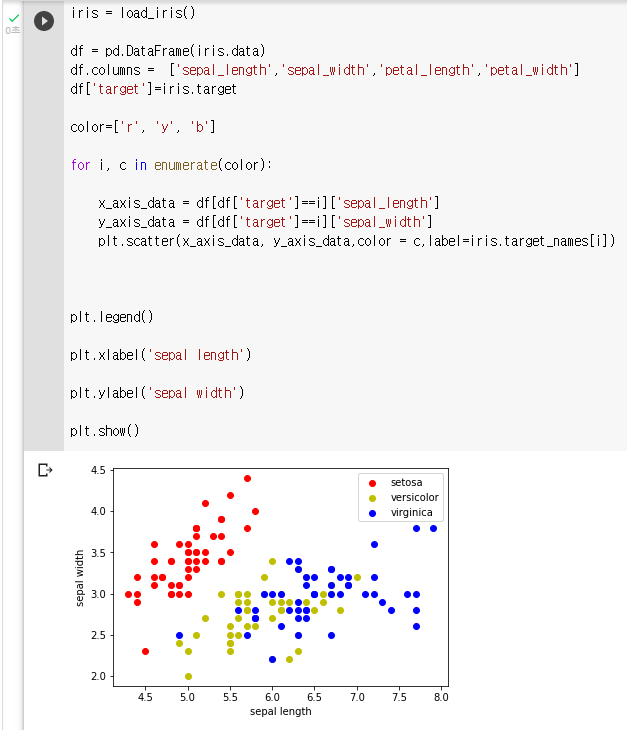
- x축 : sepal length y축 : sepal width로 하여 품종 데이터 분포 시각화
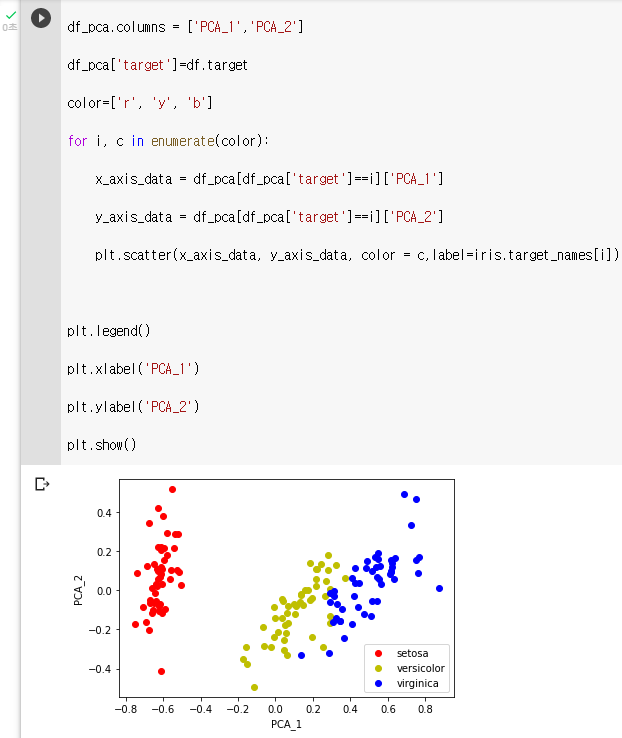
- PCA를 이용해 차원 축소 후 변환된 데이터 셋을 2차원 상에서 시각화.
: 데이터 로딩
: setosa는 빨간색, versicolor는 노란색, virginica는 파란색color=['r', 'y', 'b']: setosa의 target 값은 0, versicolor는 1, virginica는 2
: 각 target 별로 다른 색으로 scatter plotfor i, c in enumerate(color): x_axis_data = df[df['target']==i]['sepal_length'] y_axis_data = df[df['target']==i]['sepal_width'] plt.scatter(x_axis_data, y_axis_data,color = c,label=iris.target_names[i]) plt.legend() plt.xlabel('sepal length') plt.ylabel('sepal width') plt.show()
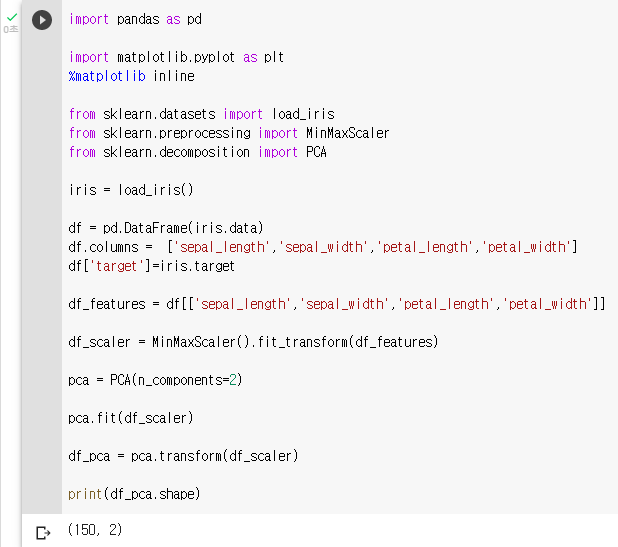
: Target 값을 제외한 모든 속성 값을 MinMaxScaler를 이용하여 변환
: 'sepal_length','sepal_width','petal_length','petal_width'df_features = df[['sepal_length','sepal_width','petal_length','petal_width']] df_scaler = MinMaxScaler().fit_transform(df_features): PCA를 이용하여 4차원 변수를 2차원으로 변환
pca = PCA(n_components=2): fit( )과 transform( ) 을 호출하여 PCA 변환 / 데이터 반환
pca.fit(df_scaler) df_pca = pca.transform(df_scaler) print(df_pca.shape)
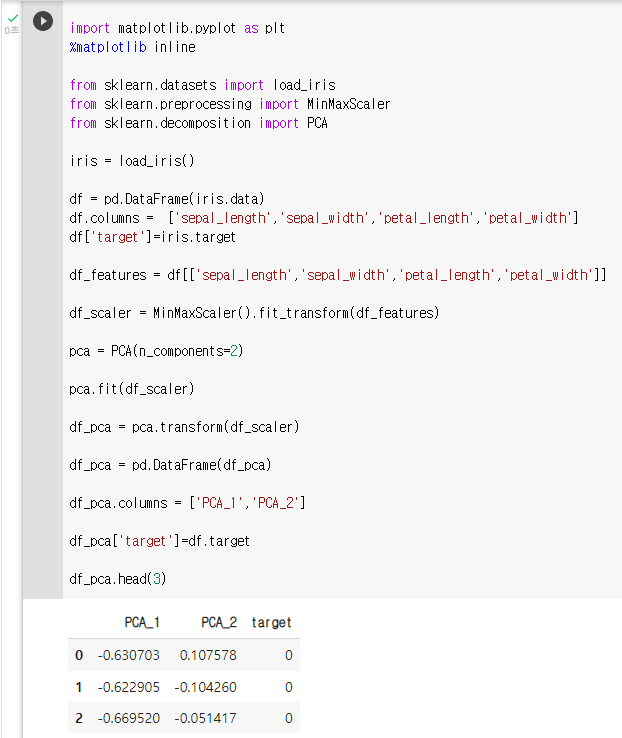
: PCA 변환된 데이터의 컬럼명을 각각 PCA_1, PCA_2로 지정df_pca = pd.DataFrame(df_pca) df_pca.columns = ['PCA_1','PCA_2'] df_pca['target']=df.target df_pca.head(3)
: setosa는 빨간색, versicolor는 노란색, virginica는 파란색color=['r', 'y', 'b']: setosa의 target 값은 0, versicolor는 1, virginica는 2
: 각 target 별로 다른 색으로 scatter plotfor i, c in enumerate(color): x_axis_data = df_pca[df_pca['target']==i]['PCA_1'] y_axis_data = df_pca[df_pca['target']==i]['PCA_2'] plt.scatter(x_axis_data, y_axis_data, color = c,label=iris.target_names[i]) plt.legend() plt.xlabel('PCA_1') plt.ylabel('PCA_2') plt.show()
6. 연속형 변수 변환 (1)
: 머신러닝 모델링을 하다 보면 제한된 변수로 성능을 끌어 올리는 데는 한계가 있다.
: 정형 데이터의 경우 데이터 증강은 제한적이기 때문에 효율적인 파생 변수를 추가하는 것이 중요하다.
연속형 변수를 범주형 변수로 변환
- 수치 범위 구간을 직접 지정해 레이블링 하기 (ㅇ)
- 판다스의 cut() 함수로 레이블링 하기
: train 데이터 확인
train.head()
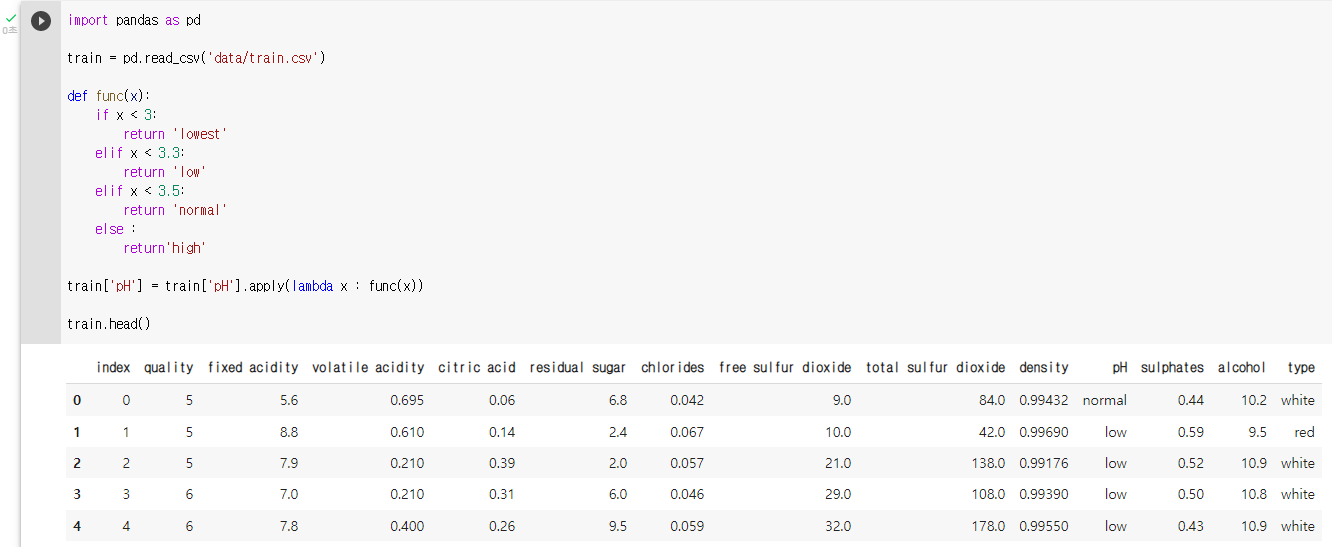
: train 데이터의 pH 변수를 구간이 4개인 범주형 변수로 변환
: pH < 1 -> lowest
: 1<= pH < 2 -> low
: 2 <= pH < 3-> normal
: 3 <= pH -> highdef func(x): if x < 3: return 'lowest' elif x < 3.3: return 'low' elif x < 3.5: return 'normal' else : return'high' train['pH'] = train['pH'].apply(lambda x : func(x)): 변환 후 데이터 확인
train.head()
7. 연속형 변수 변환 (2)
연속형 변수를 범주형 변수로 변환
- 수치 범위 구간을 직접 지정해 레이블링 하기
- 판다스의 cut() 함수로 레이블링 하기 (ㅇ)
: 직접 수치 범위를 나눌 경우 조금 더 세밀하게 조정가능 하다는 장점이 있지만, 여러 변수에 한번에 적용하기에는 어렵고 각각의 변수에 맞는 범위를 지정하기에는 많은 시간이 소요 된다.
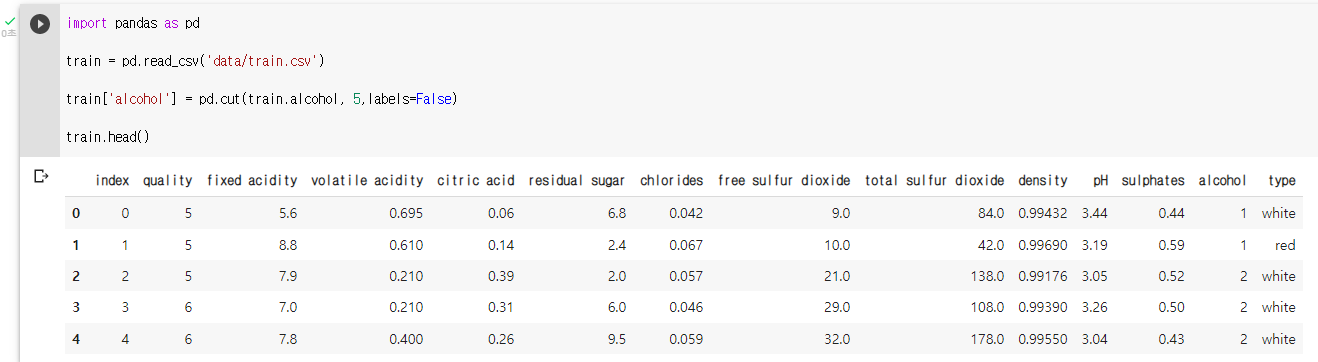
train 데이터의 alcohol 변수를 구간이 5개인 범주형 변수로 변환
train['alcohol'] = pd.cut(train.alcohol, 5,labels=False)
8. Polynomial Features (1)
: Polynomial Features 라는 라이브러리를 이용해 파생 변수를 생성
: PolynomialFeatures라이브러리는 sklearn에 내장되어 있는 라이브러리이다.
: 이 라이브러리를 이용하면 현재 데이터를 다항식 형태로 변환 시킬 수 있다.
: 데이터에 x1,x2 변수가 있다 하면 PolynomialFeatures 라이브러리를 이용해
1, x1, x2, x1^2, x1*x2, x2^2 로 간단하게 변환 시킬 수 있다.
: numpy 라이브러리 호출
: PolynomialFeatures 라이브러리 호출
: 임의 데이터 생성from sklearn.preprocessing import PolynomialFeatures import numpy as np X = np.arange(6).reshape(3, 2) df = pd.DataFrame(X) df.columns = ['x_1','x_2'] df: poly_features정의 , 차원은 2로 설정
poly_features = PolynomialFeatures(degree=2): fit_transform 메소드를 통해 df 데이터 변환
df_poly = poly_features.fit_transform(df): PolynomialFeatures로 변환 된 데이터를 데이터 프레임 형태로 변환 후 df_poly로 지정
df_poly = pd.DataFrame(df_poly)
: df_poly의 컬럼을 1,x1,x2,x1^2,x1*x2,x2^2 로 변경df_poly.columns = ['1','x1','x2','x1^2','x1*x2','x2^2']
9. Polynomial Features (2)
: 와인 품질 분류 데이터에 Polynomial Features를 적용해 보고 decision tree 모델을 이용해 품질 분류 하기
실습 순서
- train 데이터 변환
- 모델 학습 (Decision Tree)
- test 데이터 변환
- 추론
- 정답 파일 생성
: 라이브러리 및 데이터 불러오기
: 범주형 변수, index 변수 제거
: 데이터 확인train.head()
: train 데이터를 PolynomialFeatures 를 이용하여 변환, 차원은 2로 설정poly_features = PolynomialFeatures(degree=2): 와인 품질 기준인 quality 변수를 제외한 나머지 변수를 포함한 데이터 변환
df = train.drop('quality',axis = 1): fit_transform 메소드를 통해 데이터 변환
df_poly = poly_features.fit_transform(df): PolynomialFeatures로 변환 된 데이터를 데이터 프레임 형태로 변환
df_poly = pd.DataFrame(df_poly): DecisionTreeClassifier 모델을 변환된 train 데이터로 학습
from sklearn.tree import DecisionTreeClassifier model = DecisionTreeClassifier() model.fit(df_poly,train['quality']): test 데이터 변환
: 차원은 2로 설정poly_features = PolynomialFeatures(degree=2): fit_transform 메소드를 통해 데이터 변환
test_poly = poly_features.fit_transform(test): PolynomialFeatures로 변환 된 데이터를 데이터 프레임 형태로 변환
test_poly = pd.DataFrame(test_poly): 결괏값 추론
pred = model.predict(test_poly): 정답 파일 생성
submission = pd.read_csv('data/sample_submission.csv') submission['quality'] = pred submission.to_csv('poly.csv',index = False)
# 모델링
1. XGBoost 개념
XGBoost란?
: XGBoost는 Extreme Gradient Boosting의 약자이다.
: Boosting 기법을 이용하여 구현한 알고리즘은 Gradient Boost 가 대표적이고 알고리즘을 병렬 학습이 지원되도록 구현한 라이브러리가 XGBoost 이다.
: Regression, Classification 문제를 모두 지원하며, 성능과 자원 효율이 좋아서, 인기 있게 사용되는 알고리즘이다.
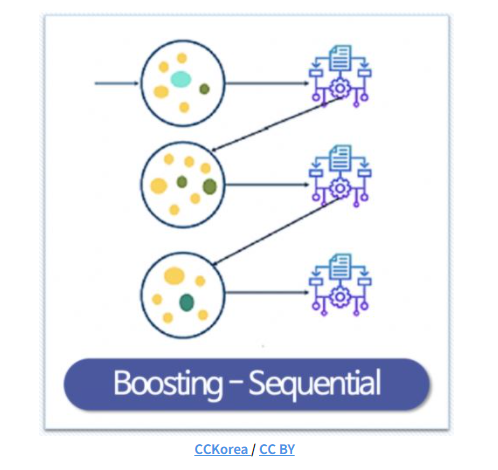
Boosting이란?
: 여러개의 성능이 높지 않은 모델을 조합해서 사용하는 앙상블 기법중 하나이다.
: 성능이 낮은 예측 모형들의 학습 에러에 가중치를 두고, 순차적으로 다음 학습 모델에 반영하여 강한 예측모형을 만든다.
: 아래 그림은 boosting 모델의 학습 예시이다.
XGBoost의 장점
- 기존 boosting 모델 대비 빠른 수행시간(병렬 처리)
- 과적합 규제 지원(Regularization)
- 분류와 회귀 task 에서 높은 예측 성능
- Early Stopping(조기 종료) 기능 제공
- 다양한 옵션을 제공해 Customizing이 용이
- 결측치를 내부적으로 처리 함
2. XGBoost 실습
: 라이브러리 및 데이터 불러오기
import pandas as pd from xgboost import XGBClassifier train = pd.read_csv('data/train.csv') test = pd.read_csv('data/test.csv') train.drop('index',axis = 1 ,inplace =True) test.drop('index',axis = 1 ,inplace =True): 데이터 확인
train.head()
: 원핫 인코딩 (pd.get_dummies())train_one = pd.get_dummies(train) test_one = pd.get_dummies(test): 모델 정의
model = XGBClassifier(): 모델 학습, X 는 train에서 quality 를 제외한 모든 변수, y 는 train의 qulity 변수
X = train_one.drop('quality',axis= 1) y = train_one['quality']: fit 메소드를 이용해 모델 학습
model.fit(X,y): predict 메소드와 test_one 데이터를 이용해 품질 예측
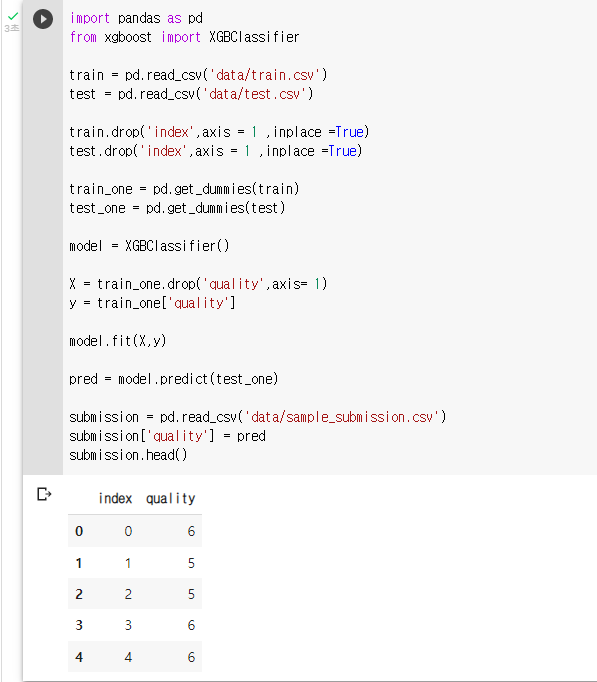
pred = model.predict(test_one): sample_submission.csv 파일을 불러와 예측된 값으로 채워 주기
submission = pd.read_csv('data/sample_submission.csv') submission['quality'] = pred submission.head()
: 정답파일 내보내기submission.to_csv('xgb_pred.csv',index = False)
3. LightGBM 개념
: 머신러닝에서 부스팅 알고리즘은 오답에 가중치를 더하면서 학습을 진행하는 알고리즘이다.
: 그 중 Gradinet Boosting Machine(GBM)은 가중치를 경사하강법(gradint boosting)으로 업데이트 했다.
: XGBoost는 GBM의 단점을 보완한 알고리즘이다.
: xgboost의 여전히 속도면에서는 조금 느리다는 단점을 보완해주기위해 탄생한 것이 LightGBM(LGBM) 이다.
LGBM의 특징
: LGBM은 기존의 gradient boosting 알고리즘과 다르게 동작된다.
: 기존 boosting 모델들은 트리를 level-wise 하게 늘어나는 방법을 사용한 반면, LGBM은 leaf wise(리프 중심) 트리 분할을 사용 한다.
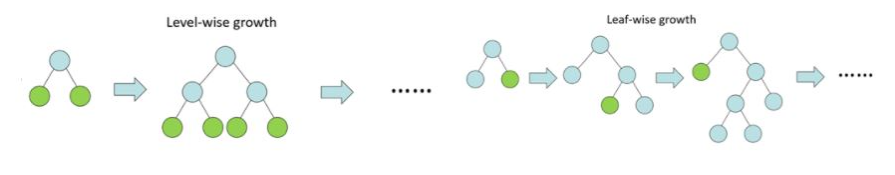 : leaf-wise 의 장점은 속도가 빠르다는 것이 가장 큰 장점이다.
: leaf-wise 의 장점은 속도가 빠르다는 것이 가장 큰 장점이다.
: 데이터 양이 많아지는 상황에서 빠른 결과를 얻는데 시간이 점점 많이 걸리고 있는데, Light GBM은 큰 사이즈의 데이터를 다룰 수 있고 실행시킬 때 적은 메모리를 차지한다.
LGBM의 장점
- 대용량 데이터 처리
- 효율적인 메모리 사용
- 빠른 속도
- GPU 지원
LGBM의 단점
: Light GBM은 Leaf-wise growth로 과적합의 우려가 다른 Tree 알고리즘 대비 높은 편이다.
: 데이터의 양이 적을 경우 Overfiitng(과적합)에 취약한 면이 있어 데이터 양의 적을 경우 사용을 자제하는 것이 좋다.
4. LightGBM 실습
: Light GBM을 이용해 와인 품질 분류를 진행한다.
: 데이터 확인
train.head()
: 원핫 인코딩 (pd.get_dummies())train_one = pd.get_dummies(train) test_one = pd.get_dummies(test): 모델 정의
model = LGBMClassifier(): 모델 학습, X 는 train에서 quality 를 제외한 모든 변수, y 는 train의 qulity 변수
X = train_one.drop('quality',axis= 1) y = train_one['quality']: fit 메소드를 이용해 모델 학습
model.fit(X,y): predict 메소드와 test_one 데이터를 이용해 품질 예측
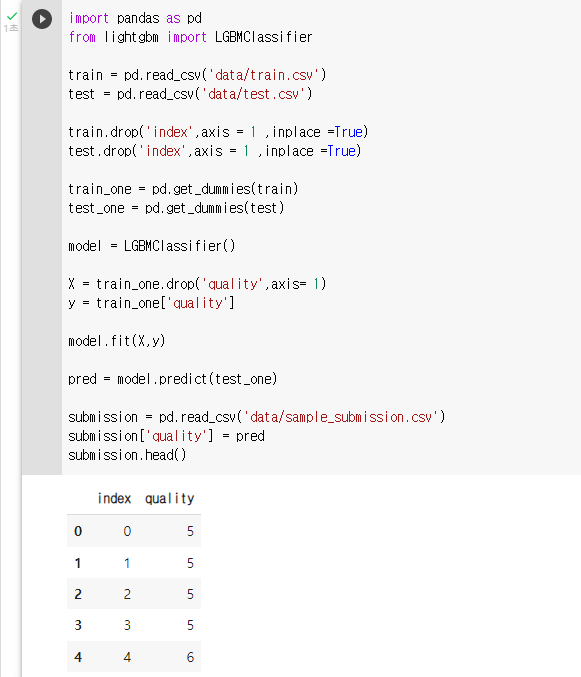
pred = model.predict(test_one): sample_submission.csv 파일을 불러와 예측된 값으로 채워 주기
submission = pd.read_csv('data/sample_submission.csv') submission['quality'] = pred submission.head()
: 정답파일 내보내기submission.to_csv('lgbm_pred.csv',index = False)
5. stratified k-fold 정의
: k-fold 교차검증에 대해 간단히 설명하면 학습 데이터 셋을 학습 데이터와 검증 데이터로 나눠 반복해서 검증 및 평가 하는 것을 의미한다.
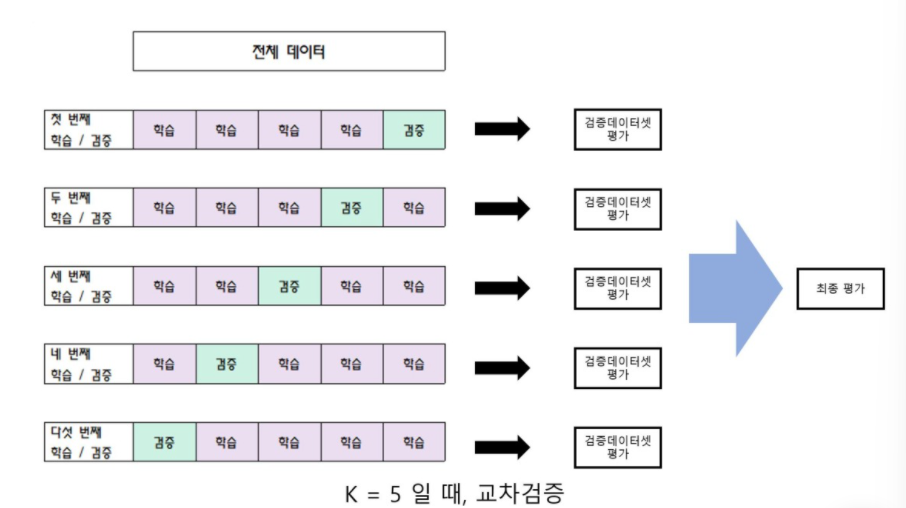
k-fold의 문제점
: k-fold의 경우 데이터 셋을 일정한 간격으로 잘라서 사용한다.
: target의 비율이 일정하지 않게 테스트 셋에 들어 갈 수 있다.
: 만약 target이 0, 1, 2 세가지로 이뤄져 있는데, 이 상황에서 데이터를 잘라서 학습 할 때 0, 1만 답으로 가지고 있는 학습데이터를 가지고 학습을 했을때는 당연하게도 모델은 2라는 답을 도출 할 수 없을 것이다.
: 마찬가지로 1, 2만 가지고 학습을 진행한다면 0이라는 답을 도출 할 수 없을 것이다. 이러한 점이 k-fold의 치명적인 문제점이다.
stratified k-fold
: k-fold의 문제점인 target 데이터의 비율을 일정하게 유지 하지 못하는 것을 일정 하게 유지하며, 교차 검증을 진행 하는 것이다.
: stratified k-fold 와 Light GBM을 이용해 와인 품질 분류를 실습한다.
: 라이브러리 및 데이터 불러오기
: 데이터 확인train.head()
: 원핫 인코딩 (pd.get_dummies())train_one = pd.get_dummies(train) test_one = pd.get_dummies(test): StratifiedKFold라이브러리를 이용해 5개의 fold로 나눔
skf = StratifiedKFold(n_splits = 5) X = train_one.drop('quality',axis = 1) y = train_one['quality'] cnt = 1 acc = 0 for train_idx, valid_idx in skf.split(X,y): train_data = train_one.iloc[train_idx] valid_data = train_one.iloc[valid_idx]: 모델 정의
model = LGBMClassifier(): train_X 는 train_data에서 quality 를 제외한 모든 변수
: train_y 는 train_data의 qulity 변수train_X = train_data.drop('quality',axis= 1) train_y = train_data['quality']: fit 메소드를 이용해 모델 학습
model.fit(train_X,train_y) valid_X = valid_data.drop('quality',axis = 1) valid_y = valid_data['quality']: predict 메소드와 valid_X 데이터를 이용해 품질 예측
pred = model.predict(valid_X): 모델 정확도 출력
print(cnt," 번째 모델 정확도 : " ,accuracy_score(pred,valid_y)) acc += accuracy_score(pred,valid_y) cnt+=1 print('모델 정확도 평균 : ',acc/5)
6. stratified k-fold 실습
: Stratified k-fold 란, k-fold의 문제점인 target 데이터의 비율을 일정 하게 유지하며, 교차 검증을 진행 하는 것이다.
7. Voting Classifier 정의
: Voting Classifier란 여러개의 모델을 결합하여 더 좋은 예측 결과를 도출 하는 앙상블 기법 중 하나이다.
: Voting Classifier에는 hard voting 방법과 soft voting 방법이 있다.
Hard Voting
: Majority Voting이라고도 한다.
: 각각의 모델들이 결과를 예측하면 각 모델의 예측을 모아 다수결 투표로 최종 예측 결과를 선정하는 방식이다.
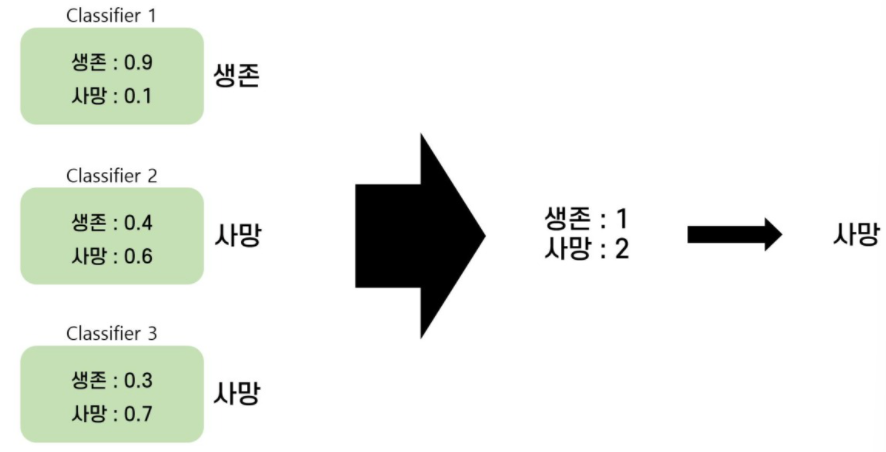 : 첫 번째 분류기는 0.9의 확률로 생존을 선택
: 첫 번째 분류기는 0.9의 확률로 생존을 선택
: 두 번째 분류기는 0.6의 확률로 사망을 선택
: 세 번째 분류기는 0.7의 확률로 사망을 선택
: 생존을 선택 한 모델이 1개, 사망을 선택한 모델이 2개 이기 때문에 Voting Classifier은 최종적으로 사망을 선택하게 된다.
Soft Voting
: Probability Voting이라고 한다.
: 각 모델들이 예측한 결과값의 확률을 합산해 최종 예측 결과를 선정한다.
: 단순히 개별 분류기의 예측 결과만을 고려하지 않고 높은 확률값을 반환하는 모델의 비중을 고려할 수 있기 때문에 Hard Voting 보다 성능이 더 좋은 편이다.
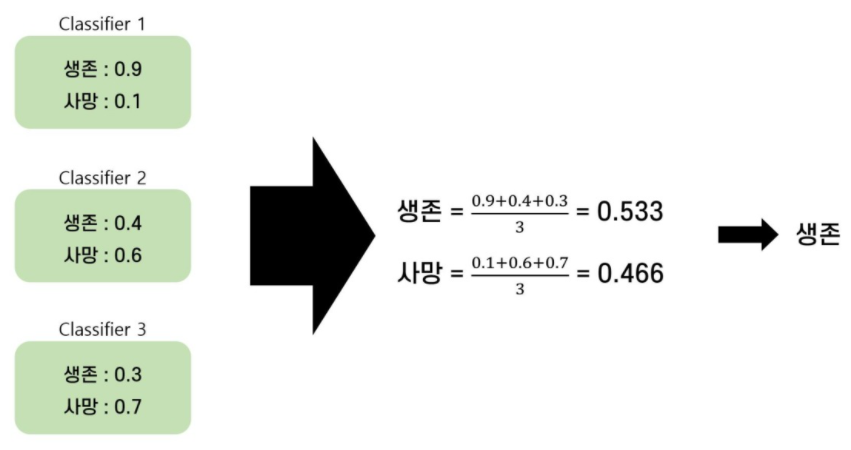 : 3개의 모델이 생존을 선택 할 확률의 평균은 0.533 이다.
: 3개의 모델이 생존을 선택 할 확률의 평균은 0.533 이다.
: 사망을 선택 할 확률의 평균은 0.466 이다.
: 최종적으로 Voting Classifier는 Hard Voting과 다르게 생존을 선택하게 된다.
8. Voting Classifier 실습
: Soft Voting을 이용하여 Voting Classifier실습을 진행한다.
: Voting Classifier에 사용할 모델은 Random Forest, Xgboost, Light GBM이다.
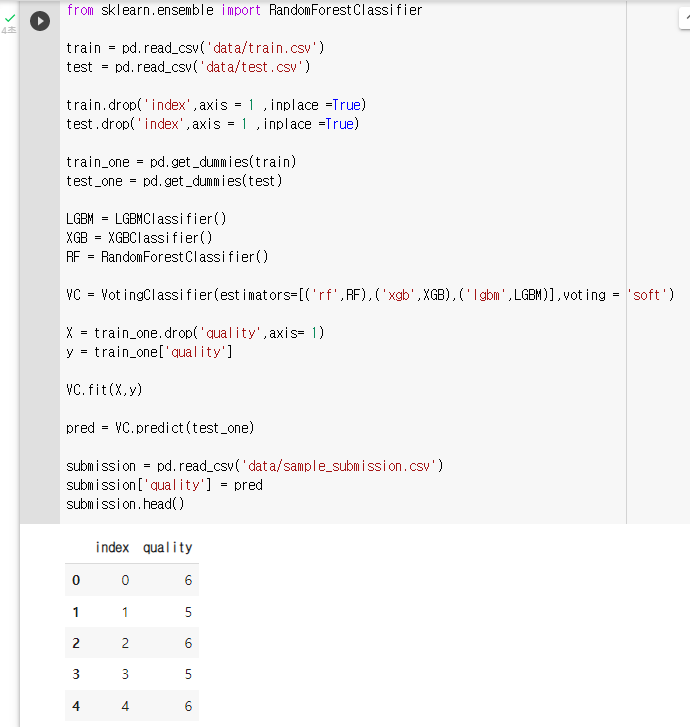
: 라이브러리 및 데이터 불러오기
: 데이터 확인train.head()
: 원핫 인코딩 (pd.get_dummies())train_one = pd.get_dummies(train) test_one = pd.get_dummies(test): 모델 정의
LGBM = LGBMClassifier() XGB = XGBClassifier() RF = RandomForestClassifier(): VotingClassifier 정의
VC = VotingClassifier(estimators=[('rf',RF),('xgb',XGB),('lgbm',LGBM)],voting = 'soft'): 모델 학습, X 는 train에서 quality 를 제외한 모든 변수, y 는 train의 qulity 변수
X = train_one.drop('quality',axis= 1) y = train_one['quality']: fit 메소드를 이용해 모델 학습
VC.fit(X,y): predict 메소드와 test_one 데이터를 이용해 품질 예측
pred = VC.predict(test_one): sample_submission.csv 파일을 불러와 예측된 값으로 채워 주기
submission = pd.read_csv('data/sample_submission.csv') submission['quality'] = pred submission.head()
: 정답파일 내보내기submission.to_csv('VC_pred.csv',index = False)
# 튜닝
1. Bayesian Optimization 복습
Bayesian Optimization
: 우리가 흔히 알고 있는 하이퍼 파라미터 튜닝방법은 Grid Search, Random Search 이다.
: Grid Search란 가능한 하이퍼파라미터 경우의 수를 일정 구간으로 나눠 구간별로 균일하게 대입 해보는 방식이다.
: 간격을 어떻게 잡을지 정하는 것이 문제고, 시간이 너무 오래걸린다는 단점이 있다.
: Random Search란 가능한 하이퍼파라미터 조합을 random하게 선택해서 대입해보는 방식이다.
: 위 두가지 튜닝방법의 문제점은 이제까지의 사전지식(실험결과) 가 반영되지 않는 다는 것이다.
: Baysian Optimization은 사전지식(실험 결과)을 반영해가며 하이퍼파라미터를 탐색한다.
: 현재까지 얻어진 모델의 파라미터와 추가적인 실험 정보를 통해 데이터가 주어 졌을 때 모델의 성능이 가장 좋을 확률이 높은 파라미터를 찾아낸다.
Bayesian Optimization 패키지 실행 단계
- 변경할 하이퍼 파라미터의 범위를 설정한다.
- Bayesian Optimization 패키지를 통해, 하이퍼 파라미터의 범위 속 값들을 랜덤하게 가져온다.
- 처음 R번은 정말 Random하게 좌표를 꺼내 성능을 확인한다.
- 이후 B번은 Bayesian Optimization을 통해 B번만큼 최적의 값을 찾는다.
2. Bayesian Optimization 실습
실습 순서
- 변경할 하이퍼 파라미터의 범위를 설정한다.
- Bayesian Optimization 에 필요한 함수 생성
- Bayesian Optimization를 이용해 하이퍼 파라미터 튜닝
: 라이브러리 불러오기
import pandas as pd from sklearn.preprocessing import MinMaxScaler, OneHotEncoder from sklearn.ensemble import RandomForestClassifier from sklearn.model_selection import KFold, train_test_split from sklearn.metrics import accuracy_score import numpy as np: 데이터를 불러와 학습시킬 준비하기
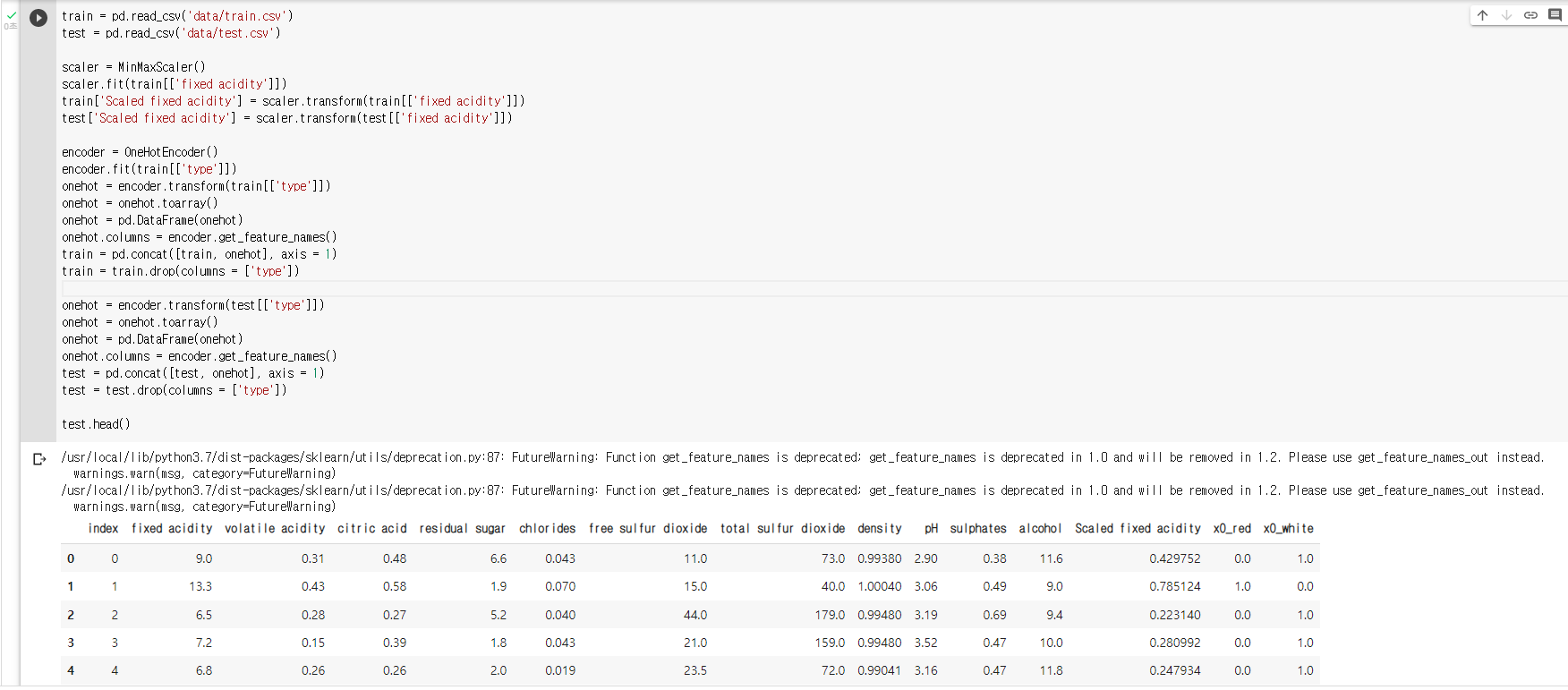
train = pd.read_csv('data/train.csv') test = pd.read_csv('data/test.csv'): Scailing
scaler = MinMaxScaler() scaler.fit(train[['fixed acidity']]) train['Scaled fixed acidity'] = scaler.transform(train[['fixed acidity']]) test['Scaled fixed acidity'] = scaler.transform(test[['fixed acidity']]): Encoding
encoder = OneHotEncoder() encoder.fit(train[['type']]) onehot = encoder.transform(train[['type']]) onehot = onehot.toarray() onehot = pd.DataFrame(onehot) onehot.columns = encoder.get_feature_names() train = pd.concat([train, onehot], axis = 1) train = train.drop(columns = ['type']) onehot = encoder.transform(test[['type']]) onehot = onehot.toarray() onehot = pd.DataFrame(onehot) onehot.columns = encoder.get_feature_names() test = pd.concat([test, onehot], axis = 1) test = test.drop(columns = ['type']) test.head()
: bayesian-optimization 설치pip install bayesian-optimization: Bayesian Optimization 불러오기
from bayes_opt import BayesianOptimization: X에 학습할 데이터를, y에 목표 변수를 저장하기
X = train.drop(columns = ['index', 'quality']) y = train['quality']: 랜덤포레스트의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
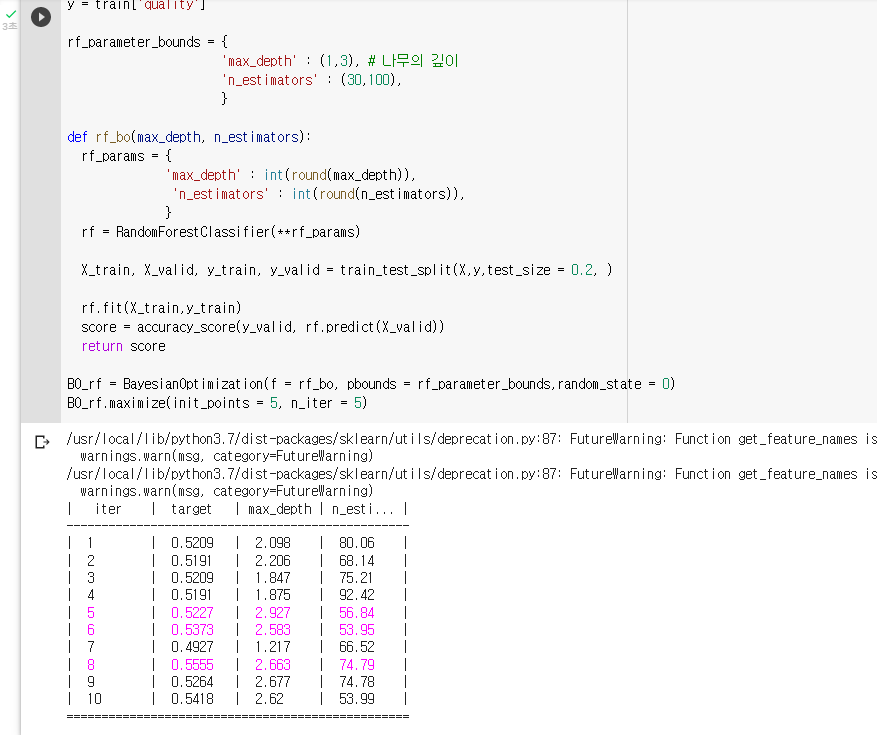
: ey는 랜덤포레스트의 hyperparameter이름이고, value는 탐색할 범위이다.rf_parameter_bounds = { 'max_depth' : (1,3), # 나무의 깊이 'n_estimators' : (30,100), }: 함수를 만든다.
1. 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2. 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3. 그 딕셔너리를 바탕으로 모델 생성
4. train_test_split을 통해 데이터 train-valid 나누기
5 .모델 학습
6. 모델 성능 측정
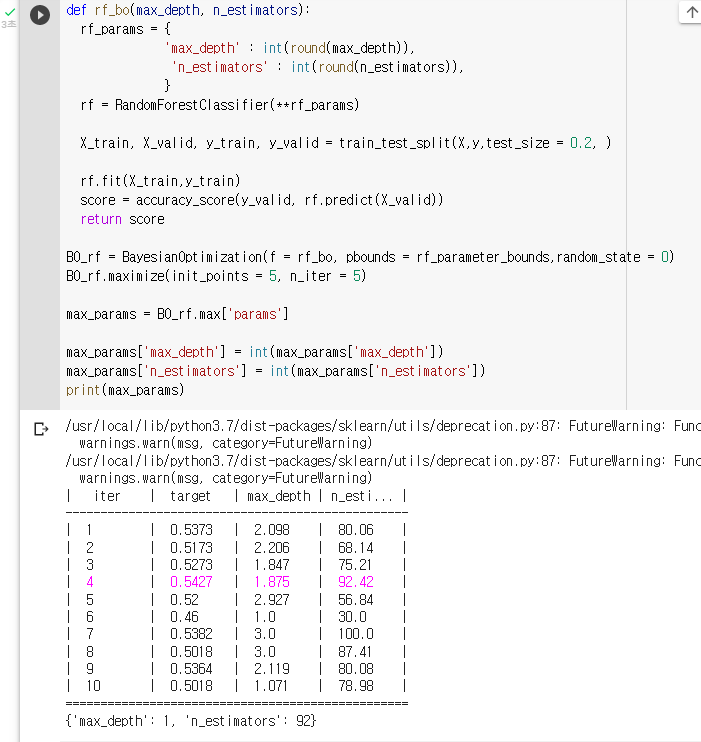
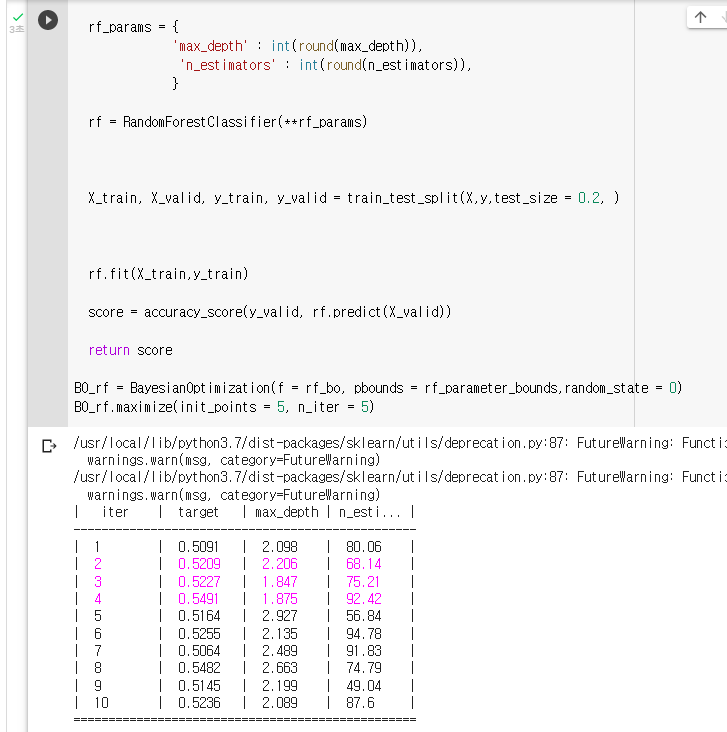
7. 모델의 점수 반환def rf_bo(max_depth, n_estimators): rf_params = { 'max_depth' : int(round(max_depth)), 'n_estimators' : int(round(n_estimators)), } rf = RandomForestClassifier(**rf_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) rf.fit(X_train,y_train) score = accuracy_score(y_valid, rf.predict(X_valid)) return score: "BO_rf"라는 변수에 Bayesian Optmization을 저장해보기
BO_rf = BayesianOptimization(f = rf_bo, pbounds = rf_parameter_bounds,random_state = 0): Bayesian Optimization을 실행해보기
BO_rf.maximize(init_points = 5, n_iter = 5)
: 하이퍼파라미터의 결과값을 불러와 "max_params"라는 변수에 저장하기max_params = BO_rf.max['params'] max_params['max_depth'] = int(max_params['max_depth']) max_params['n_estimators'] = int(max_params['n_estimators']) print(max_params)
: Bayesian Optimization의 결과를 "BO_tuend_rf"라는 변수에 저장해보세요BO_tuend_rf = RandomForestClassifier(**max_params)
3. XGBoost 튜닝
: Bayesian Optimization 을 이용해 XGBoost 모델을 튜닝한다.
XGBoost의 하이퍼 파라미터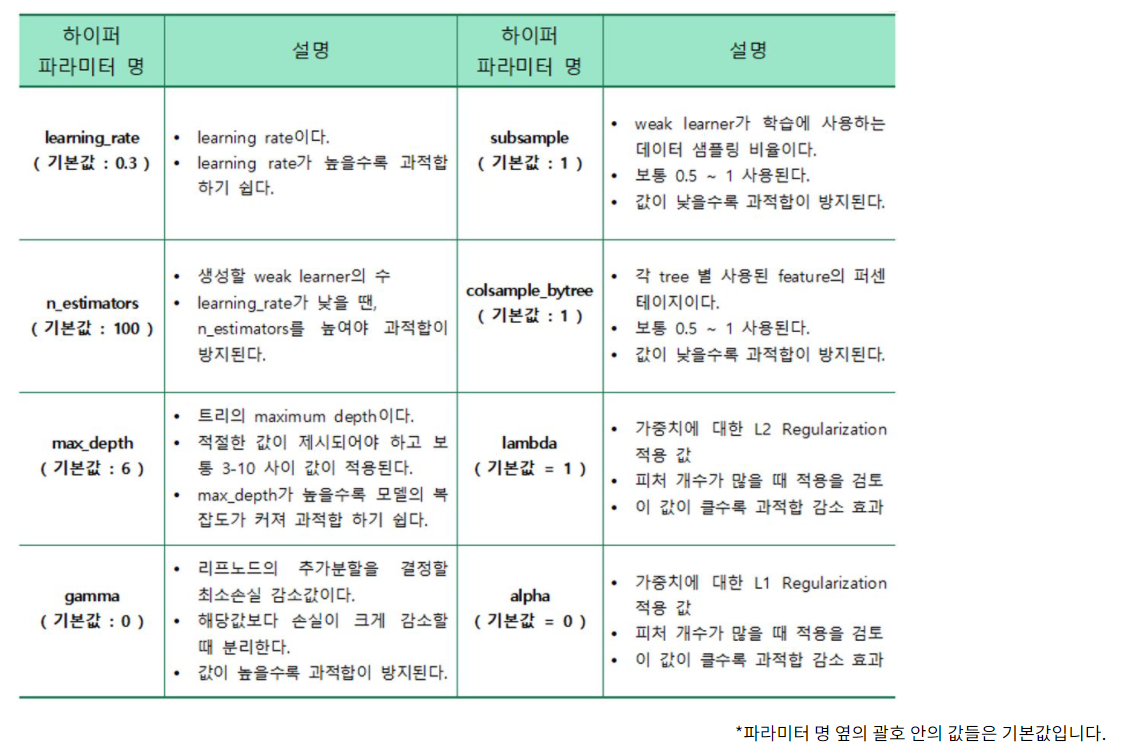
: XGBoost 파라미터중 과적합을 방지하는 gamma, max_depth, subsample 3가지 파라미터를 튜닝한다.
: X에 학습할 데이터를, y에 목표 변수를 저장하기
X = train.drop(columns = ['index', 'quality']) y = train['quality']: XGBoost의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
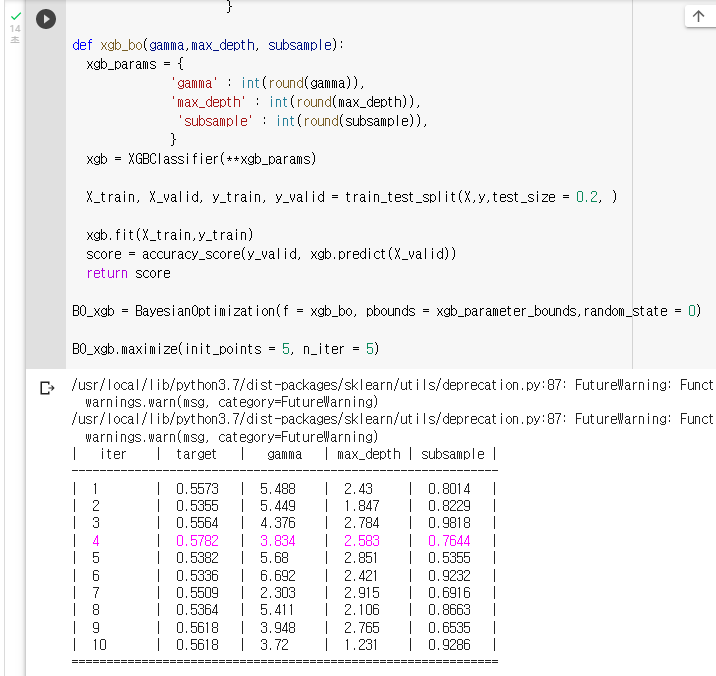
: Key는 XGBoost hyperparameter이름이고, value는 탐색할 범위이다.xgb_parameter_bounds = { 'gamma' : (0,10), 'max_depth' : (1,3), # 나무의 깊이 'subsample' : (0.5,1) }: 함수를 만든다.
1. 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2. 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3. 그 딕셔너리를 바탕으로 모델 생성
4. train_test_split을 통해 데이터 train-valid 나누기
5 .모델 학습
6. 모델 성능 측정
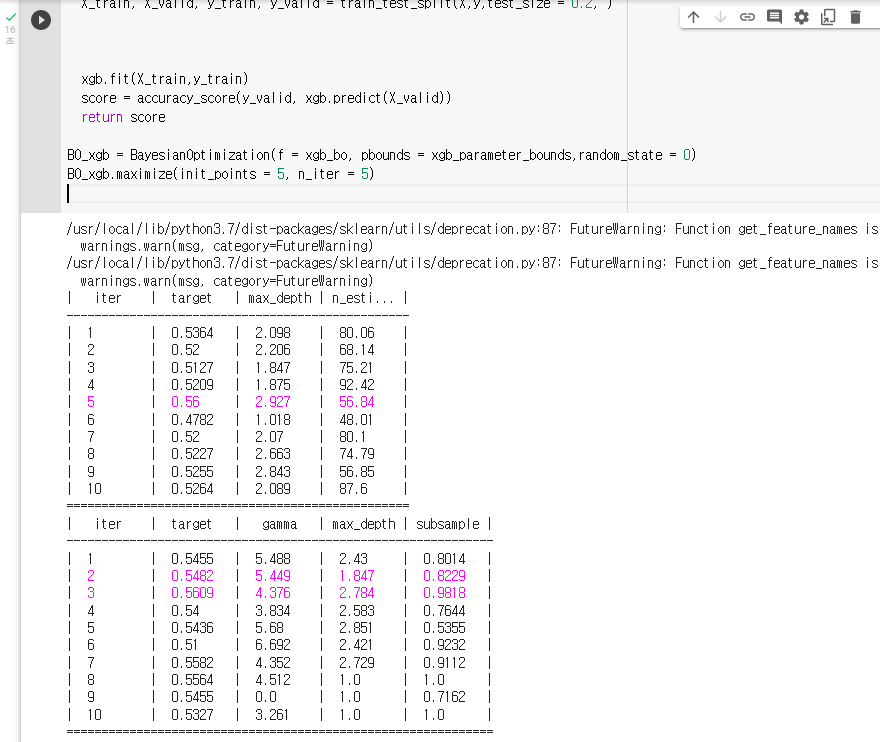
7. 모델의 점수 반환def xgb_bo(gamma,max_depth, subsample): xgb_params = { 'gamma' : int(round(gamma)), 'max_depth' : int(round(max_depth)), 'subsample' : int(round(subsample)), } xgb = XGBClassifier(**xgb_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) xgb.fit(X_train,y_train) score = accuracy_score(y_valid, xgb.predict(X_valid)) return score: "BO_xgb"라는 변수에 Bayesian Optmization을 저장하기
BO_xgb = BayesianOptimization(f = xgb_bo, pbounds = xgb_parameter_bounds,random_state = 0): Bayesian Optimization을 실행하기
BO_xgb.maximize(init_points = 5, n_iter = 5)
: 튜닝된 파라미터를 바탕으로 test 데이터 셋 예측
: 학습xgb_tune =XGBClassifier(gamma = 4.376,max_depth = 3, subsample = 0.9818) xgb_tune.fit(X,y): 예측
pred = xgb_tune.predict(test.drop(columns = ['index'] )): 정답파일 내보내기
sub = pd.read_csv('data/sample_submission.csv') sub['quality'] = pred sub.to_csv('tune_xgb.csv',index = False)
4. Light GBM 튜닝
: Bayesian Optimization 을 이용해 Light GBM모델을 튜닝한다.
: Light GBM 파라미터중 주요한 n_estimators, max_depth, subsample 3가지 파라미터를 튜닝한다.
: X에 학습할 데이터를, y에 목표 변수를 저장하기
X = train.drop(columns = ['index', 'quality']) y = train['quality']: LGBM의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
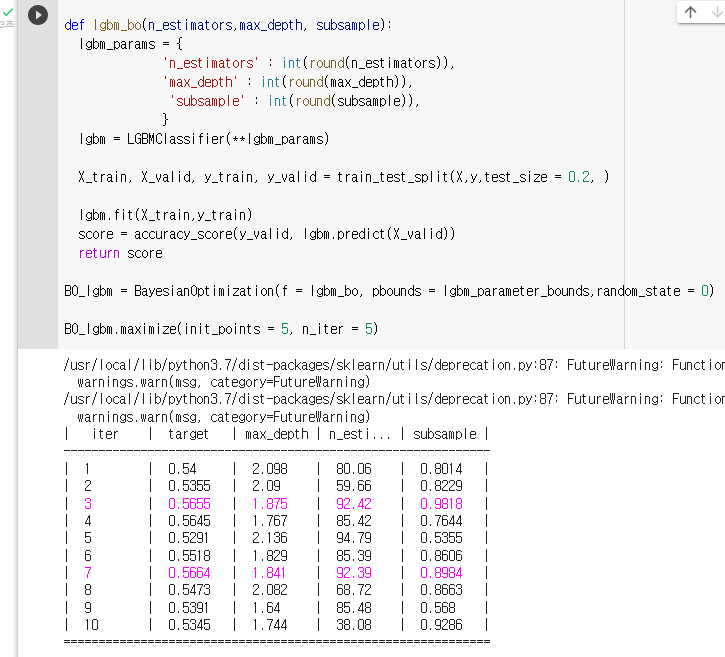
: Key는 LGBM hyperparameter이름이고, value는 탐색할 범위이다.lgbm_parameter_bounds = { 'n_estimators' : (30,100), 'max_depth' : (1,3), # 나무의 깊이 'subsample' : (0.5,1) }: 함수를 만든다.
1. 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2. 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3. 그 딕셔너리를 바탕으로 모델 생성
4. train_test_split을 통해 데이터 train-valid 나누기
5 .모델 학습
6. 모델 성능 측정
7. 모델의 점수 반환def lgbm_bo(n_estimators,max_depth, subsample): lgbm_params = { 'n_estimators' : int(round(n_estimators)), 'max_depth' : int(round(max_depth)), 'subsample' : int(round(subsample)), } lgbm = LGBMClassifier(**lgbm_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) lgbm.fit(X_train,y_train) score = accuracy_score(y_valid, lgbm.predict(X_valid)) return score: "BO_xgb"라는 변수에 Bayesian Optmization을 저장하기
BO_lgbm = BayesianOptimization(f = lgbm_bo, pbounds = lgbm_parameter_bounds,random_state = 0): Bayesian Optimization을 실행하기
BO_lgbm.maximize(init_points = 5, n_iter = 5)
: 튜닝된 파라미터를 바탕으로 test 데이터 셋 예측
: 학습lgbm_tune =LGBMClassifier(n_estimators = 43 ,max_depth = 3, subsample = 1) lgbm_tune.fit(X,y): 예측
pred = lgbm_tune.predict(test.drop(columns = ['index'] )): 정답파일 내보내기
sub = pd.read_csv('data/sample_submission.csv') sub['quality'] = pred sub.to_csv('tune_lgbm.csv',index = False)
5. 모델 튜닝 / Voting Classifier(1)
: Random Forest, XGBoost, Light GBM 총 3개의 모델을 튜닝 하고 Voting Classifier로 만든다.
순서
- 개별 모델을 basian optimization로 튜닝
- 튜닝 된 모델을 Voting Classifier로 생성
- Voting Classifier 학습 및 test 데이터 예측
- 최종 정답 파일 제출
: X에 학습할 데이터를, y에 목표 변수를 저장해주세요
X = train.drop(columns = ['index', 'quality']) y = train['quality']: 랜덤포레스트의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
: Key는 랜덤포레스트의 hyperparameter이름이고, value는 탐색할 범위이다.rf_parameter_bounds = { 'max_depth' : (1,3), # 나무의 깊이 'n_estimators' : (30,100), }: 함수를 만든다.
1. 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2. 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3. 그 딕셔너리를 바탕으로 모델 생성
4. train_test_split을 통해 데이터 train-valid 나누기
5 .모델 학습
6. 모델 성능 측정
7. 모델의 점수 반환def rf_bo(max_depth, n_estimators): rf_params = { 'max_depth' : int(round(max_depth)), 'n_estimators' : int(round(n_estimators)), } rf = RandomForestClassifier(**rf_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) rf.fit(X_train,y_train) score = accuracy_score(y_valid, rf.predict(X_valid)) return score: "BO_rf"라는 변수에 Bayesian Optmization을 저장한다.
BO_rf = BayesianOptimization(f = rf_bo, pbounds = rf_parameter_bounds,random_state = 0): Bayesian Optimization을 실행하기
BO_rf.maximize(init_points = 5, n_iter = 5)
: X에 학습할 데이터를, y에 목표 변수를 저장하기X = train.drop(columns = ['index', 'quality']) y = train['quality']: XGBoost의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
: Key는 XGBoost hyperparameter이름이고, value는 탐색할 범위이다.xgb_parameter_bounds = { 'gamma' : (0,10), 'max_depth' : (1,3), 'subsample' : (0.5,1) }: 함수를 만든다.
1. 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2. 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3. 그 딕셔너리를 바탕으로 모델 생성
4. train_test_split을 통해 데이터 train-valid 나누기
5 .모델 학습
6. 모델 성능 측정
7. 모델의 점수 반환def xgb_bo(gamma,max_depth, subsample): xgb_params = { 'gamma' : int(round(gamma)), 'max_depth' : int(round(max_depth)), 'subsample' : int(round(subsample)), } xgb = XGBClassifier(**xgb_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) xgb.fit(X_train,y_train) score = accuracy_score(y_valid, xgb.predict(X_valid)) return score: "BO_xgb"라는 변수에 Bayesian Optmization을 저장하기
BO_xgb = BayesianOptimization(f = xgb_bo, pbounds = xgb_parameter_bounds,random_state = 0): Bayesian Optimization을 실행하기
BO_xgb.maximize(init_points = 5, n_iter = 5)
: X에 학습할 데이터를, y에 목표 변수를 저장하기X = train.drop(columns = ['index', 'quality']) y = train['quality']: LGBM의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
: Key는 LGBM hyperparameter이름이고, value는 탐색할 범위이다.lgbm_parameter_bounds = { 'n_estimators' : (30,100), 'max_depth' : (1,3), # 나무의 깊이 'subsample' : (0.5,1) }: 함수를 만든다.
1. 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2. 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3. 그 딕셔너리를 바탕으로 모델 생성
4. train_test_split을 통해 데이터 train-valid 나누기
5 .모델 학습
6. 모델 성능 측정
7. 모델의 점수 반환def lgbm_bo(n_estimators,max_depth, subsample): lgbm_params = { 'n_estimators' : int(round(n_estimators)), 'max_depth' : int(round(max_depth)), 'subsample' : int(round(subsample)), } lgbm = LGBMClassifier(**lgbm_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) lgbm.fit(X_train,y_train) score = accuracy_score(y_valid, lgbm.predict(X_valid)) return score: "BO_lgbm"라는 변수에 Bayesian Optmization을 저장하기
BO_lgbm = BayesianOptimization(f = lgbm_bo, pbounds = lgbm_parameter_bounds,random_state = 0): Bayesian Optimization을 실행하기
BO_lgbm.maximize(init_points = 5, n_iter = 5): 모델 정의 (튜닝된 파라미터로)
LGBM = LGBMClassifier(max_depth = 2.09,n_estimators=60, subsample = 0.8229) XGB = XGBClassifier(gamma = 4.376, max_depth = 2.784, subsample = 0.9818) RF = RandomForestClassifier(max_depth = 3.0, n_estimators = 35.31): VotingClassifier 정의
VC = VotingClassifier(estimators=[('rf',RF),('xgb',XGB),('lgbm',LGBM)],voting = 'soft')
6. 모델 튜닝 / Voting Classifier(2)
: Voting Classifier을 이용해 train 데이터를 학습하고 test 데이터 셋을 예측
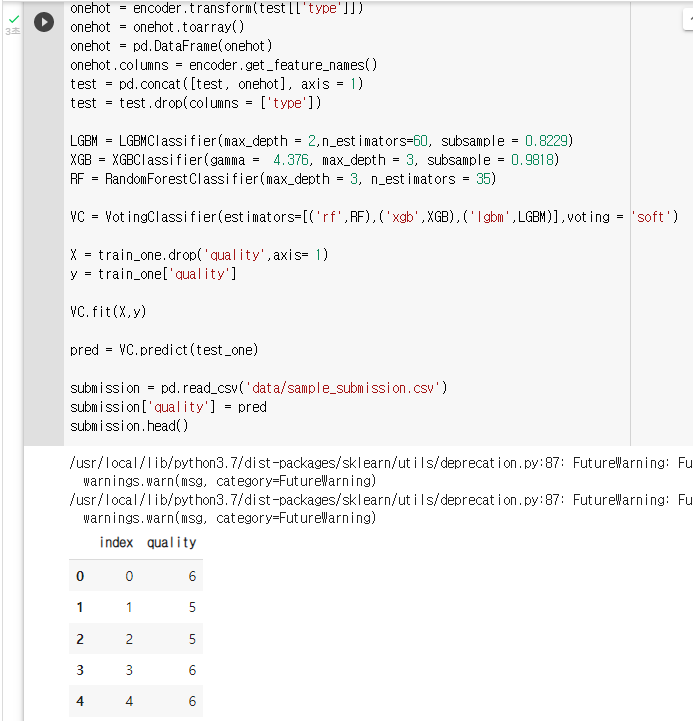
: 모델 정의 (튜닝된 파라미터로)
LGBM = LGBMClassifier(max_depth = 2,n_estimators=60, subsample = 0.8229) XGB = XGBClassifier(gamma = 4.376, max_depth = 3, subsample = 0.9818) RF = RandomForestClassifier(max_depth = 3, n_estimators = 35): VotingClassifier 정의
VC = VotingClassifier(estimators=[('rf',RF),('xgb',XGB),('lgbm',LGBM)],voting = 'soft') X = train_one.drop('quality',axis= 1) y = train_one['quality']: fit 메소드를 이용해 모델 학습
VC.fit(X,y): predict 메소드와 test_one 데이터를 이용해 품질 예측
pred = VC.predict(test_one): sample_submission.csv 파일을 불러와 예측된 값으로 채워 주기
submission = pd.read_csv('data/sample_submission.csv') submission['quality'] = pred submission.head()