[논문 리뷰] Unet 3+: A full-scale connected unet for medical image segmentation
2022 하계 Paper Review

본 Paper Review는 고려대학교 스마트생산시스템 연구실 2022년 하계 논문 세미나 활동입니다.
논문의 전문은 여기에서 확인 가능합니다.
Abstract
-
딥러닝 기반의 semantic segmentation이 트렌드임.
-
UNet은 encoder-decoder 구조의 딥러닝 semantic segmentation 모델임.
-
Multi-scale features를 결합하는 것이 정확한 segmentation의 핵심.
-
UNet++은 nested & dense skip connections 사용해서 UNet을 개조한 것.
-
하지만 기존 두 모델은 충분히 분할해주지 못해서 개선의 여지가 큼.
-
본 논문에서 제시하는 UNet 3+는 full-scale skip connections과 deep supervision 통해서 성능을 높임. (둘에 대해서는 뒤에서 자세히 살펴볼 것)
-
해당 모델은 특히 다양한 scale을 보이는 organ (장기) 이미지에 대해서 좋은 분할 성능을 보임.
-
또한 UNet 3+는 network parameters를 줄여 computation effiency를 높임.
-
추가로 hybrid loss funciton과 classification-guided module를 고안함.
1. Introduction
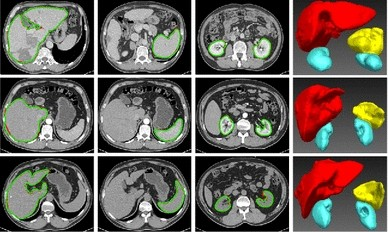
의료 분야에서 organ (장기) 의료 이미지를 segmentation하는 작업은 중요합니다.
따라서 CNN 기반의 여러 가지 segmentation 모델들이 개발되었는데 다음과 같습니다.
- Fully convolutional neural networks (FCNs), UNet, PSPNet, DeepLab
특히 encoder-decoder 구조의 UNet이 의료 이미지 segmentation에 주로 쓰입니다.
UNet은 encoder에서 나온 low-level detailed feature maps를 decoder의 high-level semantic feature maps와 결합하는 Skip connections 방식을 사용합니다.
하지만 이들의 결합은 약했고, 이를 보완하기 위해 nested & dense skip connections를 도입한 UNet++이 등장했습니다. 위 두가지를 적용함으로써 모델은 encoder와 decoder 사이에 존재하는 semantic gap을 줄이게 됩니다.
해당 모델들이 좋은 성능을 보임에도 불구하고, full scales로부터의 정보는 충분히 담아내지 못했습니다.
이게 왜 문제가 되냐면, 다양한 선행 연구에서도 나타나듯이 different scale에서의 feature maps는 서로 다른 정보를 담고 있기 때문입니다.

- Low-level detailed feature maps는 organs에 boundary를 그려줄 만큼 풍분한 공간적 정보를 담고 있고, High-level semantic feature maps는 organs의 위치가 어디인지 정도의 대략적인 위치 정보만을 담고 있습니다.
이러한 정교한 signals들은 down- & up- sampling하면서 점점 희석됩니다.
따라서 이를 극복하고자 본 논문은 UNet 3+ 구조를 제안하는데 이는 multi-scale features를 완전하게 사용하고자 제안된 U-shape의 구조입니다.
UNet 3+의 방법론은 encoder와 decoder 사이의 inter-connection 방식을 수정하고, 동시에
full scales로부터의 fine-grained details와 coarse-grained semantics 정보를 포착하기 위해서
decoder들 사이의 inter-conncection 방식을 도입합니다.
계층적 representation을 full-scale aggregated feature maps로부터 학습하기 위해서, 각 output를 hybrid loss function으로 연결하여 정확한 segmentation을 할수 있도록 합니다.
성능도 좋아졌지만 network parameters를 줄여서 computation efficiency도 향상시킵니다.
특히 의료분야에서 segmentation의 정확도를 높이는 것이 중요하기 때문에 non-organ 이미지에 대한 false positives를 줄여야 합니다.
이를 위해서 일반적으로 Attention Mechanism을 도입하거나 CRF와 같은 pre-defined refinement 접근법을 사용합니다.
이러한 방법들과 달리, 본 논문은 segmentation 작업을 수행함으로써 input image가 organ을 포함하고 있는지를 예측해내는 classification task도 가능합니다.
정리하자면 본 논문의 Contribution 4가지는 다음과 같습니다.
1) full-scale skip connections를 도입해서 multi-scale features를 전부 사용할 수 있는 UNet 3+ 모델을 고안
2) hybrid loss function 최적화하여 full-scale aggregated feature maps로부터 계층적 representation을 학습할 수 있도록 하는 deep supervision 사용
3) organ image가 아닌 image에 대해서 over-segmentation을 줄이기 위해 image-level 분류를 학습하는 classification-guided module 제안
4) liver & spleen dataset에 대해서 실험하여 UNet 3+가 기존 모델들에 비해 성능 향상을 이루었는지 확인
2. Methods
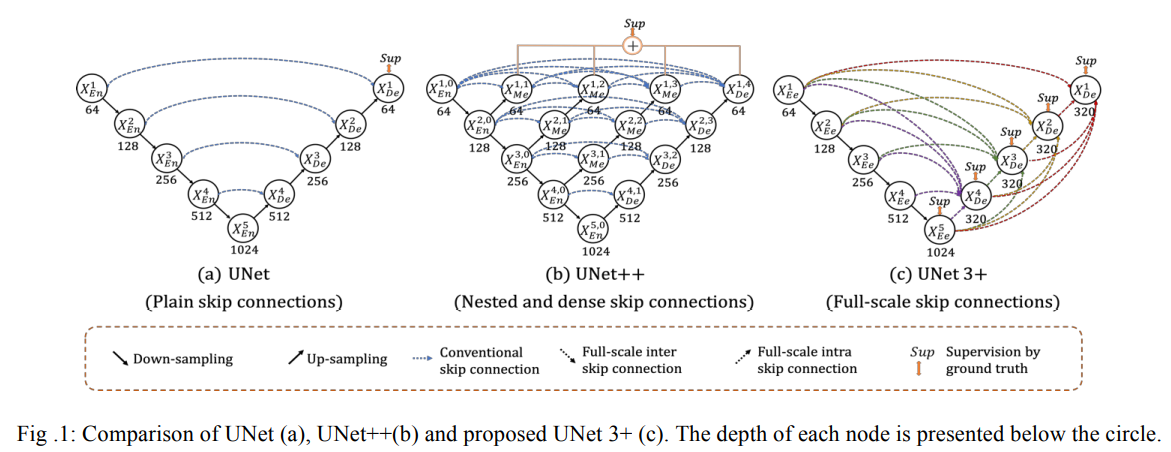
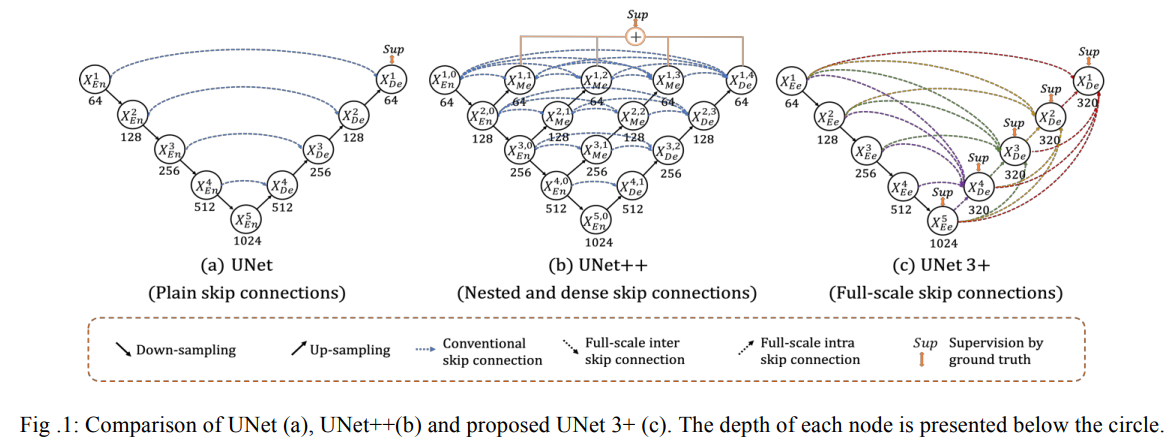
Fig1은 UNet, UNet++, UNet 3+ 각각의 구조를 나타냅니다.
UNet 3+ 구조가 이전 두 구조와 다른 점은 skip-connections 방법을 수정하고, full-scale deep supervision을 사용해서 더 적은 parameters를 학습에 이용하고 이를 통해 segementation map에 더 정확한 위치 정보와 boundary를 보장할 수 있도록 하는 것입니다.
따라서 Full-scale Skip Connections와 Full-scale Deep Supervision이 UNet 3+ 모델의 핵심입니다.
2.1. Full-scale Skip Connections
본 논문에서 제안된 full-scale skip connections 방식은 encoder와 decoder 사이의 skip connections를 수정하면서 동시에 decoder 간의 skip connections을 추가합니다.
그렇게 하는 이유는 기존 모델 UNet과 UNet++이 full scales로부터 충분한 정보를 획득하지 못해서 organ의 위치와 boundary를 정확하게 분리해내지 못했기 때문입니다.
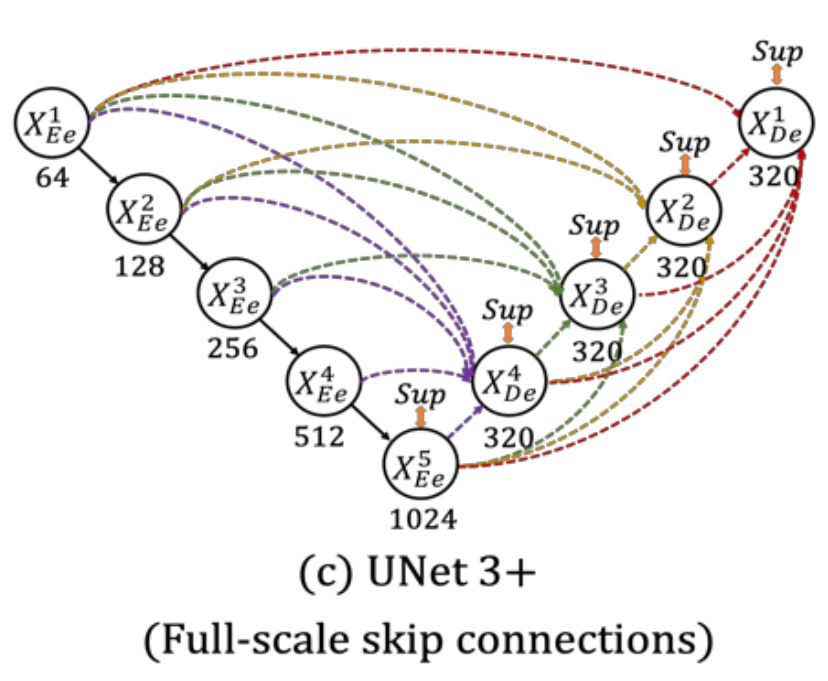
이를 극복하기 위해서 UNet 3+는 각 decoder layer에 더 작거나 같은 스케일의 encoder feature maps와 더 큰 스케일의 decoder feature maps를 함께 사용해서 fine-grained details와 coarse-grained semantics를 동시에 보장하게 됩니다.
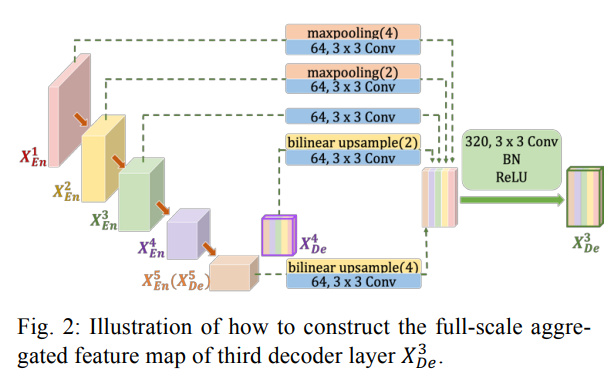
Fig 2에서는 3번째 decoder layer에서 어떻게 feature maps들이 결합되어 사용되는지를 보여줍니다.
기존 UNet과 유사하게 3번째 decoder는 3번째 encoder로부터 feature map을 받습니다. 하지만 추가로 더 낮은 scale 그러니까 1, 2번째 encoder로부터도 feature maps를 받습니다. 받을 때에는 non-overlapping max pooling 과정을 거칩니다.
뿐만 아니라, 더 큰 scale의 decoder로부터도 feature maps를 받는데 이 경우에는 4, 5번째 decoder로부터 feature maps를 받게됩니다. 이 때는 bilinear interpolation을 사용하게 됩니다.
그렇게 모인 5개의 feature maps를 통합하는데 불필요한 정보를 줄이면서 채널의 수를 통합합니다. 이 경우에는 3x3 크기의 64개의 Convolutional 필터를 사용하는걸 알 수 있습니다.
얕고 깊은 정보들을 매끄럽게 통합하기 위해서 feature aggregation mechanism을 거치게 되는데 위 그림에서 확인할 수 있듯이 3x3 크기의 320개의 Convolutional 필터를 거치고 Batch Normalization, ReLU 활성화함수를 거치는 것을 볼 수 있습니다.
예시로 설명한 위 과정을 수식으로 나타낸다면 다음과 같습니다.
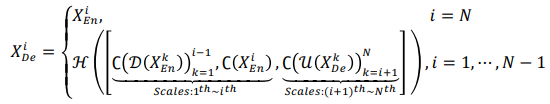
각각의 notation은 다음과 같습니다.
| Notation | Meaning |
|---|---|
| C | convolution |
| H | feature aggregation mechanism (convolution, BN, ReLU) |
| D | down-sampling |
| U | up-sampling |
| [ ] | concatenation |
또한, UNet 3+가 기존 UNet과 UNet++ 보다 parameters 측면에서 더 적게 사용해서 효율적이라고 판단한 이유는 다음과 같습니다.
일단 UNet, UNet++, UNet 3+의 Encoder 구조는 동일하기에 encoder에서 학습에 사용되는 parameter의 수는 동일합니다. 하지만 Decoder 구조의 차이가 있습니다.
(1) UNet
UNet의 경우 Decoder와 Encoder가 깊이가 대칭적이기 때문에 i번째 decoder의 채널의 개수는 32X2^i 입니다.
이 경우 i번째 decoder의 파라미터의 수는 다음과 같습니다.
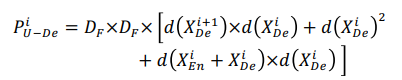
여기서 D_F는 convolution kernel size, d()는 노드의 깊이를 의미합니다.
(2) UNet++
UNet++의 i번째 decoder의 파라미터 수는 다음과 같습니다.
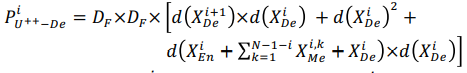
UNet에 비해 추가적으로 Sigma 항이 더해진걸 알 수 있고 이를 통해 UNet보다 UNet++의 파라미터 수가 더 크다는 것을 알 수 있습니다.
(3) UNet 3+
반면 UNet 3+의 decoder feature map은 N scales로부터 단순하게 오기 때문에 64XN개의 채널만을 같습니다. 따라서 i번째 decoder의 파라미터 수는 다음과 같습니다.

위 수식을 통해 UNet 3+의 파라미터 수가 UNet, UNet++의 비해 적다는 것을 파악할 수 있었습니다.
2.2. Full-scale Deep Supervision
Full-scale aggregated feature maps로부터 계층적인 representations를 학습하기 위해서 full-scale deep supervision이 채택됐습니다.
이는 UNet++에서 full-resolution feature map을 만들기위해 사용되었던 deep supervision과 다르게,
각 decoder stage로부터 나오는 side output에 ground truth와의 supervision을 한 것입니다.
방법은 각 decoder stage의 마지막 layer에 3x3 convolution layer, bilinear up-sampling, sigmoid function을 거치는 것입니다.
추가로 organ의 boundary를 더 정확하게 분리하기 위해서,
본 논문은 multi-scale structural similarity index (MS-SSIM) loss function을 제안합니다.
해당 손실함수는 fuzzy boundary에 더 높은 가중치를 주는 역할을 수행합니다.
이를 통해 지역적 분포 차이가 커질수록, MS-SSIM 값이 커지도록하여 흐릿한 경계에 더 집중하게 됩니다.
수식은 아래와 같습니다.
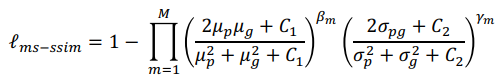
위 notation 중 p와 g는 NxN 사이즈의 패치를 crop하는데,
전자는 segmentation 결과이고 후자는 ground truth입니다.
M은 scale의 total number, 나머지 notation은 p와 g의 평균, 표준편차, 공분산을 의미합니다.
베타 값과 감마 값은 각 scale에서의 두 component들의 중요도,
C1, C2는 0으로 나눠지지 않도록하기 위한 아주 작은 수를 부여합니다.
마지막으로 다른 논문에서 제시된 focal loss & IoU loss를 MS-SSIM loss와 결합하여 hybrid loss를 만듭니다.
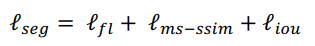
3개의 loss function을 결합함으로써, pixel, patch, map 계층적 segmentation이 전부 가능케합니다.
따라서, large-scale, fine-structures 전부 깔끔한 경계로 구분이 됩니다.
2.3. Classification-guided Module (CGM)
대부분의 medical image segmentations에서 non-organ image에 대한 false-positives는 발생하게 됩니다.
이유는 얕은 층에 남아있는 배경에서 noisy 정보를 담고있고 over-segmentation을 야기하기 때문입니다.
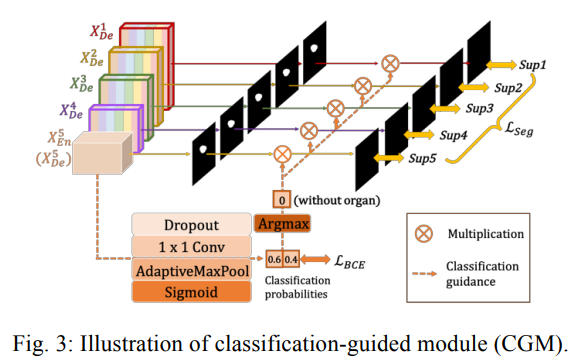
따라서 본 논문의 저자는 이를 해결하고자 추가적인 classification을 수행하게 됩니다.
방법은 Fig. 3에 나타나듯이, 마지막 decoder를 Dropout, 1x1 Conv, maxpooling, sigmoid를 거치게 하여 2차원 tensor를 만들고 이를 통해 각 decoder에서 나온 feature map에서 organ이 있는지 없는지를 분류하게 됩니다.
이는 binary classification하는 간단한 문제이기에 module이 단순하게 Binary Cross Entropy loss function을 최적화하여 정확한 분류 결과를 도출합니다.
따라서 non-organ image의 over-segmentataion 문제에 대한 해결책이 됩니다.
3. Experiments and Results
3.1. Datasets and Implementation
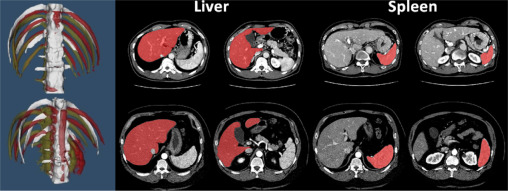
실험에는 liver(간)와 spleen(비장) 두 organ의 image가 사용되었습니다.
먼저 liver의 경우 103개의 training set, 28개의 testing set 총 131개의 3D CT Scan 이미지가 있습니다.
다음으로 spleen의 경우 training set, testing set 각각 40, 9개의 CT 이미지가 존재합니다.
실험 결과 비교를 위해 Dice coefficient 평가 지표가 사용되었고 수식은 다음과 같습니다.
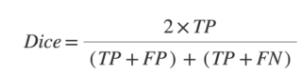
3.2. Comparison with UNet and UNet++
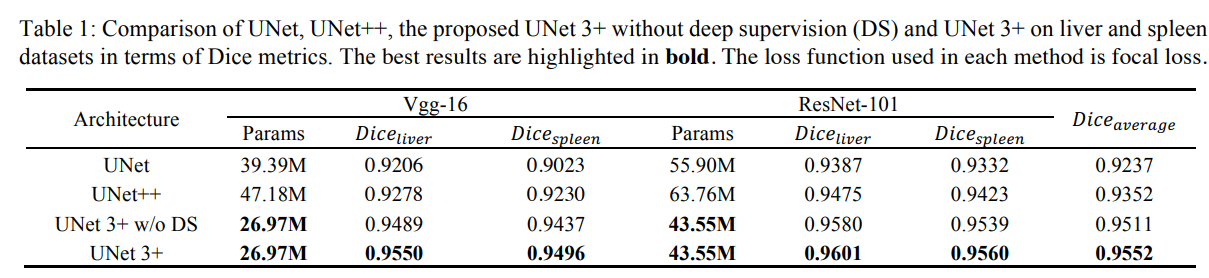
Table 1은 실험 결과입니다. 단순히 봐도 알 수 있듯이, UNet 3+가 기존 두 모델보다 성능도 좋고 더 적은 Parameter를 학습에 사용함을 알 수 있습니다. 나아가 Full-scale Deep Supervision을 사용한 모델이 같은 수의 Parameter를 학습에 사용함과 동시에 더 발전된 성능을 보이는 것을 알 수 있습니다.
모든 모델들은 Vgg-16과 ResNet101 기반으로 짜여졌습니다.
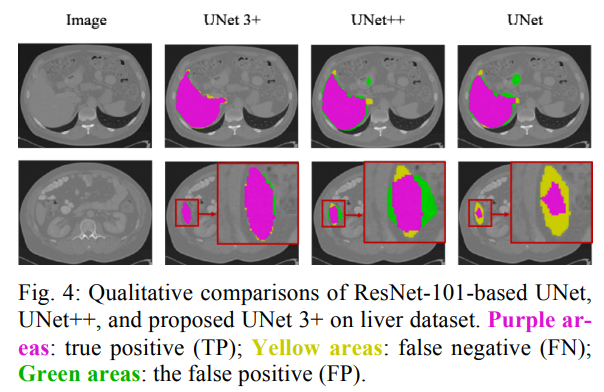
시각적으로 살펴보아도 UNet 3+ 모델이 거의 오류없이 segementation하는 것을 알 수 있습니다.
3.3. Comparison with the State of the Art

마지막으로 5개의 SOTA model들과 비교했을 때에도 두 데이터셋 모두에서 UNet 3+의 뛰어난 성능을 발견할 수 있습니다.
Conclusion
- 정확한 segmentation과 효율적인 네트워크 구조를 위해, full scale에서 feature maps을 최대로 활용할 수 있는 deep supervision을 활용한 full-scale connected 모델 UNet 3+를 제안함.
- CGM과 hybrid loss function을 추가로 제안하여 더 정확한 position-aware와 boundary-aware segmentation map을 제시함.
- 실험 결과 또한 기존 SOTA 모델들에 비해 더욱 좋은 성능을 보여줌.
함께 읽으면 좋을 논문
End-to-End Object Detection with Transformers
(리뷰) End-to-End Object Detection with Transformers
