[Paper Review] Self-supervised learning improves dMMR/MSI detection from histology slides across multiple cancers
Paper Review

Self-supervised learning improves dMMR/MSI detection from histology slides across multiple cancers
앞서 언급했던 논문과 비슷하게 Attention-based aggregation, weakly supervised learning with self-supervised learning와 관련된 내용을 담고 있는 논문이다. 추가로 explainability 내용도 조금 나온다.
Introduction
논문에서 소개한 모델의 타겟인 Microsatellite Instability (MSI) 는 DNA repair system의 붕괴로 인한 short DNA motifs의 비정상적인 반복에 의해 나타나는, tumor cell에서 자주 등장하는 phenotype 중 하나이다. MSI tumor (phenotype) 은 대장암 발병 초기에 좋은 예후를 보여주는 등 dMMR (MMR deficient tumors) 와 MSI 진단은 환자의 therapeutic decision와 care 측면에서 중요하다. 하지만 이를 진단하는 기존의 방법들 (IHC, PCR assay, NGS) 은 비용이 많이 들기에 WSI만을 이용하여 이를 pre-screening 하는 모델이 연구되어 왔다.
이번 연구에서는 self-supervised learning을 포함한 2가지의 feature extraction 방법과 Deep MIL을 포함한 3가지의 tile aggregation을 조합한 실험을 통해 dMMR/MSI detection 모델 성능을 측정하였으며, 다른 organ의 이미지에서도 robust하게 학습할 수 있음을 발견했다. 또한 MIL 방법 중 하나인 Chowder 모델로부터 각 tile score로 slide를 visualization하는 등 explainability 관련한 내용도 조금 담고 있다.
Method
Proposed pipeline
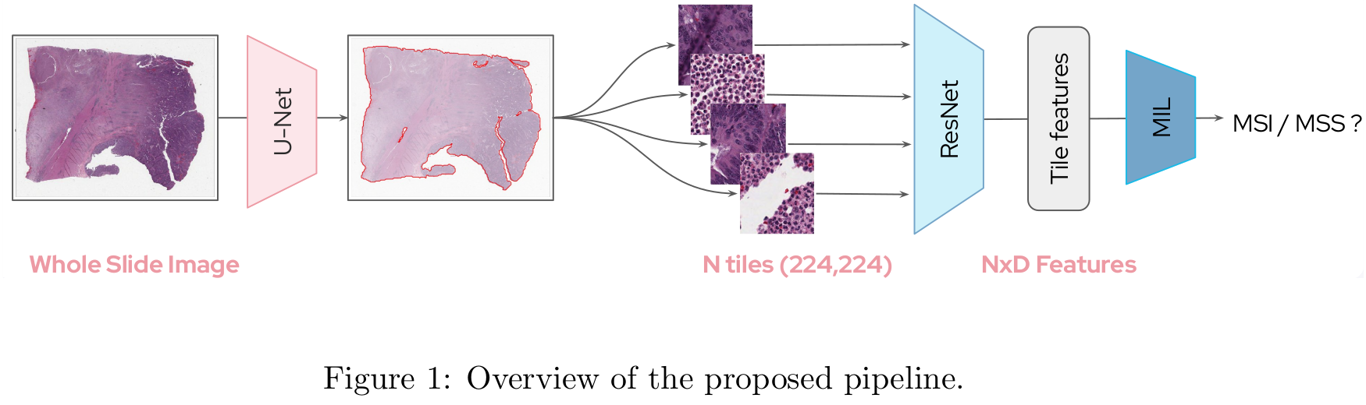
- U-net 기반의 neural network를 통한 background 제거
- Segmentation to N tiles, 224 x 224 pixels
- Feature embedding with pre-trained CNN
- N x D features are aggregated with MIL
Benchmarked 2 different feature extractors (ResNet-50 pretrained with supervised learning on ImageNet: D=256, or with SSL on TCGA: D=2048) and 3 multiple instance learning models (MeanPool, Chowder and DeepMIL)
Feature extractors
ImageNet feature extraction
ResNet-50 pretrained using supervised learning on the ImageNet-1k dataset 에서 last layer을 사용함. Auto-encoder를 사용하여 =256 으로 dimension을 낮춤. (Chowder에서의 유의미한 성능 향상 때문)
MoCo feature extraction
Feature extraction 방법으로 self-supervised learning, 그 중에서도 dynamic dictionary 를 활용한 contrastive learning, MoCo v2(Momentum Contrast)를 사용함. 구체적으로 ResNet-50 model을 사용하였고 기존 MoCo 논문과 동일한 parameter와 data augmentation 방식을, 기존보다 큰 ResNet backbone을 사용하였다. (the bottleneck number of channels is twice larger in every block)
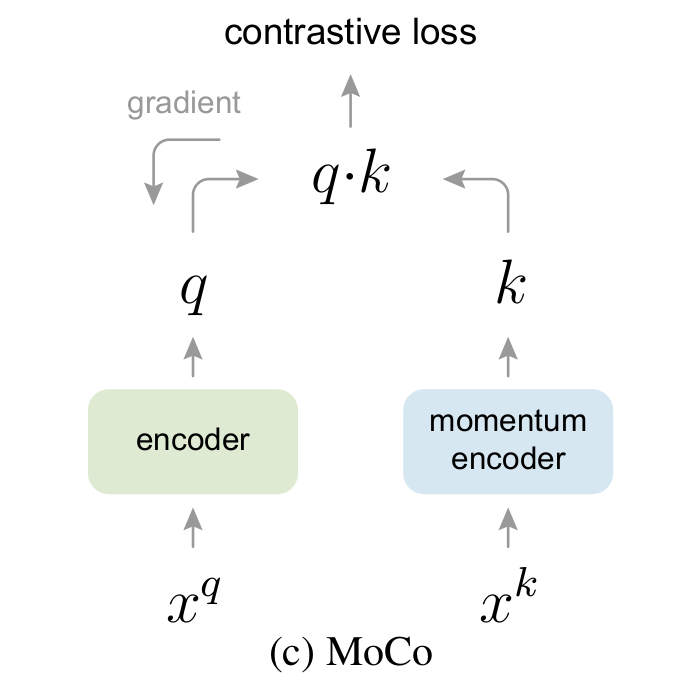
MoCo는 query encoder와 key encoder에 의해 encoding된 visual representation 간의 contrastive loss를 통해 학습한다. 이 때 query encoder는 back propagation을 통해 학습되지만, key encoder은 dynamic한 특성 때문에 발생하는 consistency 이슈로, back propagation 대신 query encoder로부터의 momentum update를 통해 학습한다. Query encoder를 , parameter를 , key encoder의 그것들을 각각 , 라 했을 때 momentum update는 다음과 같이 이루어진다.
MoCo v2에서는 MoCo의 시조버전 이후 성능이 더 좋게 나온 SimCLR에서 크게 3가지를 벤치마킹하여 발전시킨 모델이다. 간단히 요약하면 1) 학습에만 MLP 구조의 projection head를 사용하고, 이후엔 제외하고 representation 을 추출한 것, 2) 여러 data augmentation를 시도하여 Gaussian blurring을 추가한 것, 3) cosine learnig rate schedule 가 되겠다. 자세한 내용은 MoCo 논문을 참고하도록 하자.
Multiple Instance Learning methods
MeanPool
Mean function을 활용하여 각 instance를 aggregation 한다. 이후 L2 penalization을 활용했다고 한다.
Chowder
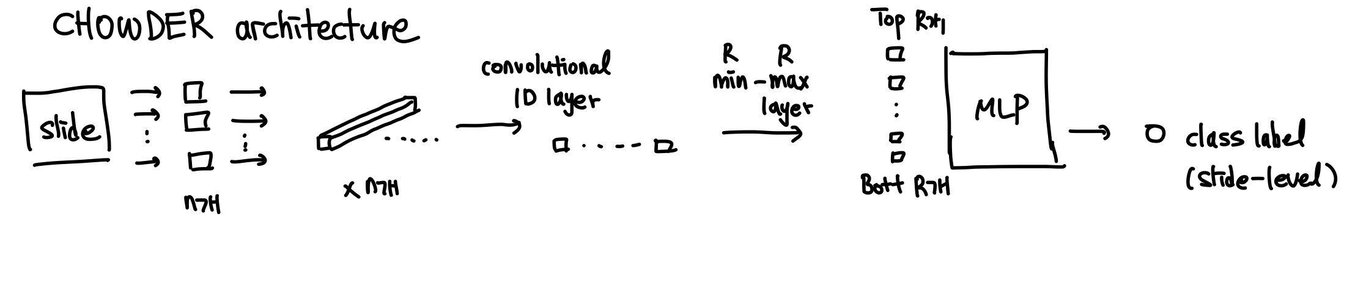
각각의 tile이 tile-level descriptors로 1차 변환되고, convolutional layer를 통해 value로 변환된 후 sorting을 통해 top R개, bot R개를 추출한 것으로 MLP를 거쳐 slide-level label을 내는 방식이다.
Attention-based Deep MIL
Attention을 이용한 MIL pooling의 수식은 다음과 같다.
이에 더하여 Gated Attention layer이 적용된 attention based model이 사용되었다. 수식은 다음과 같다.
Datasets
Three different cohorts were used in this study and are summarized in Table below.
(TCGA-CRC, TCGA-Gastric, PAIP)

Kather이 붙어 있는 cohort는 Kather의 연구에서 활용된 TCGA dataset의 variants이다. 모든 데이터셋에서 FFPE (formalin-fixed paraffin-embedded) sample만이 선택되어 사용되었다.
Results

먼저 사용된 세가지 방식의 MIL pooling을 비교하였다. 모두 MoCo-Kather cohort를 pre-train된 feature를 이용해 Kather 논문에서 사용된 train / test split으로 결과를 얻었다. TCGA-CRC-KATHER, TCGA-GASTRIC-KATHER 두 cohort에서 Chowder 방식을 사용한 모델에서 AUC 성능이 가장 높게 측정되었다.
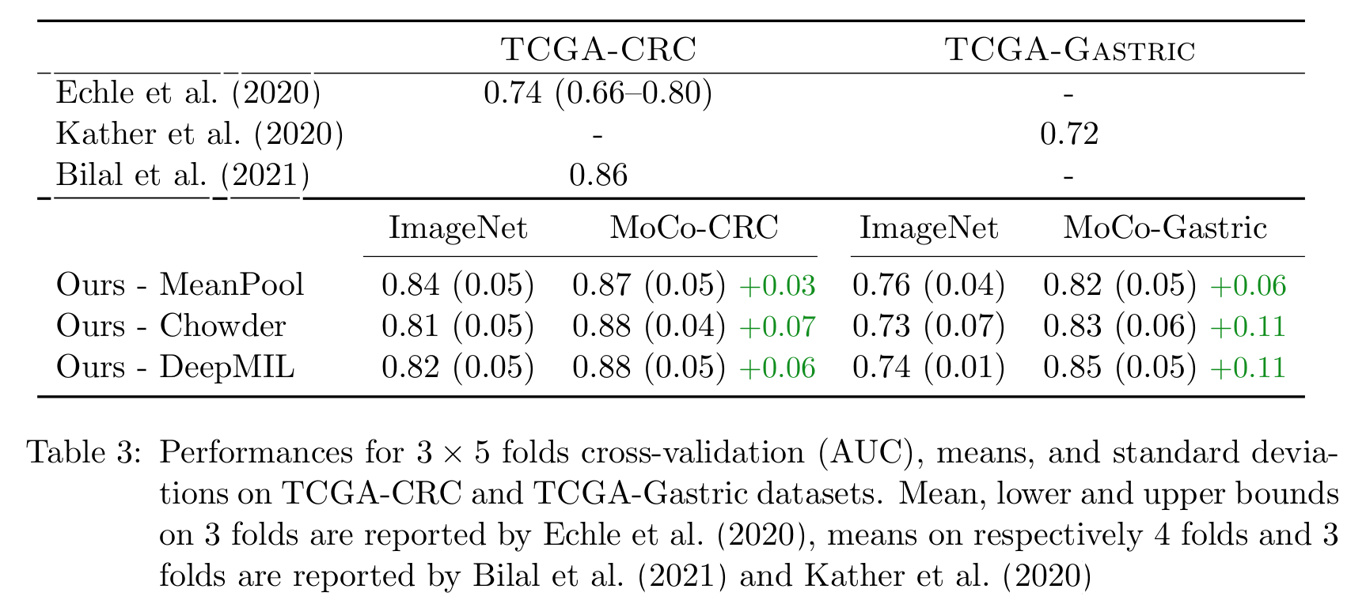
다음으로 MoCo v2를 활용한 feature extractor의 성능을 비교하기 위해 3 x 5 fold cross-validation (5 fold cv, repeated 3 times, 15 distinct splits on the full TCGA cohorts) 를 시행하였고, MoCo를 사용한 모델에서 AUC가 더 높게 측정되는 것을 확인하였다.
External validation on CRC dataset PAIP
저자는 앞서 진행된 실험이 SSL을 사용하여 label이 없었음에도 불구하고, TCGA cohort를 활용한 pretraining 때문에 train/test split independence assumption을 부수었다고 지적한다. 이에 따라 다른 independent cohort인 PAIP에 대해 추가 실험을 진행하였다.

TCGA-CRC로 pre-train된 feature extractor를 활용하여 다른 cohort인 PAIP dataset에 대해 같은 실험을 수행하였고, MoCo-CRC pretrained backbone의 성능을 다시 한번 확인하였다.
Transfer CRC to Gastric
각 Feature extractor의 robustness를 확인하기 위해, TCGA-CRC에서 train을 시킨 후 TCGA-Gastric에서 evalute 되었다. 정리하자면, pre-train은 각 모델이 ImageNet, CRC, CRC+Gastric로 진행하였고 이후 train을 CRC에서, test를 Gastric에서 한 것이다.
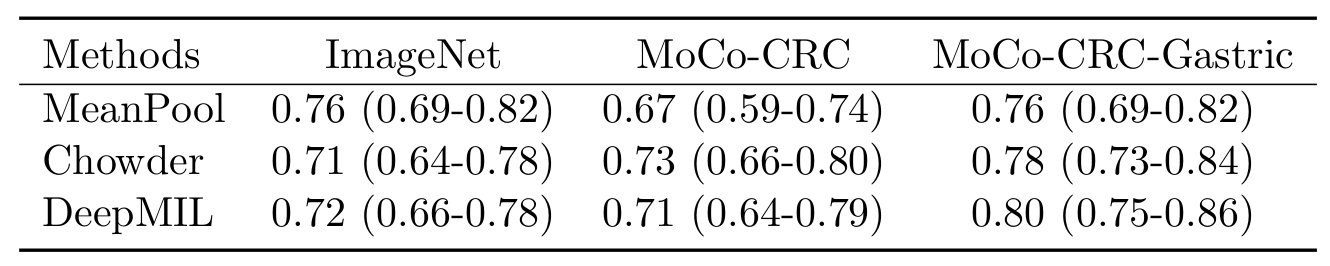
MoCo-CRC-Gastric model에서 일관되게 가장 좋은 성능을 보였다. 또한 저자는 ImageNet, MeanPool의 결과인 0.76은 PAIP에서의 0.61이라는 수치와 비교했을 때 꽤 좋은 성능을 보였다 설명한다. 마지막으로 저자는 위 결과가 지금껏 transfer setting from one organ to another에서 가장 좋은 성능이라 설명한다.
Interpretability
Chowder MIL 방식에서 각 tile이 convolutional 1D layer를 거쳐 single score로 표현하고, 이를 sorting 하여 top/bot tile만 MLP의 input으로 넣는다고 설명한 바 있다. 저자는 ImageNet와 MoCo feature extractor를 활용했을 때의 interpretability를 비교 측정하기 위해 다음과 같이 whole slide image 상에 tile의 score를 표현하였다.
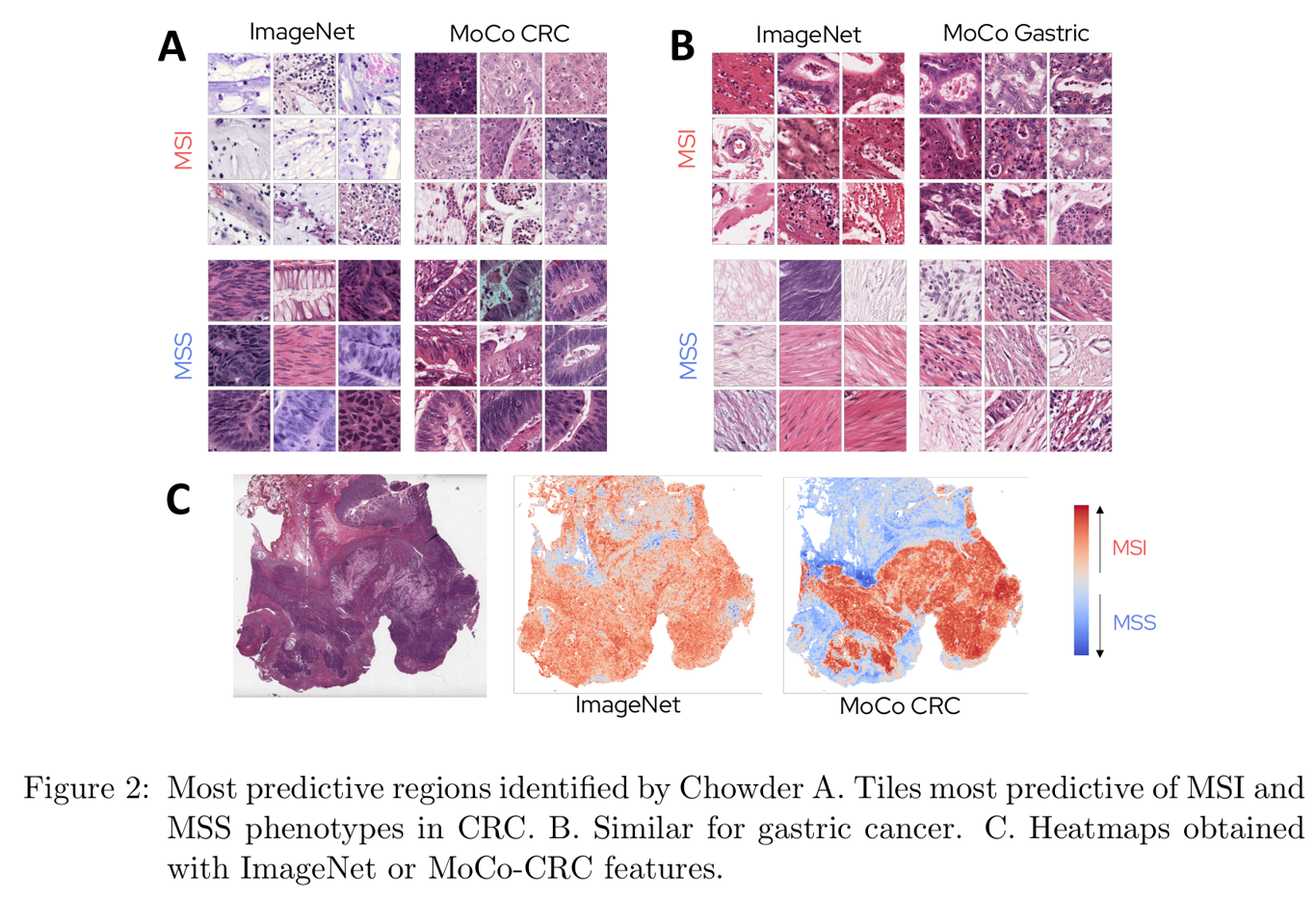
Lowest scored tile (MSS) 은 MSI pattern과 관련없는 muscle cell과 같은 non tumoral tissue를 가리켰다.
ImageNet 기반 모델이 within and outside tumor region을 가리킨 것에 반하여, MoCo-based model은 명확하게 tumor epithelium, 즉 poor differentiation of epithelial cell과 tumor infiltrating lymphocytes (TILs) 를 MSI tumor과 관련된 pattern으로 가리켰다.
Conclusion
- attention-based aggregation: Gated attention layer를 활용한 deep MIL framework를 적용함.
- weakly supervised learning with self-supervised learning: MoCo v2 모델을 적용하여 TCGA dataset에서 pre-train된 feature extractor를 사용함.
- explainability: Chowder 방식의 MIL 에서 tile이 얼마나 MSI와 관련되어 있는지가 single score로 표현된 것을 활용하여, slide 상에 각 tile의 score를 표현함.
이 논문에서는 SSL 그 중에서도 MoCo V2를 사용한 feature extractor를 통해 MSI prediction에서 좋은 성능을 보였다. (sota라고 한다.) Extensive CV를 활용하여 이 모델이 ImageNet 기반 모델보다 더 뛰어남을 보였고, generalization 측면에서도 결과가 더 잘 나옴을 확인하였다. 마지막으로 서로 다른 organ에서 train된 결과를 통해 sota 결과를 얻었다고 설명한다.
궁금했던 점
- Organ transfer setting에서 내가 이해한 바가 맞는지 잘 모르겠다. pre-train과 train가 각 데이터셋과 잘 짝지어졌는지 확신이 들지 않는다. . . .
- Chowder에서 어떻게 하나의 tile을 single value만으로 표현했는데도 저렇게 모델이 학습되는지.. 잘 모르겠다. 사실 어디가 이해가 안 가는지도 모르는 상태이다. 뭐가 문제인지는 정확하게는 모르지만 낯설고 이상하고 찝찝한 느낌.. 꼭 다시와서 다시 봐야겠다.
참고:
https://arxiv.org/pdf/1802.02212.pdf (Chowder)
https://arxiv.org/pdf/1802.04712.pdf (DeepAttnMIL)
https://arxiv.org/abs/1911.05722 (MoCo)
https://arxiv.org/abs/2003.04297 (MoCo v2)
