[Paper Review] Pan-cancer integrative histology-genomic analysis via multimodal deep learning
Paper Review
Pan-cancer integrative histology-genomic analysis via multimodal deep learning

CLAM 으로 익숙한 하버드의 mahmood lab 의 연구가 얼마 전에 공개되었다. TCGA data의 14개 암종에 대해, diagnostic tissue image 와 molecular profile data 를 융합하여 feature 를 만들어내었고 이를 survival analysis 에 활용하였다. c-index 등의 ranking performance 뿐만 아니라 interpretability 면에서도 훌륭한 성능을 보여주었다.
Introduction
- recent DL-based approaches는 survival label 과 같은 outcome-based label 을 활용하여 objective and prognostic molecular feature 를 찾고자 함.
- joint image-omic biomarker ex) oligodendroglioma and astrocytoma histolgoies with IDH1 mutation and 1p/19q-co-deletion status 는 정교한 patient stratification 가능케 함.
- multimodal fusion 은 improve precision & assist discover biomarker
Method
- joint image-omic biomarker 를 발굴하기 위해, MMF (multimodal fusion) algorithm 을 제안함.
- MMF: H&E WSI, molecular profile feature (mutation status, copy-number variation, RNA seq)
- survival outcome prediction 을 수행함과 동시에, how histopathology/molecular features + their interaction 이 low-, high-risk patient 와 correlation 을 이루는지 분석 수행.
- 1) attention-based 2) attribution-based method 를 통해 explainability 확보.
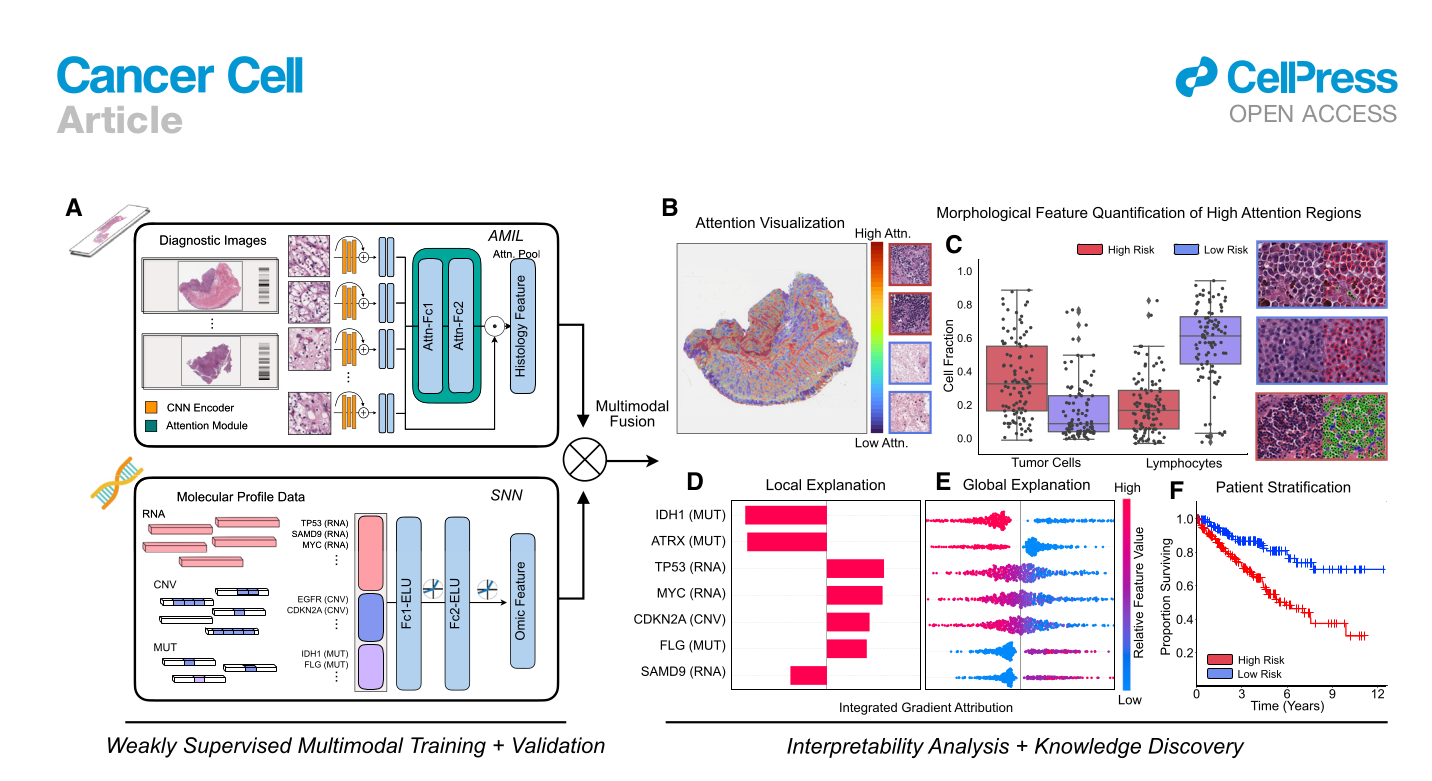
1) attention-based Multiple Instance Learning for processing WSI
2) Self-Normalizing Networks (SNN) for processing molecular profile data
3) multimodal fusion layer for tegrating WSIs and molecular profile data
dataset description & WSI preprocessing
TCGA project 의 14개 암종, 5720명 환자의 데이터
public CLAM 의 repository 를 활용하여 automated tissue segmentation 진행
image patch size 256 x 256, ResNet50 pretrained by ImageNet -> 1024 dimensional feature vector
AMIL
따로 clustering 기법 등을 사용하지 않은, AMIL를 처음 제안한 논문의 방법을 인용하고 사용함.
WSI processing 후 WSI bag 은 , (patch 개수 X 1024) 로 표현됨. 환자 수를 N 이라 했을 때 attention pooling 의 대상이 되는 matrix 는 이고, 이를 다음 식을 통해 로 변환.
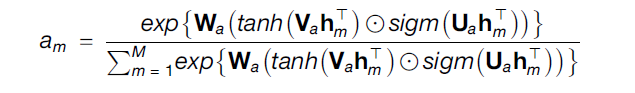

자세한 설명은 생략하지만, 직관적으로 설명하면, WSI 의 patch 여러 개의 feature vector 들을, feature vector 의 관계와 중요도를 판단하는 학습 가능한 network 를 통해, 하나의 feature vector 로 aggregate 해주는 계산임. 이렇게 생성된 vector 는 와 sigmoid activation 을 거쳐, negative-log-likelihood function for discrete time survival modling 에 활용된다. 또한 last fully-connected layer 는 WSI representation 을 위해 사용되고 이는 multimodal fusion layer 에 활용된다.
SNN
FCN 의 variation 중 하나로, high-dimensional low-sample size (HDLSS) sscenenarios 를 배우기 위한 architecture 중 하나이다. 자세한 내용은 모르지만 HDLSS property 를 띄는 molecular data 를 로 encoding 하기 위해 사용된다. 이는 multimodal fusion layer 에 활용된다.
Multimodal fusion layer
두 representation , 의 모든 bimodal interaction 을 잡아내기 위해, Kronecker Product 를 사용하여 새로운 differentiable fusion tensor 을 계산한다.
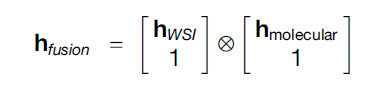
이 때, unimodal feature 도 잡아내고 서로 다른 modality의 feature collinearty 를 줄이기 위해 bias 처럼 1 을 concat 해준 뒤 Kronecker Product 를 계산하였다. 또한 이와 더불어, gating-based attentnion mechanism 을 추가적으로 적용하여, 각 modality 의 expressiveness 를 control 하고자 하였다. (i는 각 modality)
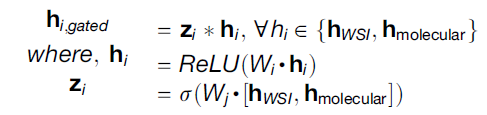
각 modality representation vector 에 대해 gated mechanism 을 먼저 적용한 후 fusion matrix 를 계산하였다. 이후 size 256의 두 개의 hidden layer 를 거쳤고, survival analysis 를 위한 cross entropy 처럼 생긴 loss function 을 계산하여 모델을 업데이트 하였다.
Results
model performances of PORPOISE and understanding impact of multimodal training
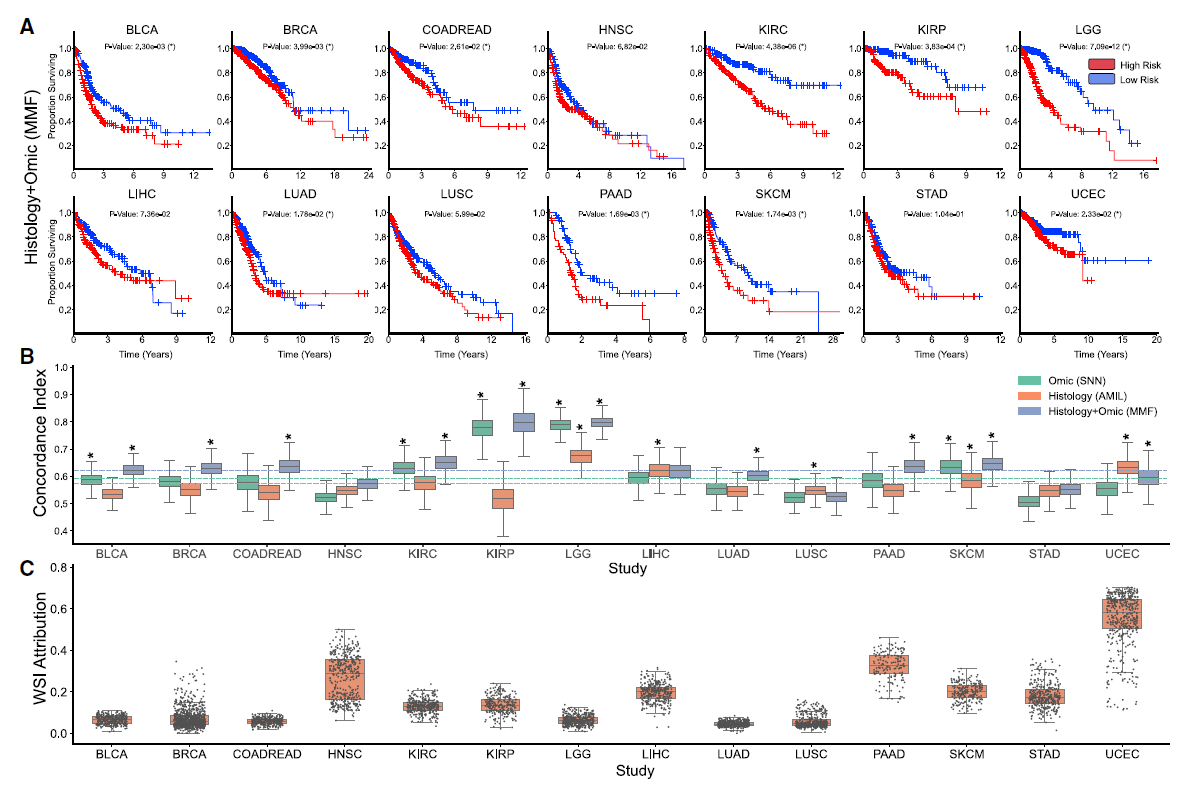
분석 대상이었던 모든 암종에 대한 성능 정보를 한 눈에 볼 수 있는 figure 이다. KM curve 를 봤을 때, 14개 중 4개 (HNSC, LIHC, LUSC, STAD) 를 제외하고 11개에 대해 significance 를 얻었다. c-index 의 경우 14개 중 2개 (LIHC, UCEC) 를 제외하고 12개에 대해 SNN, AMIL 즉 unimodality 정보만 사용한 것 보다 MMF 의 c-index 가 더 뛰어났다. fusion layer 에 들어가기 전 각 modality feature 에 대해 gate layer 를 통해 각 modality 의 attribution 을 계산하여 나타낸 결과, 대부분의 암종에 대해 molecular feature 의 지분이 압도적으로 컸지만, 그 중 LIHC, STAD, UCEC 는 WSI attribution 이 높게 나타났다.
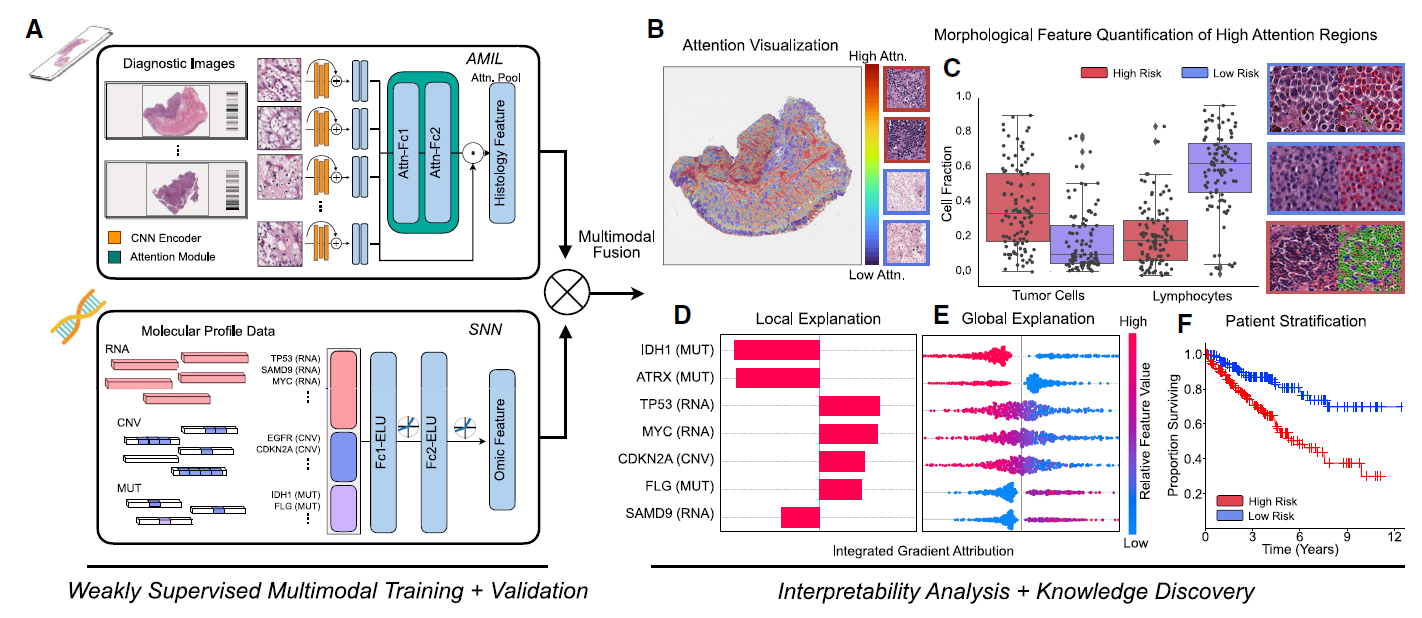
Quantitative performance, local model explanation, and global interpretability analyses of PORPOISE on ... cancer
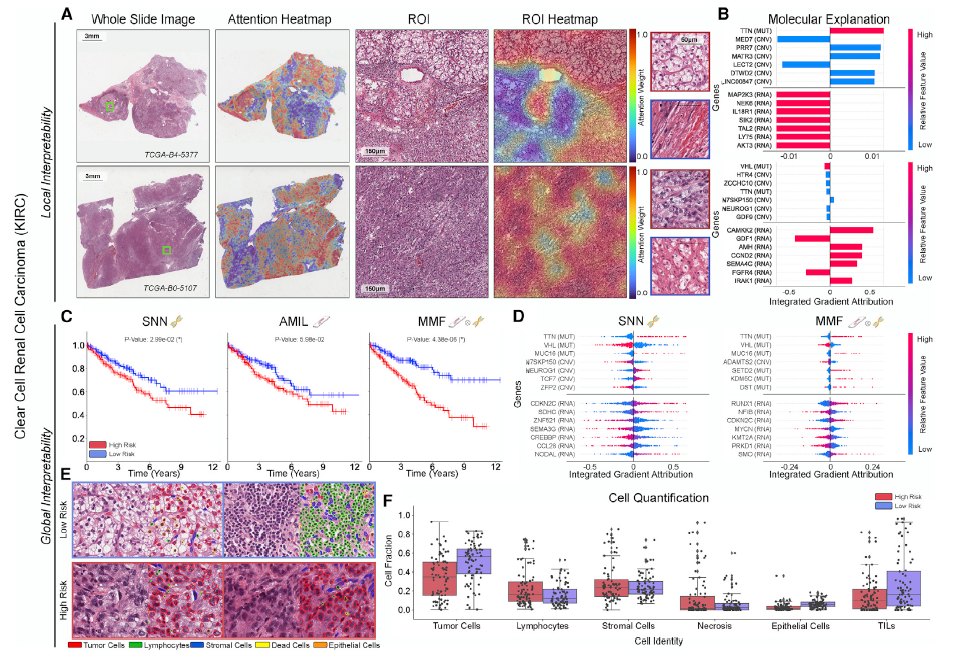
위와 같은 분석 자료가 supplementary data 를 포함하여 14개 암종 모두에 대해 주어졌다. Whole Slide Image 를 patch 화 하고 AMIL 의 attention score 를 이용하여 global 한 heatmap 과 ROI heatmap 를 A 에 나타내었다. B, D 는 SHAP 을 이용한 feature attribution visualization 결과이다. 각 모델에서 어떤 feature 가 의사 결정에 얼마나 영향을 미쳤는지를 알려준다. 예를 들어 B 는 local interpretability, 즉 individual sample 의 각 feature 가 얼마나, 어떻게 model risk prediction 에 영향을 미쳤는지를 보여주는데, x axis 는 attribution value 로 얼마나 에 해당하고 y axis 는 attribution 을 sorting 한 것이다. continuous colormap 은 해당 gene 이 mutation (1) 일 때 영향을 주는지 wildtype (0) 일 때 영향을 주는지를 나타내는 지표이다. D 는 global interpretability, 즉 해당 cancer cohort 전반에 대해 어떤 feature 가 의사결정에 얼마나 어떻게 영향을 미쳤는지를 보여준다. 자세한 내용은 SNN 쪽이라 일단은 남겨두도록 하자. C는 unimodality 를 사용했을 때와 fusion을 사용했을 때 각각 score 를 구한 뒤 KM curve 를 나타낸 결과이다. 전반적으로 unimodality 만을 사용했을 때 보다 multimodality 를 사용했을 때 두 group 이 더 잘 나뉘고 logrank test 결과 p value도 더 signifcant 하게 계산되었다. 마지막으로 E 는 attention score 가 높은 상위 1% patch (약 135개 정도) 만을 추출하여, HoverNet 을 이용해 cell segmentation 을 수행한 결과이다. 이 결과를 정리하여 cell quantification 까지 수행하였고, 특히 대부분의 암종에 대해 TIL (tumor infiltrating lymphocyte) 관련한 유의미한 결과를 얻었다.
다음부터는 논문에서 언급한 중요 결과의 요약이다.
- 12/14 cancer type 에서 MMF 는 highest c-index 달성
- 7/14 cancer type 에서 MMF 는 AMIL 보다 stratification significance 를 더 확보함.
- Kronecker product 를 이용한 fusion 을 사용했을 때 c-index 가 제일 잘 나옴.

- 8/14 cancer type 에서 low/high risk group 간 lymphocyte cell fraction 의 significant difference 를 보임.
- 6/14 cancer type 에서 low/high risk group 간 tumor cell fraction 의 significant difference 를 보임.
- LGG cohort 에서 IHD1 mutation 이 아주 중요한 prognostic factor 로 계산되었고, 이는 기존 연구들과 consistent 함.
- PAAD cohort 에서 전반적으로 innate immunity 와 inflammatory cell signaling 에 관여하는 gene 이 중요하다 계산됨.
- tumor-infiltrating lymphocyte (TIL) presence 가 9/14 cancer type 에서 significant difference 를 보임.
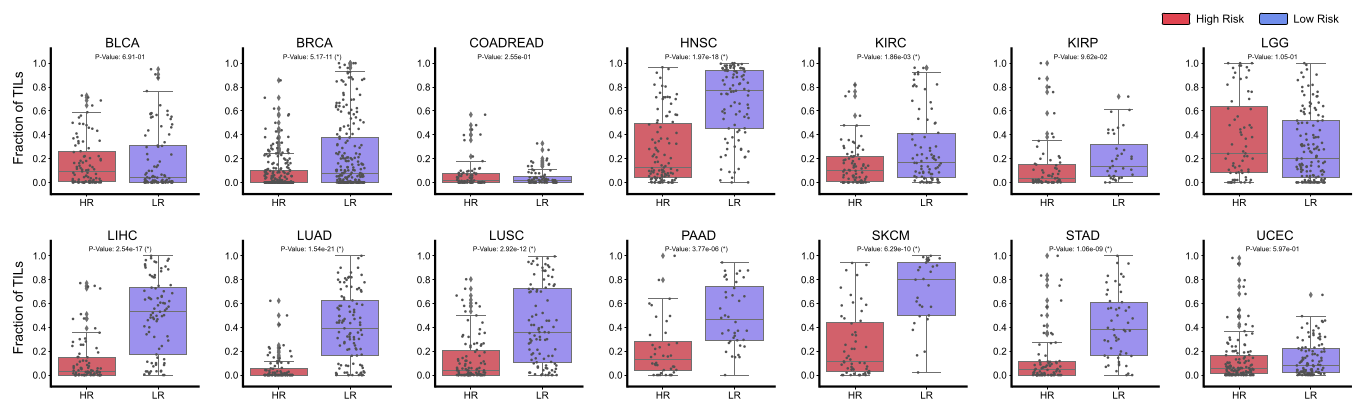
이때, TIL presence 는 highest-attended patch 에서 high tumor-immune cell co-localization 여부 (저자의 heuristic) 를 이용해 판단하였다. 20개의 cell 이 있고, 10개 이상의 lymphocyte 가 존재하고, 5개 이상의 tumor cell 이 존재할 때 TIL 이 존재한다 라고 정의하였다. 저자는 한계점에 TIL presence 와 statistical significance 를 평가하기 위해 post hoc analyses 가 필요하다 라고 덧붙였다.
Discussion
PAAD 의 경우, AMIL 이 prognostic 하지 않다 계산되었고 SNN 보다도 성능이 좋지 않았지만, multimodal integration 을 통해 성능이 향상됨과 동시에, WSI의 attribution score 비교적 높았다. 그 반대로 BRCA, COADREAD, LUAD 의 경우, uni-modality 일 때보다 multimodal integration 을 통해 성능이 향상되었고 WSI 대신 molecular feature 의 attribution score 가 높았다. 앞선 결과들을 포함한 논문의 전반의 설명을 증거로, computational support system for therapeutic decision-making 을 통해 향후 genotype-phenotype correlation-based analyses 는 shared + modality-specific 한 prognostic information 을 찾고 single/joint biomarker 를 발굴하는데에 큰 도움이 될 것이라 주장한다.
