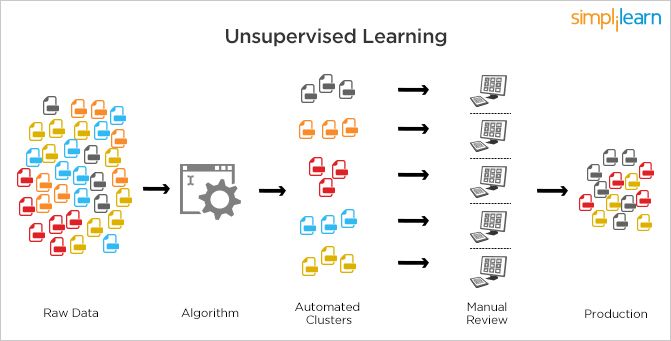
출처 : Simplilearn
비지도학습(Unsupervised learning)
- 지도학습과 달리 training data의 정답(혹은 label)이 없는 데이터가 주어지는 학습방법을 말합니다.
다르게 말하면, 비지도학습은 주어진 데이터가 어떻게 구성되어 있는지 스스로 알아내는 방법이라고도 말할 수 있습니다.
아무도 정답을 알려주지 않은 채 오로지 데이터셋의 특징(feature) 및 패턴을 기반으로 모델 스스로가 판단하는 것이니까요.
지도학습의 분류(classification) 문제를 생각해보면, 분류를 하기 위한 데이터가 필요하고
그러한 데이터의 대한 정답(label)이 필요합니다.
- 지도학습은 데이터를 training할 때 아래와 같은 작업이 필요합니다.
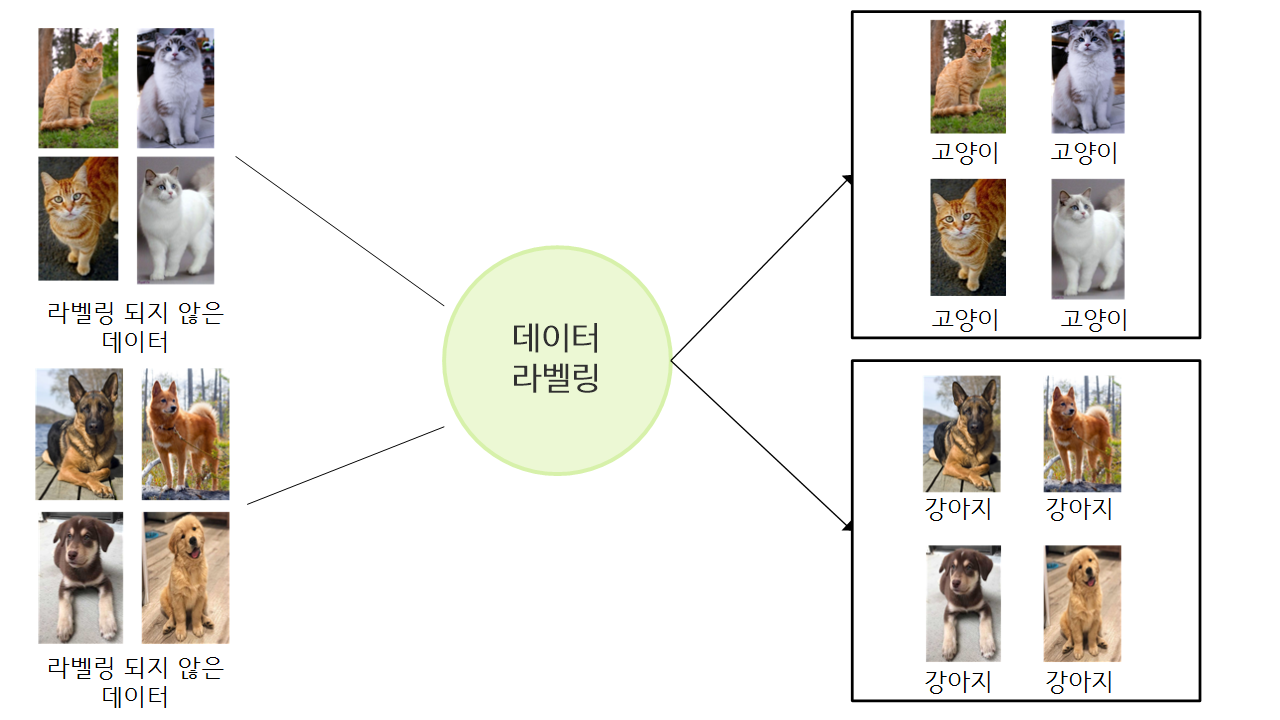
모든 데이터셋에 각각에 대한 정보가 명시되어 있는 경우 라벨링을 따로 해주지 않아도 됩니다.
그렇지만, 정보가 명시되어 있지 않는 경우 이를 해결하기 위해 많은 인적 자원이 소요되는데요.
이러한 문제를 해결하기 위해, 라벨링이 되어 있지 않은 데이터들 내에서 비슷한 특징이나 패턴을 가진 데이터들끼리 군집화한 후, 새로운 데이터가 어떤 군집에 속하는지를 추론하는 비지도학습과 같은 방법론이 제시되었습니다.
비지도학습의 대표적인 예시로는 군집화(클러스터링, clustering)가 있지만, 비지도학습이라는 용어는 정답이 없는 데이터를 이용한 학습 전체를 포괄하는 용어이기 때문에 클러스터링 외에도 차원 축소(dimensionality reduction) 및 이를 이용한 데이터 시각화, 생성 모델(generative model) 등 다양한 task를 포괄하는 개념입니다.
현재 포스팅에선 클러스터링의 K-means와 DBSCAN 알고리즘, 차원 축소의 PCA, T-SNE의 방법에 대해 다루어 보겠습니다.
클러스터링
비지도학습이 필요한 상황에서는 지도학습에서는 있었던 데이터 X가 무엇인지에 대한 y (label)이 없습니다.
그렇기에 y가 될 수 있는 분류 기준(클래스 또는 카테고리)도 없다고 할 수 있습니다.
군집화(클러스터링)이란 그렇게 명확한 분류 기준이 없는 상황에서도 데이터들을 분석하여 가까운(또는 유사한) 것들끼리 묶어 주는 작업을 말합니다.
K-means 알고리즘
-
k 값이 주어져 있을 때, 주어진 데이터들을 k 개의 클러스터로 묶는 알고리즘으로 대표적인 클러스터링 기법 중 하나입니다.
-
sklearn의 make_blob()을 활용하여 k개의 중심점을 기준으로 무작위 데이터 생성할 수 있습니다.
make_blob의 매개 변수 :
-
n_samples (int or array-like, default : 100) :
- 클러스터간에 나누어질 총 포인트 수 입니다.
- 리스트 형태로 들어온 경우 시퀀스의 각 요소는 클러스터 당 샘플 수를 나타냅니다.
-
n_features (int, default : 2) :
- 각 샘플의 기능 수입니다.
-
centers (int , ndarray of shape (n_centers, n_features), default : None) :
- 생성 할 센터 수 또는 고정 된 센터 위치입니다. n_samples가 int이고 centers가 None이면 3 개의 센터가 생성됩니다. n_samples가 배열과 유사한 경우 중심은 None이거나 n_samples의 길이와 동일한 길이의 배열이어야합니다.
-
cluster_std (float or array-like of float , default : 1.0)
- 군집의 표준 편차입니다.
-
center_box (tuple of float (min, max) , default=(-10.0, 10.0) )
- 중심이 무작위로 생성 될 때 각 클러스터 중심에 대한 경계 상자입니다.
-
shuffle (bool, default : True)
- 샘플을 섞습니다.
-
random_state (int, RandomState instance or None, default : None)
- 데이터 세트 생성을위한 난수 생성을 결정합니다. (시드 고정)
-
return_centers (bool, default : False)
- True이면 각 군집의 중심을 반환합니다.
K-means 알고리즘은 아래와 같이 사용할 수 있습니다.
- 5개의 중심점을 가진 무작위 데이터 생성
%matplotlib inline
from sklearn.datasets import make_blobs
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import random
# 중심점이 5개인 100개의 점 데이터를 무작위로 생성합니다.
points, labels = make_blobs(n_samples=100, centers=5, n_features=2, random_state=29)
# 데이터를 좌표에 그려보기
# 축 그리기
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
# 점 데이터를 X-Y grid에 시각화하기
ax.scatter(points[:, 0], points[:, 1], c='black', label='random generated data')
# 점 데이터 그리기
ax.legend()
ax.grid()
- 생성한 데이터에 K-means 알고리즘 적용
from sklearn.cluster import KMeans
# 위에서 생성한 무작위 점 데이터(points)에
# 클러스터의 수(K)가 5인 K-means 알고리즘을 적용
kmeans_cluster = KMeans(n_clusters=5)
#points에 대하여 K가 5일 때의 K-means iteration을 수행
kmeans_cluster.fit(points)
# 시각화 하기
color_dict = {0: 'red', 1: 'blue', 2:'green', 3:'brown', 4:'indigo'}
# 점 데이터를 X-Y grid에 시각화합니다.
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
# K-means clustering의 결과대로 색깔별로 구분하여 점에 색칠한 후 도식
for cluster in range(5):
cluster_sub_points = points[kmeans_cluster.labels_ == cluster] # 전체 무작위 점 데이터에서 K-means 알고리즘에 의해 군집화된 sub data를 분리합니다.
ax.scatter(cluster_sub_points[:, 0], cluster_sub_points[:, 1], c=color_dict[cluster], label='cluster_{}'.format(cluster)) # 해당 sub data를 plot합니다.
# 점 데이터 그리기
ax.legend()
ax.grid()- 그렇지만, K-means 알고리즘은 주어진 데이터의 분포에 따라 우리가 의도하지 않은 결과를 나타낼 수 있습니다.
# 원형 분포
from sklearn.datasets import make_circles
# 원형 분포 데이터 생성
circle_points, circle_labels = make_circles(n_samples=100, factor=0.5, noise=0.01)
# 원형 분포를 가지는 points 100개 생성
# 캔버스 생성
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
# 원형 분포에 대해 K-means 수행
circle_k_means = KMeans(n_clusters=2)
circle_k_means.fit(circle_points)
color_dict = {0: 'blue', 1: 'red'}
for cluster in range(2):
cluster_sub_points = circle_points[circle_kmeans.labels_ == cluster]
ax.scatter(cluster_sub_points[:, 0], cluster_sub_points[:, 1], c=color_dict[cluster], label='cluster -{}'.format(cluster))
ax.set_title('circle data, K=2')
ax.legend()
ax.grid()
- 위를 실행 시키면 원 모양으로 제대로 군집화 되어 있지 않은 모습을 확인할 수 있다.
K-means 알고리즘을 사용하기 어려운 상황:
-
군집의 개수(K 값)를 미리 지정해야 하기 때문에 이를 알거나 예측하기 어려운 경우에는 사용하기 어렵다.
-
유클리드 거리가 가까운 데이터끼리 군집이 형성되기 때문에 데이터의 분포에 따라 유클리드 거리가 멀면서 밀접하게 연관되어 있는 데이터들의 군집화를 성공적으로 수행하지 못할 수 있다.
다음으로, DBSCAN에 대해 얘기하겠습니다.
DBSCAN 알고리즘
DBSCAN (Density Based Spatial Clustering of Applications with Noise) 알고리즘은 가장 널리 알려진 밀도(density) 기반의 군집 알고리즘입니다.
- K-means 알고리즘을 사용하여 해결하기 어려웠던 문제들을 DBSCAN 알고리즘을 활용해 해결할 수 있습니다.
DBSCAN 알고리즘의 특징
- 군집의 개수, 즉 K-means 알고리즘에서의 K 값을 미리 지정할 필요가 없습니다.
- 유클리드 거리 기반의 K-means 알고리즘 방식과 달리,
조밀하게 몰려 있는 클러스터를 군집화하는 방식을 사용하기 때문에 원 모양의 군집이 아닌 불특정한 형태의 군집도 찾을 수 있습니다.
DBSCAN 알고리즘의 동작
DBSCAN을 이해하는 데 필요한 변수와 용어 정리
- epsilon: 클러스터의 반경
- minPts: 클러스터를 이루는 개체의 최솟값
- core point: 반경 epsilon 내에 minPts 개 이상의 점이 존재하는 중심점
- border point: 군집의 중심이 되지는 못하지만, 군집에 속하는 점
- noise point: 군집에 포함되지 못하는 점
DBSCAN에서는 epsilon과 minPts 값이 미리 지정해 주어야 하는 변수입니다.
DBSCAN 알고리즘의 동작 순서
- 임의의 점 p를 설정하고, p를 포함하여 주어진 클러스터의 반경(elipson)안에 포함되어 있는 점들의 갯수를 셉니다.
- 만일 해당 원에 minPts 이상의 점이 포함되어 있으면, 해당 점 p를 core point로 간주하고 원에 포함된 점들을 하나의 클러스터로 묶습니다.
- 해당 원에 minPts 미만의 점이 포함되어 있으면, 무시합니다.
- 모든 점에 대하여 돌아가면서 1~3 번의 과정을 반복하는데, 만일 새로운 점 가 core point가 되고 이 점이 기존의 클러스터(p를 core point로 하는)에 속한다면, 두 개의 클러스터는 연결되어 있다고 하며 하나의 클러스터로 묶어줍니다.
- 모든 점에 대하여 클러스터링 과정을 끝냈지만, 어떤 점을 중심으로 하더라도 클러스터에 속하지 못하는 점이 있으면 이를 noise point로 간주해요.
그리고 특정 군집에는 속하지만 core point가 아닌 점들을 border point라고 부릅니다.
K-means vs DBSCAN 알고리즘
K-means로는 잘 처리 되지 않았던 예시가 DBSCAN에서는 어떻게 처리가 되는지 확인해 봅시다.
from sklearn.cluster import DBSCAN
fig = plt.figure()
ax= fig.add_subplot(1, 1, 1)
color_dict = {0: 'red', 1: 'blue', 2: 'green', 3:'black',4:'yellow'}
# n 번째 클러스터 데이터의 색깔 도식을 결정하는 color dictionary
# 원형 분포 plot
epsilon, minPts = 0.2, 3 # 초기 epsilon, minPts 값을 설정
circle_dbscan = DBSCAN(eps=epsilon, min_samples=minPts) # 위에서 생성한 원형 분포 데이터에 DBSCAN setting
circle_dbscan.fit(circle_points) # 3) ~ 5) 과정을 반복
n_cluster = max(circle_dbscan.labels_)+1 # 3) ~5) 과정의 반복으로 클러스터의 수 도출
print(f'# of cluster: {n_cluster}')
print(f'DBSCAN Y-hat: {circle_dbscan.labels_}')
# DBSCAN 알고리즘의 수행결과로 도출된 클러스터의 수를 기반으로 색깔별로 구분하여 점에 색칠한 후 도식
for cluster in range(n_cluster):
cluster_sub_points = circle_points[circle_dbscan.labels_ == cluster]
ax.scatter(cluster_sub_points[:, 0], cluster_sub_points[:, 1], c=color_dict[cluster], label='cluster_{}'.format(cluster))
ax.set_title('DBSCAN circle data')
ax.legend()
ax.grid()
위 코드에서 확인할 수 있듯이, epsilon과 minPts 값을 잘 조절해주면 DBSCAN 알고리즘에 따라 클러스터의 수를 명시해주지 않아도 적절한 클러스터의 개수를 설정하여 주어진 데이터에 대한 군집화를 수행할 수 있습니다.
위의 결과만 보면 DBSCAN이 더 좋아보이지만, 군집화할 데이터의 수가 많아질수록 DBSCAN의 알고리즘 수행시간이 급격하게 늘어나는 단점을 가지고 있습니다.
차원 축소 (Dimensionality reduction)
주성분분석(PCA): 차원 축소(Dimensionality reduction) 알고리즘
- 가장 중요한 요소가 무엇인지 우선 순위를 정해서 해당 요소를 기준으로 필터링을 하고, 특정 요소는 고려하지 않습니다.
비지도학습에서는 데이터를 나타내는 여러 특징(feature)들 중에서 어떤 특징이 가장 그 데이터를 잘 표현(represent)하는지 알게 해주는 특징 추출(feature extraction)의 용도로 사용된다.
PCA 대수적 설명 :
PCA는 데이터 분포의 주성분을 찾아주는 방법입니다.
( 여기서 주성분이라는 의미는 데이터의 분산이 가장 큰 방향벡터를 의미해요. )
PCA는 데이터들의 분산을 최대로 보존하면서, 서로 직교(orthogonal)하는 기저(basis, 분산이 큰 방향벡터의 축)들을 찾아 고차원 공간을 저차원 공간으로 사영(projection)합니다..
또한 PCA에서는 기존 feature 중 중요한 것을 선택하는 방식이 아닌 기존의 feature를 선형 결합(linear combination)하는 방식을 사용하고 있습니다.
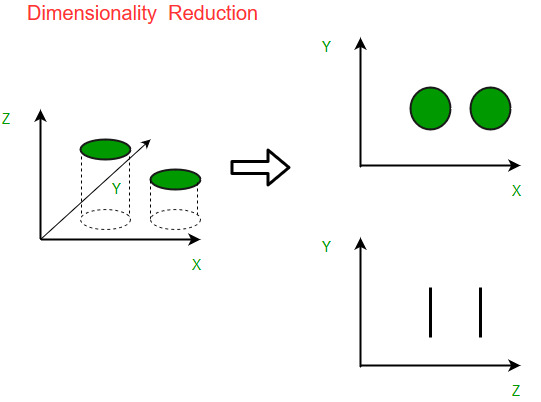 (출처 : https://www.geeksforgeeks.org/dimensionality-reduction/)
(출처 : https://www.geeksforgeeks.org/dimensionality-reduction/)
위의 그림과 같이 타원형 분포로 되어있는 데이터들이 있을 때, 차원의 수는 줄이면서 데이터 분포의 분산을 최대한 유지하기 위해 가장 분산이 긴 축을 첫 기저로 잡고, 그 기저에 직교하는 축 중 가장 분산이 큰 값을 다음 기저로 잡게 돼요. 이 과정을 반복하게 되면 차원의 수를 최대로 줄이면서 데이터 분포의 분산을 그대로 유지할 수 있답니다. 이것을 차원 축소라고 합니다.
위 그림에서라면 여러 개의 차원으로 구성된 데이터들을 2개의 차원으로 축소해도 정보의 손실을 최소화하여 데이터의 분포를 충분히 표현할 수 있다는 의미를 가지고 있습니다.
여기서 차원 축소의 핵심 개념으로 사영(projection)이라는 용어가 등장합니다.
X-Y-Z 좌표축상에 존재하는 데이터를 X-Y, Y-Z 좌표축에 사영(projection) 했다는 것은 각각 Z, X 좌표축을 무시했다는 뜻이 됩니다.
위의 그림과 같이 X-Y, Y-Z를 사영했을때 특성을 더 잘 살리면서 차원을 축소한 것은 X-Y라고 볼 수 있습니다.
- Y-Z는 원기둥이 선으로 축소되었고, X-Y는 원으로 축소 되었기 때문입니다.
주성분(Principal Component) 방향 or pc축
PCA는 차원축소를 시도하되, 주어진 좌표축 방향이 아니라, 가장 분산이 길게 나오는 기저(basis) 방향을 찾아서 그 방향의 기저만 남기고, 덜 중요한 기저 방향을 삭제하는 방식으로 진행됩니다.
- 이렇게 찾은 가장 중요한 기저를 부르는 말입니다.
import pandas as pd
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
# color dictionary
color_dict = {0: 'red', 1: 'blue', 2:'red', 3:'blue'}
target_dict = {0: 'malignant_train', 1: 'benign_train', 2: 'malignant_test', 3:'benign_test'}
#Train data에 PCA 알고리즘 적용
train_X_ = StandardScaler().fit_transform(train_X) # 불러온 데이터에 대한 정규화 -> 각 column의 range of value가 전부 다르기 때문에 정규화를 진행해 주어야 합니다.
train_df = pd.DataFrame(train_X_, columns=cancer['feature_names'])
pca = PCA(n_components=2) # 주성분의 수를 2개, 즉 기저가 되는 방향벡터를 2개로 하는 PCA 알고리즘 수행
pc = pca.fit_transform(train_df)
위의 코드와 같이 train data에 PCA 알고리즘을 적용할 수 있습니다.
위의 그림에서 왼쪽이 PCA를 하고 분류를한 결과이고 오른쪽이 PCA를 하지 않고 분류를 한 결과입니다.
PCA를 하면서 차원 축소를 하고 feature의 수가 적어졌지만, feature의 수가 더 많은 오른쪽 결과 보다 더 좋은 분류를 나타냅니다.
-
이렇듯 PCA는 feature의 수가 적더라도, 중요한 feature만을 이용한 분류를 이용하면 정확도를 더 높일 수 있습니다.
-
그리고 PCA는 주로 선형적인 데이터의 분포(키와 몸무게 등)를 가지고 있을 때, 정보가 가장 잘 보존됩니다.
T-SNE
T-SNE(T-Stochastic Neighbor Embedding)은 시각화에 많이 쓰이는 알고리즘입니다.
- T-SNE는 기존 차원의 공간에서 가까운 점들은, 차원축소된 공간에서도 여전히 가깝게 유지 되는 것을 목표로 하고있습니다.
T-SNE와 PCA 시각화
PCA는 데이터가 가진 고유한 물리적 정보량을 보존하는데 주력하지만, T-SNE는 고유한 물리적 정보량보다는 데이터들간의 상대적 거리를 보존하는데 주력합니다.
PCA 시각화
- PCA를 통해 차원축소를 하면서 발생하는 정보 손실의 과정 중에는 두 점 사이의 거리 정보가 함께 손실되어서 위의 분포와 같이 실제로는 먼 거리에 점들이 가까운 점으로 투영될 수 있습니다.
T-SNE 시각화
-
T-SNE는 기존 차원의 공간에서 가까운 점들은, 차원축소된 공간에서도 여전히 가깝게 유지 되고 먼거리의 점들은 저 차원에서도 먼 거리에 있기에 시각화 했을때 각 군집을 확인할 수 있다.
-
T-SNE의 이러한 점 때문에 Feature Extractor 모델이 카테고리간 분류 경계선을 뚜렷하게 유지하고 있는지를 확인하는 용도로 자주 쓰입니다.
T-SNE와 PCA 물리적 의미
-
PCA는 정보 손실을 최소화하려는 관점을 가지고 있습니다.
그 결과 추출된 PC축은 주성분이라는 물리적 의미를 유지하고 있으며, 공분산을 통해 원본 데이터를 일정 부분 복원할 수 있는 가능성을 가지고 있습니다. -
그렇지만 T-SNE은 정보 손실량에 주목하지 않습니다.
그 결과 저차원 축이 아무런 물리적 의미를 가지지 못하고 오직 시각화에만 유리할 뿐입니다.
끝으로 비지도 학습에 대한 정리를 마치면서,
지도학습 보다는 비지도학습의 개념이 좀 더 어려웠던것 같습니다.
- 잘못된 부분에 대해서 알려주시면 해당 사항을 고치겠습니다.