여러 데이터 소스를 통합 데이터셋으로 합치기
9장에서는 개괄적인 목표와 우리가 만들 시스템에서 데이터가 어떻게 흘러갈지에 대한 윤곽을 잡았다. 10장에서는 원본 데이터를 읽어들이고 전처리하는 데이터 로딩 부분에 집중하도록 한다. 우리의 목표는 원본 CT스캔 데이터와 데이터에 달아놓은 애노테이션 목록으로 훈련 샘플을 만드는 것이다.
원본 CT 데이터 파일
CT데이터는 메타데이터 헤더 정보가 포함된 .mhd 파일과 3차원 배열을 만들 원본 데이터 바이트를 포함하는 .raw 파일로, 두 가지 종류이다. CT클래스는 두 파일을 읽어 3차원 배열을 만들고 환자 좌표계를 배열에서 필요로 하는 인덱스, 행, 열 좌표로 바꿔주는 변환 행렬도 만든다. 즉 CT데이터에 적용할 좌표계 변환이 필요하다.
Series UID란
Series UID (시리즈 UID): 한 연구(Study) 내에서 스캔 또는 이미지 시리즈를 식별하는 UID이다.
이미지 시리즈(Image Series)는 의료 영상 데이터에서 관련 이미지들의 그룹이다. 하나의 이미지 시리즈는 일련의 관련 이미지들로 구성되며, 동일한 스캔 프로토콜, 스캔 조건, 스캔 부위 등의 공통 특성을 가지고 있다. 이미지 시리즈는 보통 하나의 CT 스캔 또는 MRI 스캔을 나타내며, 해당 스캔에서 얻은 연속적인 이미지들을 포함한다.
즉 이미지 시리즈는 한 인간에 대한 CT스캔1회를 나타내는 것으로 CT스캔 1회는 여러개의 이미지로 구성된다. 그리고 여러개의 이미지는 이미지 시리즈 한 단위로 귀결된다.
LUNA 애노테이션 데이터 파싱
앞서 설명한 부분보다 더 이해를 쉽게 하는 것은 직접 데이터를 로딩해보는 것이다. 데이터가 로딩된 후 어떻게 보일지를 알면 초기 실험 구조를 이해하는 데 도움이 된다.
candidates.csv의 구성을 보면 쉼표로 구분되며, 순서대로 SeriesUID, x, y, z, class를 나타낸다. UID요소가 점으로 연결되어 굉장히 복잡한 모습을 보인다. 전체 행 개수는 551,000개이며 실제 결절로 분류된 1,351개의 케이스가 존재한다.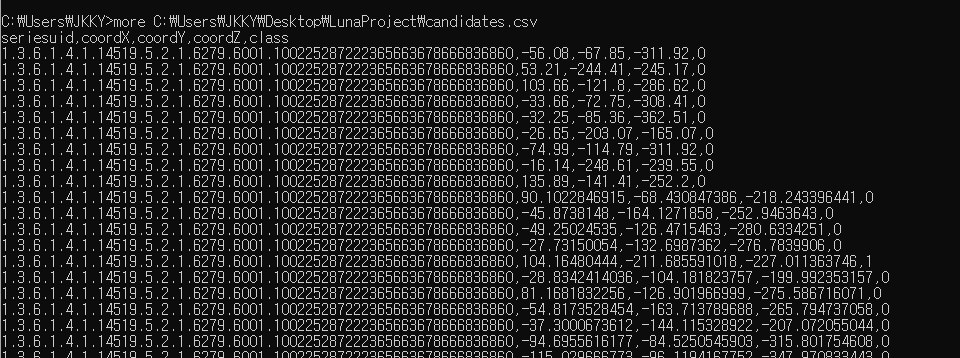
annotations.csv 파일에는 결절로 플래그된 후보들에 대한 정보가 포함되어 있다. 특별히 마지막 피처인 diameter_mm 정보를 주목할만하다.(아마 결절의 직경정보, 단위는 mm)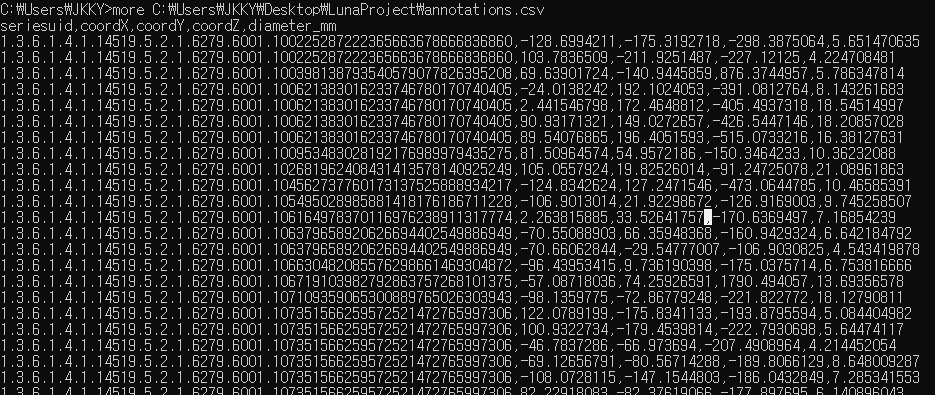
훈련셋과 검증셋
모든 표준 지도학습 작업은 데이터를 training set과 validation set으로 나눈다. 모두 실세계 데이터를 반영해야 하며, 일반적인 방식으로 다루는 것을 가정한다. 주의해야할 점은 annotations.csv에서 제공하는 위치 정보는 candidates.csv의 좌표와 정확하게 일치하지 않는 경우가 존재한다. 따라서 데이터가 불일치한 경우라면 무시하도록 하자.
애노테이션 데이터와 후보 데이터 합치기
getCandidateInfoList함수를 만들어 데이터를 합치는 역할을 부여하자.
from collections import namedtuple
CandidateInfoTuple = namedtuple(
'CandidateInfoTuple',
'isNodule_bool, diameter_mm, series_uid, center_xyz',
)
# CandidateInfoTuple이라는 이름의 네임드 튜플을 제작 했으며 속성은 총4가지이다.
# isNodule_bool : 결절의 여부(True or False)
# diameter_mm : 덩어리의 직경
# series_uid : 이미지시리즈를 식별하는 uid값
# center_xyz : 덩어리의 중심의 xyz 튜플값이 튜플은 우리가 필요로 하는 CT데이터가 빠져 있어 훈련 샘플은 아니다. 이 데이터는 우리가 사용하는 애노테이션 데이터에 대해 깔끔하게 다듬어진 통합 인터페이스를 나타낸다. 이렇게 지저분한 구조를 처리하면 훈련 루프도 깔끔해진다.
후보 정보 리스트는 결절의 상태와 결절의 직경, 순번, 중심점을 갖는다. NoduleInfoTuple 인스턴스 리스트를 만드는 함수는 인메모리 캐싱 데코레이터를 사용하고 디스크 파일 경로를 얻는다.
@functools.lru_cache(1)
def getCandidateInfoList(requireOnDisk_bool=True):
mhd_list = glob.glob('~/subset*/*.mhd')
presentOnDisk_set = {os.path.split(p)[-1][:-4] for p in mhd_list}functools.lru_cache(1)
함수에 캐시 기능을 적용하기 위한 decorator이며 캐시는 함수의 인자와 결과를 저장한다. 즉 캐시에는 하나의 결과를 저장하게 되며 인자에 대한 return값을 저장하여 다음 같은 실행때 계산없이 사용하게끔 한다. return에 뭘 반환할 것인지는 뒤에 가서 보도록 한다.
requireOnDisk_bool 파라미터를 통해 디스크상에서 시리즈UID가 발견되는 LUNA데이터만 사용하고 이에 해당하는 엔트리만 CSV파일에서 걸러 사용할 예정이다. 디스크상에서 시리즈UID가 발견되는 데이터만 사용한다는 뜻은 Luna데이터셋안의 CT데이터중 시리즈UID가 있는 것만 사용한다는 의미이다. 즉 누락된 것은 버리겠다는 뜻이다.
여기서 잠깐 정리!
subset과 annotations와 candidates정보를 다시 정리하고 가야한다.
subset은 모든 폐사진(결절존재, 결절존재x)의 uid(.mhd)와 사진들(.raw)을 담고 있으며
annotations.csv는 subset에서 결절로 분류되는 폐 데이터에 대한 정보를 가진다.(subset내의 모든 결절인 폐 사진을 포괄하진 않는 것으로 보인다. 1200여개의 결절에 대한 정보를 담고 있다.)
candidates.csv는 폐 결절 감지 알고리즘의 성능 평가와 폐 결절 후보들의 정보를 가진다. 즉 폐결절이 아닌 단순 덩어리와 같은 다른 증상의 데이터 정보도 포함될 수 있다.(55만여개의 데이터로 구성되었으며 그 중 class=1인 즉 결절일 경우가 1300여개를 나타낸다.)
후보 정보를 얻었다면 annotations.csv의 직경 정보를 합친다. 합치는 과정과 이전 과정까지 합쳐 getCandidateInfoList모듈을 완성한다.
@functools.lru_cache(1)
def getCandidateInfoList(requireOnDisk_bool=True):
mhd_list = glob.glob('data-unversioned/part2/luna/subset*/*.mhd')
presentOnDisk_set = {os.path.split(p)[-1][:-4] for p in mhd_list}
# mhd파일에서 seriesuid만 얻는다. 데이터들중 시리즈 UID가 존재하는 것들을 캐내는 작업이다.
# ex. 1.3.6.1.4.1.14519.5.2.1.6279.6001.105756658031515062000744821260 와 같은 형식의 값을 모두 얻어 집합으로 만든다.
# 정리 : subset의 모든 UID가 존재하는 폐 데이터 UID를 저장한다
diameter_dict = {} # 1200여개의 결절 정보를 담는 annotations.csv안의 위치,직경정보 담기 위한 딕셔너리
with open('data/part2/luna/annotations.csv', "r") as f:
for row in list(csv.reader(f))[1:]:
series_uid = row[0] # annotations 0번째 데이터인 uid값 저장
annotationCenter_xyz = tuple([float(x) for x in row[1:4]]) # xyz값 튜플로 저장
annotationDiameter_mm = float(row[4]) # 직경 값 저장
diameter_dict.setdefault(series_uid, []).append(
(annotationCenter_xyz, annotationDiameter_mm)
) # 딕셔너리에 uid를 키로 나머지 값을 튜플(튜플, 값)으로 저장
candidateInfo_list = [] # 후보군 리스트 뽑기
with open('data/part2/luna/candidates.csv', "r") as f:
for row in list(csv.reader(f))[1:]:
series_uid = row[0] # 후보군 UID 저장
if series_uid not in presentOnDisk_set and requireOnDisk_bool:
# 후보군uid이 전체 uid셋에 없을 경우 continue하여 현재 row에 대한 정보 파기
continue
isNodule_bool = bool(int(row[4])) # 0,1로 구성된 결절 여부 저장
candidateCenter_xyz = tuple([float(x) for x in row[1:4]]) # xyz값 튜플로 저장
candidateDiameter_mm = 0.0 # 직경값 0으로 설정(candidates.csv엔 직경정보가 없으므로)
for annotation_tup in diameter_dict.get(series_uid, []):
# annotations xyz값은 실제로 약간의 차이를 보이므로 서로의 차이가 직경의 4분의 1 이하일때만
# candidates직경값(=0)을 annotation값을 수정한다.
annotationCenter_xyz, annotationDiameter_mm = annotation_tup
for i in range(3):
delta_mm = abs(candidateCenter_xyz[i] - annotationCenter_xyz[i])
if delta_mm > annotationDiameter_mm / 4:
break
else: # break이 되지 않았다면
candidateDiameter_mm = annotationDiameter_mm
break
# 미리 정의한 namedtuple에 데이터 저장하고 list에 append
candidateInfo_list.append(CandidateInfoTuple(
isNodule_bool,
candidateDiameter_mm,
series_uid,
candidateCenter_xyz,
))
# 정렬(class=1,0 기준으로 정렬)
candidateInfo_list.sort(reverse=True)
return candidateInfo_list # 캐시 메모리에 저장 됨.개별 CT 스캔 로딩
다음은 디스크에서 CT데이터를 얻어와 파이썬 객체로 변환하여, 3차원 결절 밀도 데이터로 사용할 수 있도록 만드는 작업이다. CT데이터의 원래 포맷인 생소한 DISCOM포맷을 LUNA측이 MetalO 포맷으로 변환해놓아 사용하기 편리하다. 파일 포맷을 읽어들이기 위한 SimpleITK만 활용하면 된다.
하운스 필드 단위
인자로 SeriesUID를 받아 이미지를 가져올 모듈인 CT모듈을 만들기 이전에 하운스필드 단위에 대해 간략하게 이해하고 가자. CT 스캔 복셀은 처음 들어본 하운스필드 단위로 표시하는데, 일례로 공기는 -1000HU, 물은 0HU, 뼈는 +1000HU이다. 뼈와 공기는 양쪽의 제한 값이 된다. 아마 인간의 몸에서 공기가 가장 낮은 하운스필드 값을 가지고 뼈가 가장 높은 하운스필드 값을 가지는 것으로 이해하면 될 것 같다. 종양의 경우 대체로 0HU근처라고 한다.
class Ct:
def __init__(self, series_uid):
mhd_path = glob.glob(
'data-unversioned/part2/luna/subset*/{}.mhd'.format(series_uid)
)[0] # 어느 subset에 seriesUID가 있을지 모르니 모든 경로를 리스트화
ct_mhd = sitk.ReadImage(mhd_path)
# path경로들로 하여금 sitk.ReadImage를 사용해서 이미지 객체로 불러온다.
ct_a = np.array(sitk.GetArrayFromImage(ct_mhd), dtype=np.float32)
# 불러온 이미지 객체를 넘파이 배열(dtype=float32)로 변환한다.
ct_a.clip(-1000, 1000, ct_a)
# 하운스필드 단위를 고려하여 ct_a배열의 값을 -1000~1000 값으로 제한을 둔다.
# 제한값보다 더 큰값이나 작은값은 +-1000로 변환한다.
self.series_uid = series_uid # 속성값 인자값으로 초기화
self.hu_a = ct_a # 변환한 ct_a 속성값으로 초기화
환자 좌표계를 사용해 결절 위치 정하기
환자 좌표계
환자 좌표계는 우리가 이전 데이터에서 본 x, y, z 값을 의미하며 안타깝게도 복셀이 아니라 밀리미터 단위로 표시되어 있다. 즉 (x,y,z)에서 (I,R,C)로의 변환이 필요하다. 환자 좌표계는 특정 스캔과 무관하게 해부학적으로 관심있는 위치를 지정하기 위해 사용되어 왔다.
CT 스캔 형태와 복셀 크기
CT 스캔마다 조금씩 다른 부분 중 하나는 복셀의 크기이다. 일반적으로 복셀은 정육면체가 아니고 1.125x1.125x2.5mm 크기 또는 이와 유사한 크기를 가진다. 복셀이 정육면체가 아니기 때문에 정방형의 픽셀로 그려내면 북극과 남극이 실제보다 크게 그려지는 메르카토르식 세계지도처럼 왜곡된 이미지를 보이게 된다.
CT는 일반적으로 512x512로 인덱스 차원은 대략 총 100~250개의 단면으로 이루어진다. 즉 한번의 CT스캔에 대해 3,200만 개의 데이터 포인트가 나타난다는 것이다.
밀리미터를 복셀로 변환하기
다음과 같은 순서로 xyz를 IRC로 만든다.
1. 좌표를 XYZ체계로 만들기 위해 IRC에서 CRI로 뒤집는다.
2. 인덱스를 복셀 크기로 확대축소한다.
3. 파이썬의 @를 사용하여 방향을 나타내는 행렬과 행렬곱을 수행한다.
4. 기준으로부터 오프셋을 더한다.
